Koronawirusy
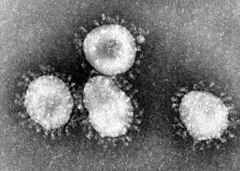 Wirus SARS (obraz z mikroskopu elektronowego) | |
| Systematyka | |
| Grupa |
Grupa IV ((+)ssRNA) |
|---|---|
| Rząd | |
| Rodzina |
Coronaviridae |
| Podrodzina |
Coronavirinae |
| Rodzaj |
Koronawirus |
| Cechy wiralne | |
| Kwas nukleinowy |
RNA |
| Liczba nici |
jedna |
| Polaryzacja kwasu nukleinowego |
dodatnia |
| Wywoływane choroby |
zakażenia dróg oddechowych |
Koronawirusy – gatunki wirusów, należących do podrodziny Coronavirinae z rodziny Coronaviridae w rzędzie Nidovirales. Podrodzina Coronavirinae podzielona została na cztery rodzaje: alfa-, beta-, gamma- i deltakoronawirusy (łac. Alphacoronavirus, Betacoronavirus, Gammacoronavirus i Deltacoronavirus). Nosicielami poszczególnych gatunków są ssaki lub ptaki[1][2].
Koronawirusy posiadają osłonkę oraz pojedynczą nić RNA o symetrii helikalnej i polarności dodatniej. Rozmiar genomu znanych koronawirusów mieści się w zakresie od 26,4 do 31,7 tys nt[2], co jest wartością niezwykle dużą jak na wirusy RNA. Nazwa „koronawirus” wywodzi się z łac. corona oznaczającego koronę lub wieniec, ponieważ osłonki wirusów w mikroskopii elektronowej wydają się „ukoronowane” pierścieniem małych, przypominających żarówki struktur. Klasa Baltimore: IV.
Koronawirusy zwierzęce
[edytuj | edytuj kod]Koronawirusy zakażają ptaki i ssaki, powodując liczne choroby układu oddechowego, nerwowego, narządów wewnętrznych czy układu pokarmowego. Do chorób wywoływanych przez koronawirusy zwierzęce należą m.in.:
- zakaźne zapalenie oskrzeli u ptactwa (wywołane przez wirus ptasiego zapalenia oskrzeli – IBV – z rodzaju gamma-koronawirus)[1][2];
- zakaźne zapalenie otrzewnej kotów (wywołane przez wirus kociego koronawirusa z rodzaju alfa-koronawirus)[1][2][3];
- epidemiczna biegunka świń[1];
- wirusowe zapalenie żołądka i jelit u bydła[1].
Koronawirusy ludzkie
[edytuj | edytuj kod]Pierwsze szczepy koronawirusa ludzkiego zidentyfikowano w latach 60. XX wieku[4]. W 1962 roku wyizolowano szczep B814 pochodzący od dziecka z objawami przeziębienia, stosując hodowlę narządową pochodzącą z tchawicy. Z powodu zaginięcia próbki przed opracowaniem metod pozwalających na identyfikację gatunku, dokładne dane o B814 nie są znane, jednakże w kolejnych latach uzyskano następne izolaty kliniczne. Dwa z nich, 229E i OC43, zidentyfikowano jako osobne gatunki[1][5].
Kolejny gatunek ludzkiego koronawirusa, znacznie groźniejszy, zidentyfikowano w 2002 roku w chińskiej prowincji Guangdong. Wirus SARS-CoV, nazywany tak od wywoływanego przez niego zespołu ciężkiej niewydolności oddechowej (ang. Severe Acute Respiratory Syndrome), spowodował falę zachorowań i 775 potwierdzonych zgonów[1].
W 2004 roku prof. Krzysztof Pyrć oraz prof. Lia van der Hoek zidentyfikowali wirusa HCoV-NL63, a rok później grupa z Hongkongu opisała wirusa HCoV-HKU1. Oba te wirusy, podobnie jak 229E i OC43, są częstą przyczyną chorób górnych i dolnych dróg oddechowych[6][7][8]. Uważa się, że koronawirusy są odpowiedzialne za 10–20% wszystkich przeziębień[potrzebny przypis].
Obecnie wyróżnia się siedem gatunków koronawirusów wywołujących infekcję u człowieka:
- ludzki koronawirus 229E – α-koronawirus[2][1];
- ludzki koronawirus OC43 (HCoV OC43) – β-koronawirus[9][2][1];
- ludzki wirus SARS (SARS-CoV-1) – β-koronawirus[2][1];
- ludzki koronawirus NL63 (HCoV NL63) – α-koronawirus[2][1];
- ludzki koronawirus HKU1 (HCoV HKU1) – β-koronawirus[2][1];
- ludzki koronawirus bliskowschodniego zespołu oddechowego (wirus MERS, MERS-CoV, HCoV-EMC/2012, ludzki betakoronawirus 2c EMC/2012) – β-koronawirus[1][10][11];
- ludzki koronawirus SARS-CoV-2[12][13][14] (początkowo 2019-nCoV, zidentyfikowany w Wuhanie) – β-koronawirus[15].
W przeszłości opisywano również inne izolaty (takie jak B814, LP, EVS, OC16, OC37, OC44, OC48, AD, PA), które najprawdopodobniej należały do jednego z 4 opisanych gatunków, jednak nie można tego jednoznacznie stwierdzić ponieważ próbki zostały utracone[16]. Koronawirusy cechują się dużym rozpowszechnieniem i częstością występowania, dużym zróżnicowaniem genetycznym i częstym występowaniem rekombinacji genetycznych, co przy rosnących okazjach do kontaktów między człowiekiem i różnymi gatunkami zwierząt powoduje, że nowi przedstawiciele tej grupy będą pojawiać się, wywołując infekcje międzygatunkowe i sporadycznie rozprzestrzeniając się[14].
Zobacz też
[edytuj | edytuj kod]- choroba COVID-19
- wirus SARS-CoV-2
- wirus SARS
- pandemia COVID-19 w Polsce
- pandemia COVID-19 na świecie
Przypisy
[edytuj | edytuj kod]- ↑ a b c d e f g h i j k l m Krzysztof Pyrć, Ludzkie koronawirusy, „Postępy Nauk Medycznych”, XXVIII (4B), Borgis, 2015, s. 48–54 [dostęp 2020-01-21].
- ↑ a b c d e f g h i Coronaviridae – Positive Sense RNA Viruses – Positive Sense RNA Viruses (2011) [online], International Committee on Taxonomy of Viruses (ICTV) [dostęp 2020-01-21] (ang.).
- ↑ Javier A. Jaimes i inni, A Tale of Two Viruses: The Distinct Spike Glycoproteins of Feline Coronaviruses, „Viruses”, 12 (1), 2020, s. 83, DOI: 10.3390/v12010083, PMID: 31936749, PMCID: PMC7019228 [dostęp 2023-06-13] (ang.).
- ↑ Jeffrey S. Kahn, Kenneth McIntosh, History and recent advances in coronavirus discovery, „The Pediatric Infectious Disease Journal”, 24 (11 Suppl), 2005, S223–S227, DOI: 10.1097/01.inf.0000188166.17324.60, PMID: 16378050 [dostęp 2021-04-09] (ang.).
- ↑ J.S.M. Peiris, Coronaviruses, [w:] Douglas D. Richman, Richard J. Whitley, Frederick G. Hayden (red.), Clinical Virology, John Wiley & Sons, 2016, ISBN 978-1-68367-316-3 [dostęp 2020-01-22] (ang.).
- ↑ Brian M. Davis i inni, Human coronaviruses and other respiratory infections in young adults on a university campus: Prevalence, symptoms, and shedding, „Influenza and Other Respiratory Viruses”, 12 (5), 2018, s. 582–590, DOI: 10.1111/irv.12563, PMID: 29660826, PMCID: PMC6086849 [dostęp 2020-01-26] (ang.).
- ↑ Su-fen Zhang i inni, Epidemiology characteristics of human coronaviruses in patients with respiratory infection symptoms and phylogenetic analysis of HCoV-OC43 during 2010-2015 in Guangzhou, „PLOS ONE”, 13 (1), 2018, DOI: 10.1371/journal.pone.0191789, ISSN 1932-6203, PMID: 29377913, PMCID: PMC5788356 [dostęp 2020-01-26] (ang.).
- ↑ Zhi-Qi Zeng i inni, Epidemiology and clinical characteristics of human coronaviruses OC43, 229E, NL63, and HKU1: a study of hospitalized children with acute respiratory tract infection in Guangzhou, China, „European Journal of Clinical Microbiology & Infectious Diseases”, 37 (2), 2018, s. 363–369, DOI: 10.1007/s10096-017-3144-z, ISSN 1435-4373, PMID: 29214503, PMCID: PMC5780525 [dostęp 2020-01-26] (ang.).
- ↑ Lia van der Hoek i inni, Identification of a new human coronavirus, „Nature Medicine”, 10 (4), 2004, s. 368–373, DOI: 10.1038/nm1024, PMID: 15034574, PMCID: PMC7095789 [dostęp 2023-06-13] (ang.).
- ↑ Raoul J. de Groot i inni, Middle East Respiratory Syndrome Coronavirus (MERS-CoV): Announcement of the Coronavirus Study Group, „Journal of Virology”, 87 (14), 2013, s. 7790–7792, DOI: 10.1128/JVI.01244-13, ISSN 0022-538X, PMID: 23678167, PMCID: PMC3700179 [dostęp 2020-01-21] (ang.).
- ↑ Jasper F.W. Chan i inni, Is the discovery of the novel human betacoronavirus 2c EMC/2012 (HCoV-EMC) the beginning of another SARS-like pandemic?, „The Journal of Infection”, 65 (6), 2012, s. 477–489, DOI: 10.1016/j.jinf.2012.10.002, PMID: 23072791, PMCID: PMC7112628 [dostęp 2021-04-09] (ang.).
- ↑ Chih-Cheng Lai i inni, Severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) and coronavirus disease-2019 (COVID-19): The epidemic and the challenges, „International Journal of Antimicrobial Agents”, 55 (3), 2020, s. 105924, DOI: 10.1016/j.ijantimicag.2020.105924, PMID: 32081636, PMCID: PMC7127800 [dostęp 2021-04-09] (ang.).
- ↑ Surveillance case definitions for human infection with novel coronavirus (nCoV) [online], WHO, 10 stycznia 2020 [dostęp 2021-04-09] (ang.).
- ↑ a b Na Zhu i inni, A Novel Coronavirus from Patients with Pneumonia in China, 2019, „The New England Journal of Medicine”, 382 (8), 2020, s. 727–733, DOI: 10.1056/NEJMoa2001017, PMID: 31978945, PMCID: PMC7092803 [dostęp 2021-04-09] (ang.).
- ↑ Michael Letko, Andrea Marzi, Vincent Munster, Functional assessment of cell entry and receptor usage for SARS-CoV-2 and other lineage B betacoronaviruses, „Nature Microbiology”, 5 (4), 2020, s. 562–569, DOI: 10.1038/s41564-020-0688-y, PMID: 32094589, PMCID: PMC7095430 [dostęp 2021-04-09] (ang.).
- ↑ Lia van der Hoek, Krzysztof Pyrc, Ben Berkhout, Human coronavirus NL63, a new respiratory virus, „FEMS Microbiology Reviews”, 30 (5), 2006, s. 760–773, DOI: 10.1111/j.1574-6976.2006.00032.x, PMID: 16911043, PMCID: PMC7109777 [dostęp 2021-04-09] (ang.).
Bibliografia
[edytuj | edytuj kod]- Interna Harrisona. Wydawnictwo Czelej. Tom II. s. 1663, ISBN 83-88063-41-3.
