Syntaza ATP

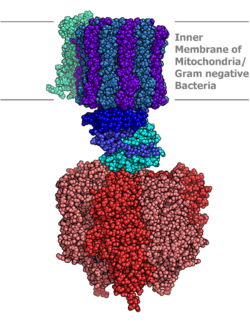
Syntaza ATP, (EC 7.1.2.2[1]) – enzym z grupy syntaz katalizujący reakcję wytwarzania ATP z ADP i fosforanu nieorganicznego Pi. Energia niezbędna do syntezy pochodzi z gradientu elektrochemicznego protonów - w wyniku tego gradientu następuje ruch protonów, a następnie ich energia przekształcana jest w energię wiązań chemicznych w ATP.
Reakcja syntezy ATP ma postać:
- ADP + Pi + 3H+out ⇌ ATP + H2O + 3H+in
Syntaza ATP znajduje się w wewnętrznej błonie mitochondriów, w błonach tylakoidów wewnątrz chloroplastów oraz w błonach komórkowych organizmów prokariotycznych. Enzym bierze udział w kluczowych procesach uzyskiwania energii przez organizmy żywe fosforylacji oksydacyjnej będącej głównym źródłem ATP wytwarzanego podczas oddychania komórkowego oraz fosforylacji fotosyntetycznej będącej źródłem ATP powstającego w fazie jasnej fotosyntezy.
Inhibitorem syntazy ATP wykorzystywanym w badaniach biologicznych jest antybiotyk – oligomycyna.
Rola fizjologiczna[edytuj | edytuj kod]

Wewnątrz błony mitochondrialnej (u eukariontów) lub w błonie komórkowej (u bakterii) następuje utlenianie NADH do NAD+. Uwolnione elektrony uczestniczą w łańcuchu oddechowym, aby napędzić przeniesienie protonów w poprzek błony (z macierzy mitochondrialnej do przestrzeni międzybłonowej) przez składające się na ten łańcuch odpowiednie przenośniki (pompy). W chloroplastach i błonach fotosyntetyzujących organizmów prokariotycznych protony przenoszone są ze stromy do wnętrza tylakoidu dzięki energii uzyskanej poprzez pochłonięcie kwantów światła przez odpowiednie kompleksy.
ATP wytwarzane jest z ADP i Pi (reszty ortofosforanowej) w wyniku działania syntazy ATP. Rotacja jej odpowiedniego segmentu umożliwia syntezę ATP. Energia niezbędna do syntezy dostarczana jest przez gradient elektrochemiczny. W procesie tym protony samoczynnie powracają z przestrzeni międzybłonowej lub wnętrza tylakoidu odpowiednio do macierzy mitochondrialnej lub stromy chloroplastu.
Budowa enzymu[edytuj | edytuj kod]
Ze względu na miejsce występowania wyróżnia się trzy podstawowe kompleksy syntazy ATP.
Są to:
- Syntaza ATP mitochondrialna określana jako FoF1 lub MFoF1
- Syntaza ATP chloroplastowa określana jako CFoF1
- Syntaza ATP prokariotyczna
Wszystkie trzy syntazy ATP zaliczane są do grupy F-ATPaz i mają zbliżoną budowę. Każda z nich składa się z dwóch domen. Pierwsza z nich – Fo – jest białkiem wewnątrzbłonowym tworzącym kanał jonowy dla jonów H+. Druga domena jest właściwą syntazą składająca się z kilkunastu polipeptydów tworzących kulista strukturę o średnicy ok. 100 Å.
Ewolucja[edytuj | edytuj kod]
Uważa się, że ewolucja syntazy ATP zachodziła modułowo. Obie domeny wraz ze swoimi wartościowościami połączyły się zyskując nową funkcję. Domena F1 wykazuje znaczne podobieństwo do heksamerycznej helikazy DNA, a domena Fo jest podobna do kompleksów tworzących motor molekularny napędzający wici komórek.
Heksamer α3β3 tworzący domenę F1 podobnie jak helikaza DNA składa się z obracającego się pierścienia z otworem pośrodku. U obu enzymów obroty pierścienia umożliwiają pełnienie funkcji. Helikaza DNA porusza się po helisie DNA przy jednoczesnej hydrolizie nukleotydów. Domena F1 wykorzystuje zmiany konformacyjne podczas obrotu podjednostki γ do przeprowadzenia reakcji enzymatycznej.
Jony H+ przepływające przez domenę Fo w bardzo podobny sposób jak przy napędzaniu motoru molekularnego poruszającego wicią. Ich wspólną cechą jest pierścień składający się z wielu alfa-helikalnych białek, które obracają się w stosunku do innych białek zużywając jednocześnie gradient protonowy jako źródło energii. Jest to jednakże dość wątły związek, ogólna struktura motorów molekularnych jest zdecydowanie większa, zawierają 30 polipeptydów, w porównaniu do 10, 11, lub 14 znanych polipeptydów domeny Fo.
Modułowa teoria pochodzenia syntazy ATP sugeruje, że dwie domeny o niezależnych funkcjach, helikaza DNA posiadająca właściwości ATPazy i motor molekularny napędzany siłą protonomotoryczną, mogły się połączyć i doprowadzić do odwrócenia właściwości ATPazy powstałej z helikazy DNA. Dalszy rozwój prowadziłby do powstania kompleksu syntazy ATP znanej dzisiaj. Alternatywnie kompleks helikazy DNA i motoru molekularnego mógł wykazywać początkowo aktywność ATPazy, która przenosiła jony H+ zużywając ATP. Dalsza ewolucja kompleksu mogłaby doprowadzić do odwrócenia przeprowadzanej reakcji i powstania funkcji spełnianej przez syntazę ATP.
Zobacz też[edytuj | edytuj kod]
- ATPazy – enzymy hydrolizujące ATP
- Paul Boyer – laureat Nagrody Nobla za badania syntezy ATP
Przypisy[edytuj | edytuj kod]
- ↑ Enzyme entry: EC 7.1.2.2, [w:] Enzyme nomenclature database, SIB Swiss Institute of Bioinformatics (ang.).
Bibliografia[edytuj | edytuj kod]
- P.D. Boyer, The binding change mechanism for ATP synthase – some probabilities and possibilities, „Biochimica et Biophysica Acta”, 1140 (3), 1993, s. 215–250, DOI: 10.1016/0005-2728(93)90063-L, PMID: 8417777.
- P.D. Boyer, The ATP synthase – a splendid molecular machine, „Annual Review of Biochemistry”, 66, 1997, s. 717–749, DOI: 10.1146/annurev.biochem.66.1.717, PMID: 9242922.
- R.J. Devenish i inni, Insights into ATP synthase assembly and function through the molecular genetic manipulation of subunits of the yeast mitochondrial enzyme complex, „Biochimica et Biophysica Acta”, 1458 (2-3), 2000, s. 428–442, DOI: 10.1016/S0005-2728(00)00092-X, PMID: 10838056.
- J. Velours i inni, Organisation of the yeast ATP synthase F0: a study based on cysteine mutants, thiol modification and cross-linking reagents, „Biochimica et Biophysica Acta”, 1458 (2-3), 2000, s. 443–456, DOI: 10.1016/S0005-2728(00)00093-1, PMID: 10838057.
- M.J. Gresser, J.A. Myers, P.D. Boyer, Catalytic site cooperativity of beef heart mitochondrial F1 adenosine triphosphatase. Correlations of initial velocity, bound intermediate, and oxygen exchange measurements with an alternating three-site model, „Journal of Biological Chemistry”, 257 (20), 1982, s. 12030–12038, PMID: 6214554.
