Kwas deoksyrybonukleinowy

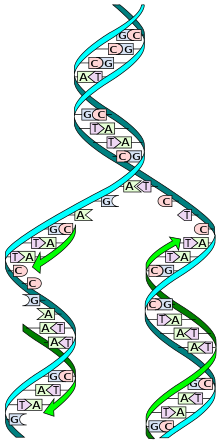
Kwas deoksyrybonukleinowy, DNA (z ang. deoxyribonucleic acid), daw. kwas dezoksyrybonukleinowy – wielkocząsteczkowy organiczny związek chemiczny z grupy kwasów nukleinowych. U eukariontów zlokalizowany jest przede wszystkim w jądrach komórek, u prokariontów – bezpośrednio w cytoplazmie, natomiast u wirusów – w kapsydach. Pełni rolę nośnika informacji genetycznej organizmów i wirusów.
Skład i budowa[edytuj | edytuj kod]
DNA jest liniowym, nierozgałęzionym biopolimerem, którego monomerami są deoksyrybonukleotydy[1]. Ich cząsteczki zbudowane są z pięciowęglowego cukru deoksyrybozy, którego grupa hydroksylowa znajdująca się przy ostatnim atomie węgla (5′) jest zestryfikowana resztą fosforanową, a pierwszy atom węgla (1′) połączony jest wiązaniem N-glikozydowym z jedną z czterech zasad azotowych, dwóch purynowych – adeniną (Ade lub A) i guaniną (Gua lub G) – oraz dwóch pirymidynowych: cytozyną (Cyt lub C) i tyminą (Thy lub T). Zasady te łącznie z deoksyrybozą tworzą deoksynukleozydy, odpowiednio dA, dG, dC i T[2][3][4].
Powszechnie spotykaną modyfikacją DNA jest występowanie 5-metylocytozyny (m5C) w wyniku metylacji cytozyny. W DNA niektórych wirusów, na przykład bakteriofagów PBS2, zamiast tyminy występuje uracyl (Ura), tworząc nukleozyd 2′-deoksyurydynę (dU)[5]. 2′-Deoksyurydyna powstaje też w wyniku deaminacji Cyt do Ura[6].
W skład cząsteczki DNA zwykle wchodzą dwa łańcuchy (DNA dwuniciowy – dsDNA), które biegną antyrównolegle (to znaczy koniec 5′ jednej nici leży naprzeciw końca 3′ drugiej nici). Łańcuchy zwijają się wokół wspólnej osi i tworzą prawoskrętną (A-DNA lub B-DNA) lub rzadziej lewoskrętną (Z-DNA) podwójną helisę[7]. Reszty cukrowe i fosforanowe, połączone ze sobą wiązaniem 5′-3′ fosfodiestrowym, znajdują się na zewnątrz helisy, natomiast zasady skierowane są do wnętrza i tworzą komplementarne pary zasad połączone według schematu:
- A=T
- G≡C
Zasady połączone są wiązaniami wodorowymi, a strukturę DNA stabilizują dodatkowo oddziaływania warstwowe pomiędzy sąsiednimi zasadami nukleinowymi[8].
Długość[edytuj | edytuj kod]
Po hipotetycznym całkowitym „rozpakowaniu” z chromosomów i połączeniu cząsteczek DNA w jedną nić, ludzkie DNA w przeciętnej komórce somatycznej miałoby około 2–2,4 m. Wynika to z rachunku: odległość pomiędzy parami zasad wynosi średnio 0,34 nm (0,34×10−9 m)[9], liczba par zasad w jądrze komórki diploidalnej wynosi w przybliżeniu 6–7 mld par zasad (jest to podwojona liczba par zasad komórki haploidalnej wynosząca 3×109[9]–3,5×109[10]), przemnożenie tych dwóch wartości daje wynik około 2 m. Najdłuższa rzeczywista cząsteczka DNA, chromosom 1[11], która zawiera 2,49×108 par zasad[12], ma długość około 8 cm.
Upakowanie w komórce[edytuj | edytuj kod]
Z powodu znacznej długości cząsteczek DNA konieczne jest ich upakowanie – do tego celu służą białka histonowe (u eukariontów) lub białka histonopodobne (u prokariontów). U eukariontów możliwe jest bardzo ścisłe upakowanie DNA w postaci chromosomu metafazowego, który jest formą najbardziej skondensowaną[13]. DNA występuje w niedzielącej się komórce eukariotycznej w postaci chromatyny, nazywanej niekiedy włóknem 30 nm[14].
Każda z nici DNA ma na jednym końcu (oznaczanym jako koniec 5′), przy ostatnim nukleotydzie trzy grupy fosforanowe przy węglu 5′ deoksyrybozy, a na drugim końcu (oznaczanym jako koniec 3′) ostatni nukleotyd posiada wolną grupę hydroksylową przy węglu 3′ deoksyrybozy. Ze względu na to, że helisa dwóch nici DNA jest spleciona w ten sposób, że jedna z nici zaczyna się od końca 5′, a druga od końca 3′, mówi się, że obie nici są względem siebie antyrównoległe[8].
Łańcuch nici DNA zawiera informację genetyczną:
- o kolejności aminokwasów w białkach (która stanowi niewielki ułamek sekwencji DNA – u człowieka około 1,5%)
- o sekwencji licznych RNA niekodujących
- regulacji ekspresji genów oraz
- sekwencji o niejasnym znaczeniu (stanowiących zdecydowaną większość sekwencji jądrowego DNA).
Sekwencja aminokwasów kodowana jest w postaci trójek nukleotydowych odpowiadających odpowiednim aminokwasom oraz kodonom terminacyjnym, podczas biosyntezy białka[15].
Zgodnie z pracą z 2016 roku informacja genetyczna w DNA jest zapisana nie tylko za sprawą sekwencji nukleotydów, ale także poprzez ułożenie nici DNA w nukleosomach[16][17].
Rodzaje DNA[edytuj | edytuj kod]
DNA rozróżnia się pod względem:
- pochodzenia w czasie – aDNA
- funkcji: cDNA, mtDNA, chlDNA/cpDNA
- struktury:
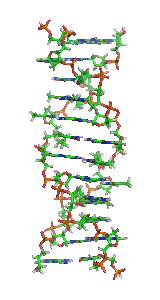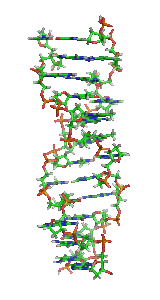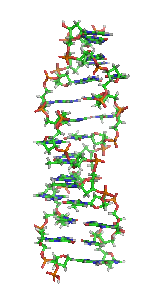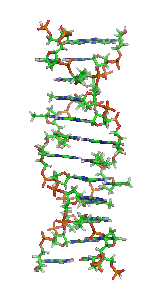
Najważniejsze z nich przedstawiono w tabeli:
| Rodzaj DNA | B-DNA | A-DNA | Z-DNA |
|---|---|---|---|
| liczba par zasad przypadająca na skręt helisy |
10,4 (ok. 10,2 dla tzw. wysp CpG) | 11 | 12 |
| kąt skręcania między sąsiednimi parami zasad (w stopniach) |
+34,6 | +39,0 | −30,0 |
| skok helisy (nm) | 3,54 | 2,53 | 4,56 |
| kierunek skręcania | prawoskrętna | prawoskrętna | lewoskrętna |
| średnica helisy (nm) | 2,37 | 2,55 | 1,84 |
| Fragment struktury | 
|

|
|
Historia poznania[edytuj | edytuj kod]

DNA zostało odkryte w roku 1869 przez Fryderyka Mieschera[24], lecz przez prawie 100 lat jego struktura pozostawała zagadką. Autorami modelu podwójnej helisy DNA są James Watson i Francis Crick, na podstawie zdjęć z rentgenowskich badań strukturalnych wykonanych przez Rosalind Franklin oraz Maurice’a Wilkinsa[25]. Pracowali oni wtedy w Medical Reserch Council Unity w Cavendish Laboratory w Cambridge[26]. Za odkrycie w roku 1953 struktury DNA Watson, Crick i Wilkins otrzymali w 1962 Nagrodę Nobla (Rosalind Franklin zmarła na raka w 1958)[27].
W 1961 miało miejsce odkrycie zasad kodu genetycznego przez Holleya, Khoranę i Nirenberga, a w roku 1977 opracowanie metody sekwencjonowania DNA przez zespoły badawcze Waltera Gilberta i Fredericka Sangera.
Porównanie DNA za pomocą RFLP jest obecnie powszechnie stosowane w kryminalistyce. Po raz pierwszy profil DNA w kryminalistyce został wykorzystany w roku 1986 przez brytyjską policję i podejrzany został uniewinniony. Pierwszym skazanym na podstawie dowodu w postaci DNA był w roku 1987 brytyjski piekarz Colin Pitchfork[28].
Zobacz też[edytuj | edytuj kod]
Zagadnienia dotyczące budowy, struktury i funkcji DNA:
Zagadnienia genetyczne:
Eksperymenty:
Inne:
Przypisy[edytuj | edytuj kod]
- ↑ The Biosynthesis of Cell Constituents, [w:] Geoffrey M. Cooper, The Cell: A Molecular Approach, wyd. 2, Washington, D.C.: ASM Press, 2000, ISBN 0-87893-119-8, OCLC 43708665 [dostęp 2022-04-16] (ang.).
- ↑ Structures of Nucleic Acids. W: D.W.S. Wong: The ABCs of Gene Cloning. Wyd. 2. 2006. ISBN 978-0-387-28679-2.
- ↑ Z.A. Shabarova, A.A. Bogdanov: Advanced Organic Chemistry of Nucleic Acids. Wiley VCH, 1994, s. 1–6.
- ↑ J. Koolman, K.H. Roehm: Color Atlas of Biochemistry. Wyd. 2. Thieme, 2005, s. 80–87. ISBN 3-13-100372-3.
- ↑ I. Takahashi, J. Marmur, Replacement of thymidylic acid by deoxyuridylic acid in the deoxyribonucleic acid of a transducing phage for Bacillus subtilis, „Nature”, 197, 1963, s. 794–795, DOI: 10.1038/197794a0, PMID: 13980287 [dostęp 2022-04-16] (ang.).
- ↑ Torkild Visnes i inni, Uracil in DNA and its processing by different DNA glycosylases, „Philosophical Transactions of the Royal Society of London. Series B. Biological Sciences”, 364 (1517), 2009, s. 563–568, DOI: 10.1098/rstb.2008.0186, PMID: 19008197, PMCID: PMC2660913 [dostęp 2022-04-16] (ang.).
- ↑ Stryer, Berg i Tymoczko 2011 ↓, Rozdział 28, Sekcja 28.1.
- ↑ a b Stryer, Berg i Tymoczko 2011 ↓, Rozdział 4, Sekcja 4.2.
- ↑ a b How big is the DNA? A scaling excercise. [dostęp 2015-02-09]. [zarchiwizowane z tego adresu (2006-09-10)].
- ↑ Robert Kincaid Murray, Daryl K. Granner, Victor W. Rodwell: Biochemia Harpera ilustrowana. Wyd. 4. Warszawa: Wydawnictwo Lekarskie PZWL, 2008, s. 392. ISBN 978-83-200-3573-5.
- ↑ Vega Genome Browser 52 – Homo sapiens – whole genome [online], Vega Genome Browser [dostęp 2022-04-16] (ang.).
- ↑ Vega Genome Browser 52 – chromosome summary – chromosome 1 [online], Vega Genome Browser [dostęp 2022-04-16] (ang.).
- ↑ Clare O’Connor: Karyotyping. [w:] Scitable [on-line]. Nature Education, 2014. [dostęp 2022-04-16]. (ang.).
- ↑ DNA Structure. Chemistry LibreTexts. (ang.).
- ↑ Stryer, Berg i Tymoczko 2011 ↓, Rozdział 4, Sekcja 4.5.
- ↑ Second layer of information in DNA confirmed. Leiden University, 2016-06-08. [dostęp 2016-06-09]. (ang.).
- ↑ Behrouz Eslami-Mossallam i inni, Multiplexing Genetic and Nucleosome Positioning Codes: A Computational Approach, „PLoS One”, 11 (6), 2016, art. nr e0156905, DOI: 10.1371/journal.pone.0156905, PMID: 27272176, PMCID: PMC4896621 [dostęp 2022-04-16] (ang.).
- ↑ L. van Dam, M.H. Levitt, BII nucleotides in the B and C forms of natural-sequence polymeric DNA: A new model for the C form of DNA, „Journal of Molecular Biology”, 304 (4), 2000, s. 541–561, DOI: 10.1006/jmbi.2000.4194, PMID: 11099379 [dostęp 2022-04-16] (ang.).
- ↑ J.M. Vargason, B.F. Eichman, P.S. Ho, The extended and eccentric E-DNA structure induced by cytosine methylation or bromination, „Nature Structural Biology”, 7 (9), 2000, s. 758–761, DOI: 10.1038/78985, PMID: 10966645 [dostęp 2022-04-16] (ang.).
- ↑ Junhua Zhao i inni, Non-B DNA structure-induced genetic instability and evolution, „Cellular and molecular life sciences: CMLS”, 67 (1), 2010, s. 43–62, DOI: 10.1007/s00018-009-0131-2, PMID: 19727556, PMCID: PMC3017512 [dostęp 2022-04-16] (ang.).
- ↑ J.F. Allemand i inni, Stretched and overwound DNA forms a Pauling-like structure with exposed bases, „Proceedings of the National Academy of Sciences of the United States of America”, 95 (24), 1998, s. 14152–14157, DOI: 10.1073/pnas.95.24.14152, PMID: 9826669, PMCID: PMC24342 [dostęp 2022-04-16] (ang.).
- ↑ Anirban Ghosh, Manju Bansal, A glossary of DNA structures from A to Z, „Acta Crystallographica. Section D, Biological Crystallography”, 59 (Pt 4), 2003, s. 620–626, DOI: 10.1107/s0907444903003251, PMID: 12657780 [dostęp 2022-04-16] (ang.).
- ↑ E. Palecek, Local supercoil-stabilized DNA structures, „Critical Reviews in Biochemistry and Molecular Biology”, 26 (2), 1991, s. 151–226, DOI: 10.3109/10409239109081126, PMID: 1914495 [dostęp 2022-04-16] (ang.).
- ↑ Ralf Dahm, Discovering DNA: Friedrich Miescher and the early years of nucleic acid research, „Human Genetics”, 122 (6), 2008, s. 565–581, DOI: 10.1007/s00439-007-0433-0, PMID: 17901982 [dostęp 2022-04-16] (ang.).
- ↑ Archiwalne artykuły z Nature z 1953 roku dotyczące odkrycia struktury DNA [online], 2003 [zarchiwizowane z adresu 2003-11-06] (ang.).
- ↑ Ferris Jabr. Święto nauki. „Świat Nauki”. Nr 7 (239), s. 42–51, 2011. Prószyński Media. ISSN 0867-6380. za: F.H.C Crick. Struktura materiału dziedzicznego. „Świat Nauki”, 1954. Prószyński Media. ISSN 0867-6380.
- ↑ The Nobel Prize in Physiology or Medicine 1962. Nobelprize. [dostęp 2013-08-03].
- ↑ Adam Skorupiński, DNA – zbawienie czy przekleństwo? [online], Londynek.net, 6 lutego 2008 [dostęp 2022-04-16].
Bibliografia[edytuj | edytuj kod]
- Lubert Stryer, Jeremy Berg, John Tymoczko: Biochemia. Wyd. 4. Wydawnictwo Naukowe PWN, 2011. ISBN 978-83-01-15811-8.
