Haplogrupa R1a1 (Y-DNA): Różnice pomiędzy wersjami
| [wersja nieprzejrzana] | [wersja przejrzana] |
Nowa mapa rozmieszczenia haplogrupy R1a |
źródła/przypisy |
||
| Linia 22: | Linia 22: | ||
[[File:R1a Europa centralna.png|thumb|Rozmieszczenie haplogrupy R1a w Europie środkowej]] |
[[File:R1a Europa centralna.png|thumb|Rozmieszczenie haplogrupy R1a w Europie środkowej]] |
||
'''R1a1 (M17)''' – w [[genetyka|genetyce]] [[haplogrupa]] występująca w męskim [[chromosom]]ie [[chromosom Y|Y]]. |
'''R1a1 (M17)''' – w [[genetyka|genetyce]] [[haplogrupa]] występująca w męskim [[chromosom]]ie [[chromosom Y|Y]]. |
||
W Europie występuje najczęściej wśród [[Słowianie|ludów słowiańskich]]: [[Serbołużyczanie|Serbów Łużyckich]] (63%), [[Polacy|Polaków]] (55%), [[Ukraińcy|Ukraińców]] (43%), [[Rosjanie|Rosjan]] (46%) i [[Białorusini|Białorusinów]] (50%) oraz [[Węgrzy|Węgrów]] ( |
W Europie występuje najczęściej wśród [[Słowianie|ludów słowiańskich]]: [[Serbołużyczanie|Serbów Łużyckich]] (63%), [[Polacy|Polaków]] (55%), [[Ukraińcy|Ukraińców]] (43%), [[Rosjanie|Rosjan]] (46%) i [[Białorusini|Białorusinów]] (50%)<ref name="doi:10.1371/journal.pone.0066499 ">{{Cytuj pismo | autor= Alena Kushniarevich,Larysa Sivitskaya, Nina Danilenko,Tadeush Novogrodskii,Iosif Tsybovsky,Anna Kiseleva,Svetlana Kotova, Gyaneshwer Chaubey,Ene Metspalu,Hovhannes Sahakyan,Ardeshir Bahmanimehr,Maere Reidla,Siiri Rootsi,Jüri Parik,Tuuli Reisberg, Alessandro Achilli,Baharak Hooshiar Kashani,Francesca Gandini,Anna Olivieri,Doron M. Behar,Antonio Torroni,Oleg Davydenko,Richard Villems | tytuł= Uniparental Genetic Heritage of Belarusians: Encounter of Rare Middle Eastern Matrilineages with a Central European Mitochondrial DNA Pool | rok=2013 | czasopismo= PLoS ONE | doi=10.1371/journal.pone.0066499 | wydanie= 6 | wolumin= 8 | miesiąc=czerwiec | pmid= | strony=14}}</ref> oraz [[Węgrzy|Węgrów]] (25%)<ref name="doi:10.1086/383203">{{Cytuj pismo | autor= Antónia Völgyi , Andrea Zalán , Enikő Szvetnik, Horolma Pamjav * | tytuł=Announcement of population data Hungarian population data for 11 Y-STR and 49 Y-SNP markers | rok=2009 | czasopismo=Forensic Science International: Genetics | doi= 10.1016/j.fsigen.2008.04.006 | wydanie= 2 | wolumin= 3 | miesiąc=marzec | pmid= | strony=}}</ref> przynależących do tureckiej grupy językowej. Istotny procent R1a występuje także u Skandynawów: [[Norwegowie|Norwegów]] (25-30%),[[Szwedzi|Szwedów]] (15-20%), [[Duńczycy|Duńczyków]] (10-15%) oraz u [[Niemcy|Niemców]] (15-20%). Ludy bałtyckie również charakteryzują się wysoką częstością występowania haplogrupy R1a: [[Litwini]] (34-45%), [[Łotysze]] (40%), [[Estończycy]] (33%)<ref name=" doi:10.1086/383203 ">{{Cytuj pismo | autor= Kristiina Tambets 1,,, Siiri Rootsi 1, Toomas Kivisild 1, Hela Help 1, Piia Serk 1, Eva-Liis Loogväli 1, Helle-Viivi Tolk 1, Maere Reidla 1, Ene Metspalu 1, Liana Pliss1, 2, Oleg Balanovsky 1,4, Andrey Pshenichnov 1,4, Elena Balanovska 4, Marina Gubina 1,5, Sergey Zhadanov 1,5, Ludmila Osipova 5, Larisa Damba 1,5, Mikhail Voevoda 5, Ildus Kutuev 1,6, Marina Bermisheva 1,6, Elza Khusnutdinova 6, Vladislava Gusar 1,7, Elena Grechanina 7, Jüri Parik 1, Erwan Pennarun 1, Christelle Richard 8, Andre Chaventre 8, Jean-Paul Moisan 8, Lovorka Barać 1,9, Marijana Peričić 1,9, Pavao Rudan 9, Rifat Terzić 10, Ilia Mikerezi 11, Astrida Krumina 3,Viesturs Baumanis 2, Slawomir Koziel 12, Olga Rickards 13, Gian Franco De Stefano 13, Nicholas Anagnou 14,16, Kalliopi I. Pappa 14,16, Emmanuel Michalodimitrakis 15, Vladimir Ferák 17, Sandor Füredi 18, Radovan Kome l19, Lars Beckman 20 and Richard Villems 1| tytuł= "The Western and Eastern Roots of the Saami—the Story of Genetic 'Outliers' Told by Mitochondrial DNA and Y Chromosomes" | rok=2004 | czasopismo= American Journal of Human Genetics | doi= 10.1086/383203 | wydanie= 4| wolumin= 74 | miesiąc=kwiecień | pmid= | strony=661–682}}</ref>. |
||
Poza Europą R1a występuje najczęściej wśród mieszkańców Indii Północnych (48-73%) i [[Azja Środkowa|Azji Środkowej]]: [[języki irańskie|irańskojęzycznych]] Iszkaszimów (68%), [[Tadżycy|Tadżyków]] (30%) i [[Pasztuni|Pasztunów]] (40-45%), [[Ludy tureckie|ludów tureckich]]: [[Kirgizi|Kirgizów]] (63%) i [[Ałtajczycy|Ałtajczyków]] (38-53%). |
Poza Europą R1a występuje najczęściej wśród mieszkańców Indii Północnych (48-73%) i [[Azja Środkowa|Azji Środkowej]]: [[języki irańskie|irańskojęzycznych]] Iszkaszimów (68%), [[Tadżycy|Tadżyków]] (30%) i [[Pasztuni|Pasztunów]] (40-45%), [[Ludy tureckie|ludów tureckich]]: [[Kirgizi|Kirgizów]] (63%) i [[Ałtajczycy|Ałtajczyków]] (38-53%). |
||
Wersja z 10:42, 18 sie 2013
Haplogrupa R1a1 (M17) Plik:Distribution Haplogroup R1a Y-DNA.svg 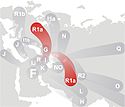
| |
| Pochodzenie | |
|---|---|
| Data powstania |
15 000 lat p.n.e. |
| Miejsce powstania |
Stepy Euroazji (Europa południowo-wschodnia, Północny Kaukaz, Azja Środkowa) |
| Mutacja wyjściowa |
Haplogrupa R1 |
| Występowanie | |
| Mutacje |
SRY-1532 |
| Etnosy |
Polacy 55%, Serbołużyczanie 63%, Ukraińcy 43%, Rosjanie 46%, Białorusini 50%, Norwegowie 25-30%, Litwini 34-45%, Łotysze 40%, Estończycy 33%, Węgrzy 30%, Niemcy i Szwedzi 15-20%, ludy Północnych Indii 48%-72%, Tadżycy 30%, Kirgizi 63%, Ałtajczycy 38%-53%, Pasztuni 40%-45%, . |
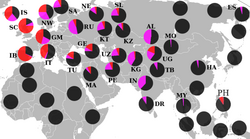

R1a1 (M17) – w genetyce haplogrupa występująca w męskim chromosomie Y. W Europie występuje najczęściej wśród ludów słowiańskich: Serbów Łużyckich (63%), Polaków (55%), Ukraińców (43%), Rosjan (46%) i Białorusinów (50%)[1] oraz Węgrów (25%)[2] przynależących do tureckiej grupy językowej. Istotny procent R1a występuje także u Skandynawów: Norwegów (25-30%),Szwedów (15-20%), Duńczyków (10-15%) oraz u Niemców (15-20%). Ludy bałtyckie również charakteryzują się wysoką częstością występowania haplogrupy R1a: Litwini (34-45%), Łotysze (40%), Estończycy (33%)[2]. Poza Europą R1a występuje najczęściej wśród mieszkańców Indii Północnych (48-73%) i Azji Środkowej: irańskojęzycznych Iszkaszimów (68%), Tadżyków (30%) i Pasztunów (40-45%), ludów tureckich: Kirgizów (63%) i Ałtajczyków (38-53%).
Rozprzestrzenienie
Na kontynencie europejskim dominuje wśród Słowian i Węgrów. Obecnie występuje z częstotliwością 40% w krajach słowiańskich z małymi odchyleniami (60% Polska, Węgry[3]). Inne badania podają 20% dla Węgrów[4]. W Europie procentowo najwięcej (63%) R1a1 jest na Łużycach, ale tylko wśród resztek słowiańskiej ludności[5].
R1a1 występuje w Europie najczęściej wśród Serbołużyczan (63%), a najwięcej jego nosicieli jest wśród Rosjan (47%, ponad 50 mln). Natomiast wariancja sekwencji rejonu mikrosatelitarnego chromosomu Y jest większa dla polskiej populacji niż kumulacyjnie dla reszty europejskiej populacji[6]. Rozprzestrzenienie R1a1 w Europie przedstawia mapa C[7] z publikacji Marijany Pericić[4]. Ona też znalazła datujące się na paleolit mikrosatelitarne zgrupowanie w badanej populacji w Macedonii, co świadczy że nosiciele R1a1 zamieszkiwali badany rejon od paleolitu. Mikrosatelitarna zmienność jest w Macedonii mniejsza niż w Polsce, a procentowy udział R1a1 trzykrotnie niższy. Badania te rzucają światło na aspekty 'wędrówki ludów' – tezy lansowanej szczególnie przez niemieckojęzycznych badaczy przełomu XIX i XX wieku, i ciągle silnie powtarzanej. Wędrówki te – o ile następowały – nie były tak masowe. Mikrosatelitarne klustery świadczą, że nosiciele R1a1 wędrowali po Eurazji, ale z Europy, a ponadto stale ich część pozostawała w badanej okolicy. Sekwencjonowanie rejonu mikrosatelitarnego chromosomu Y pozwala uzyskać dużo bardziej precyzyjne dane niż tylko mapowanie alleli. Badania te przeprowadza się jednocześnie. Chromosom Y dziedziczy się tylko w prostej linii od ojca, dziada, pradziada itd. Ten, u kogo po raz pierwszy wystąpiła haplogrupa R1a1, jest w 100% praojcem każdego mężczyzny, który posiada R1a1. R1a1 powstała w ewolucji ludzi tylko jeden raz u tylko jednego człowieka. Wszyscy posiadacze R1a1 to jego potomkowie w linii ojcowskiej.
Również w krajach Europy północnej R1a1 występuje z częstością 10-20%, przy czym najwyższe częstości odnotowano w Norwegii i Islandii, ale jest to mniej niż połowa częstości jej występowania u Polaków. Istnieją przypuszczenia, iż ta haplogrupa została tam rozprzestrzeniona przez wikingów i Słowian.
W Indiach haplogrupa R1a1 jest spotykana zarówno wśród populacji najwyższych kast (43%)[potrzebny przypis], jak i plemion (12%-4%). Znacznie wyższe procenty są wśród ludów na północ od Indii[8], co może wskazywać na kierunek rozprzestrzeniania się R1a1 do Indii.
Pochodzenie
Mutacja tworząca R1a1 powstała u osoby zamieszkującej stepy Euroazji ok. 15 000 lat temu (Europa południowo-wschodnia, Północny Kaukaz lub Azja Środkowa). Według niektórych teorii do mutacji doszło w cieplejszej strefie klimatycznej nazywanej ukraińską ostoją LGM. Badacz Semino pierwszy wykazał związek R1a1 z kurhanowcami (ich potomkowie to Scytowie i potem Sarmaci), utożsamianymi z ludami posługującymi się wczesnymi językami praindoeuropejskimi, oraz pierwszymi, którzy oswajali konie, dosiedli ich i rozprzestrzenili haplogrupę R1a1 po Europie i Azji[3].
Istnieją też poglądy, że mutacja powstała na terenie Indii lub Afganistanu i stamtąd rozprzestrzeniła się w przeciwnym kierunku[9].
Ewolucja haplogrup
R1a1 jest podgrupą haplogrupy R (M207). Jest blisko spokrewniona z haplogrupą R1b (M343), która dominuje w Europie zachodniej, i nieco dalej spokrewniona z haplogrupą R2 (M124).
- Haplogrupa R (M207)
- Haplogrupa R1 (M173)
- Haplogrupa R1a (SRY10831.2-)
- Haplogrupa R1a1 (M17)
- R1a1a M56
- R1a1b M157
- R1a1c M64.2, M87
- Haplogrupa R1a*
- Haplogrupa R1a1 (M17)
- Haplogrupa R1b (M343)
- Haplogrupa R1a (SRY10831.2-)
- Haplogrupa R2 (M124)
- Haplogrupa R1 (M173)
- ↑ Alena Kushniarevich,Larysa Sivitskaya, Nina Danilenko,Tadeush Novogrodskii,Iosif Tsybovsky,Anna Kiseleva,Svetlana Kotova, Gyaneshwer Chaubey,Ene Metspalu,Hovhannes Sahakyan,Ardeshir Bahmanimehr,Maere Reidla,Siiri Rootsi,Jüri Parik,Tuuli Reisberg, Alessandro Achilli,Baharak Hooshiar Kashani,Francesca Gandini,Anna Olivieri,Doron M. Behar,Antonio Torroni,Oleg Davydenko,Richard Villems. Uniparental Genetic Heritage of Belarusians: Encounter of Rare Middle Eastern Matrilineages with a Central European Mitochondrial DNA Pool. „PLoS ONE”. 8 (6), s. 14, czerwiec 2013. DOI: 10.1371/journal.pone.0066499.
- ↑ a b Antónia Völgyi , Andrea Zalán , Enikő Szvetnik, Horolma Pamjav *. Announcement of population data Hungarian population data for 11 Y-STR and 49 Y-SNP markers. „Forensic Science International: Genetics”. 3 (2), marzec 2009. DOI: 10.1016/j.fsigen.2008.04.006. Błąd w przypisach: Nieprawidłowy znacznik
<ref>; nazwę „doi:10.1086/383203” zdefiniowano więcej niż raz z różną zawartościąBŁĄD PRZYPISÓW - ↑ a b Semino O., Passarino G., Oefner PJ., Lin AA., Arbuzova S., Beckman LE., De Benedictis G., Francalacci P., Kouvatsi A., Limborska S., Marcikiae M., Mika A., Mika B., Primorac D., Santachiara-Benerecetti AS., Cavalli-Sforza LL., Underhill PA. The genetic legacy of Paleolithic Homo sapiens sapiens in extant Europeans: a Y chromosome perspective.. „Science (New York, N.Y.)”. 5494 (290), s. 1155–9, listopad 2000. PMID: 11073453.
- ↑ a b Pericić M., Lauc LB., Klarić IM., Rootsi S., Janićijevic B., Rudan I., Terzić R., Colak I., Kvesić A., Popović D., Sijacki A., Behluli I., Dordevic D., Efremovska L., Bajec DD., Stefanović BD., Villems R., Rudan P. High-resolution phylogenetic analysis of southeastern Europe traces major episodes of paternal gene flow among Slavic populations.. „Molecular biology and evolution”. 10 (22), s. 1964–75, październik 2005. DOI: 10.1093/molbev/msi185. PMID: 15944443.
- ↑ Behar DM., Thomas MG., Skorecki K., Hammer MF., Bulygina E., Rosengarten D., Jones AL., Held K., Moses V., Goldstein D., Bradman N., Weale ME. Multiple origins of Ashkenazi Levites: Y chromosome evidence for both Near Eastern and European ancestries.. „American journal of human genetics”. 4 (73), s. 768–79, październik 2003. DOI: 10.1086/378506. PMID: 13680527.
- ↑ Pawłowski R., Dettlaff-Kakol A., Maciejewska A., Paszkowska R., Reichert M., Jezierski G. [Population genetics of 9 Y-chromosome STR loci w Northern Poland]. „Archiwum medycyny sądowej i kryminologii”. 4 (52). s. 261–77. PMID: 14669672.
- ↑ http://mbe.oxfordjournals.org/content/vol22/issue10/images/large/molbiolevolmsi185f05_ht.jpeg
- ↑ Wells RS., Yuldasheva N., Ruzibakiev R., Underhill PA., Evseeva I., Blue-Smith J., Jin L., Su B., Pitchappan R., Shanmugalakshmi S., Balakrishnan K., Read M., Pearson NM., Zerjal T., Webster MT., Zholoshvili I., Jamarjashvili E., Gambarov S., Nikbin B., Dostiev A., Aknazarov O., Zalloua P., Tsoy I., Kitaev M., Mirrakhimov M., Chariev A., Bodmer WF. The Eurasian heartland: a continental perspective on Y-chromosome diversity.. „Proceedings of the National Academy of Sciences of the United States of America”. 18 (98), s. 10244–9, sierpień 2001. DOI: 10.1073/pnas.171305098. PMID: 11526236.
- ↑ Kivisild T., Rootsi S., Metspalu M., Mastana S., Kaldma K., Parik J., Metspalu E., Adojaan M., Tolk HV., Stepanov V., Gölge M., Usanga E., Papiha SS., Cinnioğlu C., King R., Cavalli-Sforza L., Underhill PA., Villems R. The genetic heritage of the earliest settlers persists both in Indian tribal and caste populations.. „American journal of human genetics”. 2 (72), s. 313–32, luty 2003. DOI: 10.1086/346068. PMID: 12536373.
