SARS-CoV-2: Różnice pomiędzy wersjami
| [wersja przejrzana] | [wersja nieprzejrzana] |
→Profilaktyka: Nareszcie ogłoszono obowiązek. Patrz moje inne komentarze. |
1. Uaktualnienia w sekcji Drogi szerzenia infekcji. 2. Utworzona sekcja Patogeneza \ Immunopatologia i dodane teksty według najaktualniejszych publikacji. 3. Większość tekstów z sekcji Odpowiedź immunologiczna przeniesiona została do sekcji Patogeneza \ Immunopatologia, utworzone podsekcje w celu posortowania tematycznego. 4. Informacje o wpływie grupy krwi na możliwość zakażenia wirusem przeniesione do sekcji zakaźność. |
||
| Linia 258: | Linia 258: | ||
20 stycznia 2020 r. Narodowa Komisja Zdrowia Chin ogłosiła, że możliwe jest przekazywanie wirusa z człowieka na człowieka<ref>{{Cytuj |autor = Nicky Phillips, Smriti Mallapaty, David Cyranoski |tytuł = How quickly does the Wuhan virus spread? |czasopismo = Nature |data = 2020-01-21 |data dostępu = 2020-03-11 |issn = 0028-0836 |s = d41586–020–00146-w |doi = 10.1038/d41586-020-00146-w |url = http://www.nature.com/articles/d41586-020-00146-w |język = en}}</ref><ref>{{Cytuj |autor = Zhangkai J. Cheng, Jing Shan |tytuł = 2019 Novel coronavirus: where we are and what we know |czasopismo = Infection |data = 2020-02-18 |data dostępu = 2020-03-11 |issn = 0300-8126 |doi = 10.1007/s15010-020-01401-y |url = http://link.springer.com/10.1007/s15010-020-01401-y |język = en}}</ref>, zwłaszcza gdy dwie osoby są ze sobą w bliskim kontakcie (odległość mniejsza niż 1,8 m<ref name=":2">{{Cytuj |autor = CDC, CDC |tytuł = Coronavirus Disease 2019 (COVID-19) – Transmission |data = 2020-03-04 |data dostępu = 2020-03-11 |opublikowany = Centers for Disease Control and Prevention |url = https://www.cdc.gov/coronavirus/2019-ncov/about/transmission.html |język = en}}</ref> lub odległość mniejsza niż 1–2 m<ref name=":3">{{Cytuj |tytuł = RKI - Coronavirus SARS-CoV-2 – Antworten auf häufig gestellte Fragen zum Coronavirus SARS-CoV-2 |data dostępu = 2020-03-11 |opublikowany = www.rki.de |url = https://www.rki.de/SharedDocs/FAQ/NCOV2019/FAQ_Liste.html}}</ref>). Uważa się, że rozprzestrzenia się on podobnie jak inne patogeny chorób układu oddechowego [[Droga szerzenia się zakażenia|drogą kropelkową]]<ref name=":0" /><ref name=":2" /><ref name=":3" /><ref>{{Cytuj |autor = |tytuł = Beschluss 1/2020 des ABAS vom 19.2.2020 und Begründung zur vorläufigen Einstufung des Virus SARS-CoV-2 in Risikogruppe 3 und Empfehlungen zu nicht gezielten Tätigkeiten (Labordiagnostik) und gezielten Tätigkeiten mit SARS-CoV-2. |data = |typ nośnika = pdf |url = https://www.baua.de/DE/Aufgaben/Geschaeftsfuehrung-von-Ausschuessen/ABAS/pdf/SARS-CoV-2.pdf}}</ref>. Do tej pory wirus został wykryty w wydzielinie z nosa i gardła, w plwocinie, moczu, stolcu, cieczy łzowej i krwi<ref>{{Cytuj |autor = Jasper Fuk-Woo Chan, Shuofeng Yuan, Kin-Hang Kok, Kelvin Kai-Wang To, Hin Chu |tytuł = A familial cluster of pneumonia associated with the 2019 novel coronavirus indicating person-to-person transmission: a study of a family cluster |czasopismo = The Lancet |data = 2020-02 |data dostępu = 2020-03-11 |wolumin = 395 |numer = 10223 |s = 514–523 |doi = 10.1016/S0140-6736(20)30154-9 |url = https://linkinghub.elsevier.com/retrieve/pii/S0140673620301549 |język = en}}</ref><ref>{{Cytuj |autor = Michelle L. Holshue, Chas DeBolt, Scott Lindquist, Kathy H. Lofy, John Wiesman |tytuł = First Case of 2019 Novel Coronavirus in the United States |czasopismo = New England Journal of Medicine |data = 2020-03-05 |data dostępu = 2020-03-11 |issn = 0028-4793 |wolumin = 382 |numer = 10 |s = 929–936 |doi = 10.1056/NEJMoa2001191 |url = http://www.nejm.org/doi/10.1056/NEJMoa2001191 |język = en}}</ref><ref>{{Cytuj |autor = Jianhua Xia, Jianping Tong, Mengyun Liu, Ye Shen, Dongyu Guo |tytuł = Evaluation of coronavirus in tears and conjunctival secretions of patients with SARS-CoV-2 infection |czasopismo = Journal of Medical Virology |data = 2020-02-26 |data dostępu = 2020-03-11 |issn = 0146-6615 |s = jmv.25725 |doi = 10.1002/jmv.25725 |url = https://onlinelibrary.wiley.com/doi/abs/10.1002/jmv.25725 |język = en}}</ref><ref>{{Cytuj |autor = Yu Chen, Lanjuan Li |tytuł = SARS-CoV-2: virus dynamics and host response |czasopismo = The Lancet Infectious Diseases |data = 2020-03 |data dostępu = 2020-03-26 |s = S1473309920302358 |doi = 10.1016/S1473-3099(20)30235-8 |url = https://linkinghub.elsevier.com/retrieve/pii/S1473309920302358 |język = en}}</ref>. |
20 stycznia 2020 r. Narodowa Komisja Zdrowia Chin ogłosiła, że możliwe jest przekazywanie wirusa z człowieka na człowieka<ref>{{Cytuj |autor = Nicky Phillips, Smriti Mallapaty, David Cyranoski |tytuł = How quickly does the Wuhan virus spread? |czasopismo = Nature |data = 2020-01-21 |data dostępu = 2020-03-11 |issn = 0028-0836 |s = d41586–020–00146-w |doi = 10.1038/d41586-020-00146-w |url = http://www.nature.com/articles/d41586-020-00146-w |język = en}}</ref><ref>{{Cytuj |autor = Zhangkai J. Cheng, Jing Shan |tytuł = 2019 Novel coronavirus: where we are and what we know |czasopismo = Infection |data = 2020-02-18 |data dostępu = 2020-03-11 |issn = 0300-8126 |doi = 10.1007/s15010-020-01401-y |url = http://link.springer.com/10.1007/s15010-020-01401-y |język = en}}</ref>, zwłaszcza gdy dwie osoby są ze sobą w bliskim kontakcie (odległość mniejsza niż 1,8 m<ref name=":2">{{Cytuj |autor = CDC, CDC |tytuł = Coronavirus Disease 2019 (COVID-19) – Transmission |data = 2020-03-04 |data dostępu = 2020-03-11 |opublikowany = Centers for Disease Control and Prevention |url = https://www.cdc.gov/coronavirus/2019-ncov/about/transmission.html |język = en}}</ref> lub odległość mniejsza niż 1–2 m<ref name=":3">{{Cytuj |tytuł = RKI - Coronavirus SARS-CoV-2 – Antworten auf häufig gestellte Fragen zum Coronavirus SARS-CoV-2 |data dostępu = 2020-03-11 |opublikowany = www.rki.de |url = https://www.rki.de/SharedDocs/FAQ/NCOV2019/FAQ_Liste.html}}</ref>). Uważa się, że rozprzestrzenia się on podobnie jak inne patogeny chorób układu oddechowego [[Droga szerzenia się zakażenia|drogą kropelkową]]<ref name=":0" /><ref name=":2" /><ref name=":3" /><ref>{{Cytuj |autor = |tytuł = Beschluss 1/2020 des ABAS vom 19.2.2020 und Begründung zur vorläufigen Einstufung des Virus SARS-CoV-2 in Risikogruppe 3 und Empfehlungen zu nicht gezielten Tätigkeiten (Labordiagnostik) und gezielten Tätigkeiten mit SARS-CoV-2. |data = |typ nośnika = pdf |url = https://www.baua.de/DE/Aufgaben/Geschaeftsfuehrung-von-Ausschuessen/ABAS/pdf/SARS-CoV-2.pdf}}</ref>. Do tej pory wirus został wykryty w wydzielinie z nosa i gardła, w plwocinie, moczu, stolcu, cieczy łzowej i krwi<ref>{{Cytuj |autor = Jasper Fuk-Woo Chan, Shuofeng Yuan, Kin-Hang Kok, Kelvin Kai-Wang To, Hin Chu |tytuł = A familial cluster of pneumonia associated with the 2019 novel coronavirus indicating person-to-person transmission: a study of a family cluster |czasopismo = The Lancet |data = 2020-02 |data dostępu = 2020-03-11 |wolumin = 395 |numer = 10223 |s = 514–523 |doi = 10.1016/S0140-6736(20)30154-9 |url = https://linkinghub.elsevier.com/retrieve/pii/S0140673620301549 |język = en}}</ref><ref>{{Cytuj |autor = Michelle L. Holshue, Chas DeBolt, Scott Lindquist, Kathy H. Lofy, John Wiesman |tytuł = First Case of 2019 Novel Coronavirus in the United States |czasopismo = New England Journal of Medicine |data = 2020-03-05 |data dostępu = 2020-03-11 |issn = 0028-4793 |wolumin = 382 |numer = 10 |s = 929–936 |doi = 10.1056/NEJMoa2001191 |url = http://www.nejm.org/doi/10.1056/NEJMoa2001191 |język = en}}</ref><ref>{{Cytuj |autor = Jianhua Xia, Jianping Tong, Mengyun Liu, Ye Shen, Dongyu Guo |tytuł = Evaluation of coronavirus in tears and conjunctival secretions of patients with SARS-CoV-2 infection |czasopismo = Journal of Medical Virology |data = 2020-02-26 |data dostępu = 2020-03-11 |issn = 0146-6615 |s = jmv.25725 |doi = 10.1002/jmv.25725 |url = https://onlinelibrary.wiley.com/doi/abs/10.1002/jmv.25725 |język = en}}</ref><ref>{{Cytuj |autor = Yu Chen, Lanjuan Li |tytuł = SARS-CoV-2: virus dynamics and host response |czasopismo = The Lancet Infectious Diseases |data = 2020-03 |data dostępu = 2020-03-26 |s = S1473309920302358 |doi = 10.1016/S1473-3099(20)30235-8 |url = https://linkinghub.elsevier.com/retrieve/pii/S1473309920302358 |język = en}}</ref>. |
||
Singapurscy naukowcy zalecają traktowanie kału jako zakaźnego w oparciu o wykrycie wirusa w kale oraz zakaźnego wirusa SARS-CoV w ściekach z dwóch chińskich szpitali podczas pandemii SARS 2002/2003. Aby wykluczyć możliwość infekcji [[Droga szerzenia się zakażenia|fekalno-oralnej]] oprócz infekcji [[Droga szerzenia się zakażenia|kropelkowej]] i [[Droga szerzenia się zakażenia|inhalacyjnej]], konieczne są dalsze badania zarówno dróg wydalania wirusa przez pacjenta, jak i potencjalnych zanieczyszczeń środowiskowych<ref>{{Cytuj |autor = Charleen Yeo, Sanghvi Kaushal, Danson Yeo |tytuł = Enteric involvement of coronaviruses: is faecal–oral transmission of SARS-CoV-2 possible? |czasopismo = The Lancet Gastroenterology & Hepatology |data = 2020-02 |data dostępu = 2020-03-11 |s = S2468125320300480 |doi = 10.1016/S2468-1253(20)30048-0 |url = https://linkinghub.elsevier.com/retrieve/pii/S2468125320300480 |język = en}}</ref>. W badaniu na niewielkiej grupie osób wykazano, że wysokie miano wirusa na tekstyliach i powierzchniach przedmiotów można wykryć w sypialniach i łazienkach osób zakażonych koronawirusem nawet metodą [[Reakcja łańcuchowa polimerazy|PCR]]. Ograniczeniem badania był brak hodowli wirusów w celu wykazania ich żywotności, zatem zakaźność nie została ostatecznie udowodniona, ale autorzy badania zakładają na podstawie swoich wstępnych wyników, że prawdopodobne jest przeniesienie wirusa przez zakażone przedmioty<ref>{{Cytuj |autor = Sean Wei Xiang Ong, Yian Kim Tan, Po Ying Chia, Tau Hong Lee, Oon Tek Ng |tytuł = Air, Surface Environmental, and Personal Protective Equipment Contamination by Severe Acute Respiratory Syndrome Coronavirus 2 (SARS-CoV-2) From a Symptomatic Patient |czasopismo = JAMA |data = 2020-03-04 |data dostępu = 2020-03-14 |issn = 0098-7484 |doi = 10.1001/jama.2020.3227 |url = https://jamanetwork.com/journals/jama/fullarticle/2762692 |język = en}}</ref>. |
Singapurscy naukowcy zalecają traktowanie kału jako zakaźnego w oparciu o wykrycie wirusa w kale oraz zakaźnego wirusa [[Wirus SARS|SARS-CoV]] w ściekach z dwóch chińskich szpitali podczas pandemii SARS 2002/2003. Aby wykluczyć możliwość infekcji [[Droga szerzenia się zakażenia|fekalno-oralnej]] oprócz infekcji [[Droga szerzenia się zakażenia|kropelkowej]] i [[Droga szerzenia się zakażenia|inhalacyjnej]], konieczne są dalsze badania zarówno dróg wydalania wirusa przez pacjenta, jak i potencjalnych zanieczyszczeń środowiskowych<ref>{{Cytuj |autor = Charleen Yeo, Sanghvi Kaushal, Danson Yeo |tytuł = Enteric involvement of coronaviruses: is faecal–oral transmission of SARS-CoV-2 possible? |czasopismo = The Lancet Gastroenterology & Hepatology |data = 2020-02 |data dostępu = 2020-03-11 |s = S2468125320300480 |doi = 10.1016/S2468-1253(20)30048-0 |url = https://linkinghub.elsevier.com/retrieve/pii/S2468125320300480 |język = en}}</ref>. W badaniu na niewielkiej grupie osób wykazano, że wysokie miano wirusa na tekstyliach i powierzchniach przedmiotów można wykryć w sypialniach i łazienkach osób zakażonych koronawirusem nawet metodą [[Reakcja łańcuchowa polimerazy|PCR]]. Ograniczeniem badania był brak hodowli wirusów w celu wykazania ich żywotności, zatem zakaźność nie została ostatecznie udowodniona, ale autorzy badania zakładają na podstawie swoich wstępnych wyników, że prawdopodobne jest przeniesienie wirusa przez zakażone przedmioty<ref>{{Cytuj |autor = Sean Wei Xiang Ong, Yian Kim Tan, Po Ying Chia, Tau Hong Lee, Oon Tek Ng |tytuł = Air, Surface Environmental, and Personal Protective Equipment Contamination by Severe Acute Respiratory Syndrome Coronavirus 2 (SARS-CoV-2) From a Symptomatic Patient |czasopismo = JAMA |data = 2020-03-04 |data dostępu = 2020-03-14 |issn = 0098-7484 |doi = 10.1001/jama.2020.3227 |url = https://jamanetwork.com/journals/jama/fullarticle/2762692 |język = en}}</ref>. Również nowsze badania SARS-CoV-2 wykazują obecność wirusa w kale, co sugeruje możliwość jego replikacji w przewodzie pokarmowym<ref>{{Cytuj |autor = Yongjian Wu, Cheng Guo, Lantian Tang, Zhongsi Hong, Jianhui Zhou |tytuł = Prolonged presence of SARS-CoV-2 viral RNA in faecal samples |czasopismo = The Lancet Gastroenterology & Hepatology |data = 2020-05 |data dostępu = 2020-04-09 |issn = 2468-1253 |wolumin = 5 |numer = 5 |s = 434–435 |doi = 10.1016/s2468-1253(20)30083-2 |url = https://doi.org/10.1016/S2468-1253(20)30083-2}}</ref>. |
||
Na podstawie analiz ilościowych badań [[Reakcja łańcuchowa polimerazy z odwrotną transkrypcją|RT-PCR]] nosowej części gardła (''nasopharynx'') chińscy naukowcy wywnioskowali, że wirus, podobnie jak grypa, może być przenoszony również drogą wziewną (powietrzną)<ref name=":4">{{Cytuj |autor = Lirong Zou, Feng Ruan, Mingxing Huang, Lijun Liang, Huitao Huang |tytuł = SARS-CoV-2 Viral Load in Upper Respiratory Specimens of Infected Patients |czasopismo = New England Journal of Medicine |data = 2020-02-19 |data dostępu = 2020-03-11 |issn = 0028-4793 |s = NEJMc2001737 |doi = 10.1056/NEJMc2001737 |url = http://www.nejm.org/doi/10.1056/NEJMc2001737 |język = en}}</ref><ref>{{Cytuj |autor = |tytuł = Neuartiges Virus SARS-CoV-2 (bislang 2019-nCoV) durch den ABAS in Risikogruppe 3 eingestuft und Empfehlungen zur Labordiagnostik |data = |opis = BAuA: Federal Institute for Occupational Safety and Health |url = https://www.baua.de/DE/Angebote/Aktuelles/Meldungen/2020/2020-02-19-Coronavirus.htm}}</ref>. U 17 badanych pacjentów z objawami choroby miano wirusa w nosie było wyższe niż w gardle, a występowanie szczególnie w górnych drogach oddechowych odróżnia SARS-CoV-2 od wirusa wywołującego [[SARS]]<ref name=":4" />. Testy pacjentów z obszaru [[Monachium]] nie wykazały wirulencji z próbek kału pomimo wykrycia RNA wirusa. Miano wirusa w nosogardzieli było jednak 1000 razy wyższe niż w znanych wcześniej chorobach wywołanych koronawirusami, takimi jak SARS i [[Bliskowschodni zespół oddechowy|MERS]]<ref>{{Cytuj |autor = Roman Woelfel, Victor Max Corman, Wolfgang Guggemos, Michael Seilmaier, Sabine Zange |tytuł = Clinical presentation and virological assessment of hospitalized cases of coronavirus disease 2019 in a travel-associated transmission cluster |data = 2020-03-08 |data dostępu = 2020-03-11 |doi = 10.1101/2020.03.05.20030502 |url = http://medrxiv.org/lookup/doi/10.1101/2020.03.05.20030502 |język = en}}</ref>. Dowodzi to, że wirus replikuje się już w górnych drogach oddechowych, chociaż w głębszych obszarach płuc może wchodzić w fuzję z komórkami gospodarza i namnażać się poprzez wiązanie z receptorem błonowym ACE-2 ([[Konwertaza angiotensyny|konwertaza angiotensyny 2]]; kininaza II) dzięki domenie wiążącej receptor (ang. ''Receptor Binding Domain'') zakodowanej w białku fuzyjnym S (ang. ''Spike'') wirusa SARS-CoV-2. |
Na podstawie analiz ilościowych badań [[Reakcja łańcuchowa polimerazy z odwrotną transkrypcją|RT-PCR]] nosowej części gardła (''nasopharynx'') chińscy naukowcy wywnioskowali, że wirus, podobnie jak grypa, może być przenoszony również drogą wziewną (powietrzną)<ref name=":4">{{Cytuj |autor = Lirong Zou, Feng Ruan, Mingxing Huang, Lijun Liang, Huitao Huang |tytuł = SARS-CoV-2 Viral Load in Upper Respiratory Specimens of Infected Patients |czasopismo = New England Journal of Medicine |data = 2020-02-19 |data dostępu = 2020-03-11 |issn = 0028-4793 |s = NEJMc2001737 |doi = 10.1056/NEJMc2001737 |url = http://www.nejm.org/doi/10.1056/NEJMc2001737 |język = en}}</ref><ref>{{Cytuj |autor = |tytuł = Neuartiges Virus SARS-CoV-2 (bislang 2019-nCoV) durch den ABAS in Risikogruppe 3 eingestuft und Empfehlungen zur Labordiagnostik |data = |opis = BAuA: Federal Institute for Occupational Safety and Health |url = https://www.baua.de/DE/Angebote/Aktuelles/Meldungen/2020/2020-02-19-Coronavirus.htm}}</ref>. U 17 badanych pacjentów z objawami choroby miano wirusa w nosie było wyższe niż w gardle, a występowanie szczególnie w górnych drogach oddechowych odróżnia SARS-CoV-2 od wirusa wywołującego [[SARS]]<ref name=":4" />. Testy pacjentów z obszaru [[Monachium]] nie wykazały wirulencji z próbek kału pomimo wykrycia RNA wirusa. Miano wirusa w nosogardzieli było jednak 1000 razy wyższe niż w znanych wcześniej chorobach wywołanych koronawirusami, takimi jak SARS i [[Bliskowschodni zespół oddechowy|MERS]]<ref>{{Cytuj |autor = Roman Woelfel, Victor Max Corman, Wolfgang Guggemos, Michael Seilmaier, Sabine Zange |tytuł = Clinical presentation and virological assessment of hospitalized cases of coronavirus disease 2019 in a travel-associated transmission cluster |data = 2020-03-08 |data dostępu = 2020-03-11 |doi = 10.1101/2020.03.05.20030502 |url = http://medrxiv.org/lookup/doi/10.1101/2020.03.05.20030502 |język = en}}</ref>. Dowodzi to, że wirus replikuje się już w górnych drogach oddechowych, chociaż dopiero w głębszych obszarach płuc może wchodzić w fuzję z komórkami gospodarza i namnażać się poprzez wiązanie z receptorem błonowym ACE-2 ([[Konwertaza angiotensyny|konwertaza angiotensyny 2]]; kininaza II) dzięki domenie wiążącej receptor (ang. ''Receptor Binding Domain'') zakodowanej w białku fuzyjnym S (ang. ''Spike'') wirusa SARS-CoV-2. Rozszerzenie [[Tropizm tkankowy|tropizmu tkankowego]] do obszarów gardła prowadzi do założenia, że wirus zawiera proteazę furynopodobną, która jest niezbędna na etapie cięcia enzymatycznego (ang. ''Cleaving'') w celu wejścia do komórki gospodarza i następnie replikacji w niej<ref name=":5">{{Cytuj |autor = B. Coutard, C. Valle, X. de Lamballerie, B. Canard, N.G. Seidah |tytuł = The spike glycoprotein of the new coronavirus 2019-nCoV contains a furin-like cleavage site absent in CoV of the same clade |czasopismo = Antiviral Research |data = 2020-04 |data dostępu = 2020-03-11 |wolumin = 176 |s = 104742 |doi = 10.1016/j.antiviral.2020.104742 |url = https://linkinghub.elsevier.com/retrieve/pii/S0166354220300528 |język = en}}</ref><ref>{{Cytuj |autor = Z. Galen Wo |tytuł = Sequence Analysis Indicates that 2019-nCoV Virus Contains a Putative Furin Cleavage Site at the Boundary of S1 and S2 Domains of Spike Protein |data = 2020-02-18 |data dostępu = 2020-03-11 |doi = 10.31219/osf.io/nkcrf |url = https://osf.io/nkcrf}}</ref><ref>{{Cytuj |autor = Roman Wölfel, Victor M. Corman, Wolfgang Guggemos, Michael Seilmaier, Sabine Zange |tytuł = Virological assessment of hospitalized patients with COVID-2019 |czasopismo = Nature |data = 2020-04-01 |data dostępu = 2020-04-09 |issn = 1476-4687 |s = 1–10 |doi = 10.1038/s41586-020-2196-x |url = https://www.nature.com/articles/s41586-020-2196-x |język = en}}</ref>. |
||
Nie jest jasne, czy transmisja wirusa odbywa się poprzez kontakt z zanieczyszczonymi powierzchniami i przedmiotami<ref name=":2" />. Analiza 22 badań dotyczących przeżywalności koronawirusów istotnych z medycznego punktu widzenia (takich jak [[SARS|SARS-CoV]] i [[Bliskowschodni zespół oddechowy|MERS-CoV]]) na powierzchniach pokazuje, że wirusy te mogą utrzymywać się na powierzchniach materiałów wykonanych z metalu, szkła lub tworzywa sztucznego do dziewięciu dni w temperaturze pokojowej. Pozostają jednak zakaźne przez średnio cztery do pięciu dni. Jednocześnie mogą one zostać inaktywowane odpowiednimi środkami dezynfekującymi. Według naukowców zaangażowanych we wspomniane badania odkrycia te można zastosować do SARS-CoV-2<ref>{{Cytuj |autor = G. Kampf, D. Todt, S. Pfaender, E. Steinmann |tytuł = Persistence of coronaviruses on inanimate surfaces and their inactivation with biocidal agents |czasopismo = Journal of Hospital Infection |data = 2020-03 |data dostępu = 2020-03-11 |wolumin = 104 |numer = 3 |s = 246–251 |doi = 10.1016/j.jhin.2020.01.022 |url = https://linkinghub.elsevier.com/retrieve/pii/S0195670120300463 |język = en}}</ref><ref>{{Cytuj |tytuł = Wie lang Coronaviren auf Flächen überleben und wie man sie inaktiviert |data dostępu = 2020-03-11 |opublikowany = idw-online.de |url = https://idw-online.de/de/news731164 |język = de}}</ref>. |
Nie jest jasne, czy transmisja wirusa odbywa się poprzez kontakt z zanieczyszczonymi powierzchniami i przedmiotami<ref name=":2" />. Analiza 22 badań dotyczących przeżywalności koronawirusów istotnych z medycznego punktu widzenia (takich jak [[SARS|SARS-CoV]] i [[Bliskowschodni zespół oddechowy|MERS-CoV]]) na powierzchniach pokazuje, że wirusy te mogą utrzymywać się na powierzchniach materiałów wykonanych z metalu, szkła lub tworzywa sztucznego do dziewięciu dni w temperaturze pokojowej. Pozostają jednak zakaźne przez średnio cztery do pięciu dni. Jednocześnie mogą one zostać inaktywowane odpowiednimi środkami dezynfekującymi. Według naukowców zaangażowanych we wspomniane badania odkrycia te można zastosować do SARS-CoV-2<ref>{{Cytuj |autor = G. Kampf, D. Todt, S. Pfaender, E. Steinmann |tytuł = Persistence of coronaviruses on inanimate surfaces and their inactivation with biocidal agents |czasopismo = Journal of Hospital Infection |data = 2020-03 |data dostępu = 2020-03-11 |wolumin = 104 |numer = 3 |s = 246–251 |doi = 10.1016/j.jhin.2020.01.022 |url = https://linkinghub.elsevier.com/retrieve/pii/S0195670120300463 |język = en}}</ref><ref>{{Cytuj |tytuł = Wie lang Coronaviren auf Flächen überleben und wie man sie inaktiviert |data dostępu = 2020-03-11 |opublikowany = idw-online.de |url = https://idw-online.de/de/news731164 |język = de}}</ref>. |
||
Temperatura wpływa na aktywność i przeżywalność wirusa SARS-CoV-2, który jest wysoce stabilny w temperaturze 4 °C i może w niej przetrwać znacznie dłużej niż 14 dni. W temperaturze pokojowej (22 °C) i wyższej przeżywalność wirusa nie przekracza 14 dni, a w temperaturze 70 °C czas inaktywacji wirusa jest zredukowany do 5 minut. W temperaturze pokojowej wirus SARS-CoV-2 jest wyjątkowo stabilny w szerokim zakresie pH, wynoszącym od 3 do 10<ref>{{Cytuj |autor = Alex Chin, Julie Chu, Mahen Perera, Kenrie Hui, Hui-Ling Yen |tytuł = Stability of SARS-CoV-2 in different environmental conditions |czasopismo = medRxiv |data = 2020-03-27 |data dostępu = 2020-03-28 |s = 2020.03.15.20036673 |doi = 10.1101/2020.03.15.20036673 |url = https://www.medrxiv.org/content/10.1101/2020.03.15.20036673v2 |język = en}}</ref>. |
Temperatura wpływa na aktywność i przeżywalność wirusa SARS-CoV-2, który jest wysoce stabilny w temperaturze 4 °C i może w niej przetrwać znacznie dłużej niż 14 dni. W temperaturze pokojowej (22 °C) i wyższej przeżywalność wirusa nie przekracza 14 dni, a w temperaturze 70 °C czas inaktywacji wirusa jest zredukowany do 5 minut. W temperaturze pokojowej wirus SARS-CoV-2 jest wyjątkowo stabilny w szerokim zakresie pH, wynoszącym od 3 do 10<ref>{{Cytuj |autor = Alex Chin, Julie Chu, Mahen Perera, Kenrie Hui, Hui-Ling Yen |tytuł = Stability of SARS-CoV-2 in different environmental conditions |czasopismo = medRxiv |data = 2020-03-27 |data dostępu = 2020-03-28 |s = 2020.03.15.20036673 |doi = 10.1101/2020.03.15.20036673 |url = https://www.medrxiv.org/content/10.1101/2020.03.15.20036673v2 |język = en}}</ref>. |
||
| ⚫ | Mydło inaktywuje wirusa poprzez niszczenie jego otoczki lipidowej<ref>{{Cytuj |autor = Ed Yong |tytuł = Why the Coronavirus Has Been So Successful |data = 2020-03-20 |data dostępu = 2020-03-21 |opublikowany = The Atlantic |url = https://www.theatlantic.com/science/archive/2020/03/biography-new-coronavirus/608338/ |język = en}}</ref><ref>{{Cytuj |tytuł = WHO potwierdza: w wodzie koronawirusa nie ma |data dostępu = 2020-03-21 |opublikowany = Inżynieria.com |url = https://inzynieria.com/toptematcovid19/wiadomosci/58016,who-potwierdza-w-wodzie-koronawirusa-nie-ma}}</ref>. Badania potwierdziły również skuteczność środków dezynfekujących na bazie alkoholu w walce z SARS-CoV-2<ref>{{Cytuj |autor = Annika Kratzel, Daniel Todt, Philip V’kovski, Silvio Steiner, Mitra L. Gultom |tytuł = Efficient inactivation of SARS-CoV-2 by WHO-recommended hand rub formulations and alcohols |czasopismo = bioRxiv |data = 2020-03-17 |data dostępu = 2020-03-21 |s = 2020.03.10.986711 |doi = 10.1101/2020.03.10.986711 |url = https://www.biorxiv.org/content/10.1101/2020.03.10.986711v1 |język = en}}</ref>. |
||
W badaniu z udziałem dziewięciu pacjentek, które były zakażone SARS-CoV2 w ostatnim trymestrze ciąży, każde z dziewięciorga dzieci okazało się wolne od infekcji wirusem po urodzeniu przez cesarskie cięcie. Na tej podstawie autorzy badania stwierdzili, że wirus nie przenosi się w łonie matki<ref>{{Cytuj |autor = Huijun Chen, Juanjuan Guo, Chen Wang, Fan Luo, Xuechen Yu |tytuł = Clinical characteristics and intrauterine vertical transmission potential of COVID-19 infection in nine pregnant women: a retrospective review of medical records |czasopismo = The Lancet |data = 2020-03 |data dostępu = 2020-03-11 |wolumin = 395 |numer = 10226 |s = 809–815 |doi = 10.1016/S0140-6736(20)30360-3 |url = https://linkinghub.elsevier.com/retrieve/pii/S0140673620303603 |język = en}}</ref>. Do 6 lutego 2020 roku chińskie instytucje zdrowia publicznego zarejestrowały tylko dziewięcioro niemowląt, u których wykazano dowody na obecność wirusa. Autorzy badania za możliwą przyczynę tej niewielkiej liczby wskazują stosunkowo dużą liczbę przypadków skąpoobjawowych wśród dzieci, a także niedostateczny system raportowania przypadków<ref>{{Cytuj |autor = Min Wei, Jingping Yuan, Yu Liu, Tao Fu, Xue Yu |tytuł = Novel Coronavirus Infection in Hospitalized Infants Under 1 Year of Age in China |czasopismo = JAMA |data = 2020-02-14 |data dostępu = 2020-03-11 |issn = 0098-7484 |doi = 10.1001/jama.2020.2131 |pmid = 32058570 |pmc = PMC7042807 |url = https://jamanetwork.com/journals/jama/fullarticle/2761659 |język = en}}</ref>. |
W badaniu z udziałem dziewięciu pacjentek, które były zakażone SARS-CoV2 w ostatnim trymestrze ciąży, każde z dziewięciorga dzieci okazało się wolne od infekcji wirusem po urodzeniu przez cesarskie cięcie. Na tej podstawie autorzy badania stwierdzili, że wirus nie przenosi się w łonie matki<ref>{{Cytuj |autor = Huijun Chen, Juanjuan Guo, Chen Wang, Fan Luo, Xuechen Yu |tytuł = Clinical characteristics and intrauterine vertical transmission potential of COVID-19 infection in nine pregnant women: a retrospective review of medical records |czasopismo = The Lancet |data = 2020-03 |data dostępu = 2020-03-11 |wolumin = 395 |numer = 10226 |s = 809–815 |doi = 10.1016/S0140-6736(20)30360-3 |url = https://linkinghub.elsevier.com/retrieve/pii/S0140673620303603 |język = en}}</ref>. Do 6 lutego 2020 roku chińskie instytucje zdrowia publicznego zarejestrowały tylko dziewięcioro niemowląt, u których wykazano dowody na obecność wirusa. Autorzy badania za możliwą przyczynę tej niewielkiej liczby wskazują stosunkowo dużą liczbę przypadków skąpoobjawowych wśród dzieci, a także niedostateczny system raportowania przypadków<ref>{{Cytuj |autor = Min Wei, Jingping Yuan, Yu Liu, Tao Fu, Xue Yu |tytuł = Novel Coronavirus Infection in Hospitalized Infants Under 1 Year of Age in China |czasopismo = JAMA |data = 2020-02-14 |data dostępu = 2020-03-11 |issn = 0098-7484 |doi = 10.1001/jama.2020.2131 |pmid = 32058570 |pmc = PMC7042807 |url = https://jamanetwork.com/journals/jama/fullarticle/2761659 |język = en}}</ref>. |
||
Ponieważ SARS-CoV-2 jest uznawany za wirus odzwierzęcy, przypuszcza się, że możliwa jest transmisja pomiędzy niektórymi zwierzętami i ludźmi<ref name=":0" /><ref name=":1" /><ref name=":43"/>. Jednocześnie [[Światowa Organizacja Zdrowia]] stwierdziła, że nie ma wystarczających dowodów na to, że zwierzęta domowe, takie jak koty czy psy, mogą zarazić się koronawirusem<ref>{{Cytuj |tytuł = Myth busters |data dostępu = 2020-03-11 |opublikowany = www.who.int |url = https://www.who.int/emergencies/diseases/novel-coronavirus-2019/advice-for-public/myth-busters |język = en}}</ref>. Dyskusyjny jest pojedynczy przypadek z 28 lutego 2020 roku „lekko pozytywnego” wyniku testu na [[COVID-19]] u psa w [[Hongkong]]u, przy czym w oświadczeniu wydanym przez Ministerstwo Rolnictwa, Rybołówstwa i Rezerwatów Przyrody Hongkongu (ang. ''Hong Kong Agriculture, Fisheries and Conservation Department'') oświadczono o braku dowodów na możliwość infekcji COVID-19 u zwierząt domowych czy możliwości przeniesienia infekcji ze zwierzęcia domowego na człowieka<ref>{{Cytuj |tytuł = Detection of low level of COVID-19 virus in pet dog |data dostępu = 2020-03-11 |opublikowany = www.info.gov.hk |url = https://www.info.gov.hk/gia/general/202002/28/P2020022800013.htm}}</ref>. |
Ponieważ SARS-CoV-2 jest uznawany za wirus odzwierzęcy, przypuszcza się, że możliwa jest transmisja pomiędzy niektórymi zwierzętami i ludźmi<ref name=":0" /><ref name=":1" /><ref name=":43"/>. Jednocześnie [[Światowa Organizacja Zdrowia]] stwierdziła, że nie ma wystarczających dowodów na to, że zwierzęta domowe, takie jak koty czy psy, mogą zarazić się koronawirusem<ref>{{Cytuj |tytuł = Myth busters |data dostępu = 2020-03-11 |opublikowany = www.who.int |url = https://www.who.int/emergencies/diseases/novel-coronavirus-2019/advice-for-public/myth-busters |język = en}}</ref>. Dyskusyjny jest pojedynczy przypadek z 28 lutego 2020 roku „lekko pozytywnego” wyniku testu na [[COVID-19]] u psa w [[Hongkong]]u, przy czym w oświadczeniu wydanym przez Ministerstwo Rolnictwa, Rybołówstwa i Rezerwatów Przyrody Hongkongu (ang. ''Hong Kong Agriculture, Fisheries and Conservation Department'') oświadczono o braku dowodów na możliwość infekcji COVID-19 u zwierząt domowych czy możliwości przeniesienia infekcji ze zwierzęcia domowego na człowieka<ref>{{Cytuj |tytuł = Detection of low level of COVID-19 virus in pet dog |data dostępu = 2020-03-11 |opublikowany = www.info.gov.hk |url = https://www.info.gov.hk/gia/general/202002/28/P2020022800013.htm}}</ref>. |
||
| ⚫ | Mydło inaktywuje wirusa poprzez niszczenie jego otoczki lipidowej<ref>{{Cytuj |autor = Ed Yong |tytuł = Why the Coronavirus Has Been So Successful |data = 2020-03-20 |data dostępu = 2020-03-21 |opublikowany = The Atlantic |url = https://www.theatlantic.com/science/archive/2020/03/biography-new-coronavirus/608338/ |język = en}}</ref><ref>{{Cytuj |tytuł = WHO potwierdza: w wodzie koronawirusa nie ma |data dostępu = 2020-03-21 |opublikowany = Inżynieria.com |url = https://inzynieria.com/toptematcovid19/wiadomosci/58016,who-potwierdza-w-wodzie-koronawirusa-nie-ma}}</ref>. Badania potwierdziły również skuteczność środków dezynfekujących na bazie alkoholu w walce z SARS-CoV-2<ref>{{Cytuj |autor = Annika Kratzel, Daniel Todt, Philip V’kovski, Silvio Steiner, Mitra L. Gultom |tytuł = Efficient inactivation of SARS-CoV-2 by WHO-recommended hand rub formulations and alcohols |czasopismo = bioRxiv |data = 2020-03-17 |data dostępu = 2020-03-21 |s = 2020.03.10.986711 |doi = 10.1101/2020.03.10.986711 |url = https://www.biorxiv.org/content/10.1101/2020.03.10.986711v1 |język = en}}</ref>. |
||
Ze względu na rozmiar (od 60 do 140 nm) wirus jest przepuszczany przez maski z filtrami FFP1, P1,FFP2, P2, N95, N99, FFP3, P3, N100 (filtracja wszystkich cząstek o średnicy 0,3 mikrona lub większych)<ref>{{Cytuj |tytuł = COVID-19 a praca lekarza dentysty |data dostępu = 2020-03-29 |opublikowany = www.termedia.pl |url = https://www.termedia.pl/zalecenia/COVID-19-a-praca-lekarza-dentysty,37275.html}}</ref>. |
Ze względu na rozmiar (od 60 do 140 nm) wirus jest przepuszczany przez maski z filtrami FFP1, P1,FFP2, P2, N95, N99, FFP3, P3, N100 (filtracja wszystkich cząstek o średnicy 0,3 mikrona lub większych)<ref>{{Cytuj |tytuł = COVID-19 a praca lekarza dentysty |data dostępu = 2020-03-29 |opublikowany = www.termedia.pl |url = https://www.termedia.pl/zalecenia/COVID-19-a-praca-lekarza-dentysty,37275.html}}</ref>. |
||
| Linia 319: | Linia 319: | ||
|} |
|} |
||
Nie istnieje wiele badań koncentrujących się na przyczynach różnic w zachorowalności i śmiertelności wirusa u różnych płci i w różnych grupach wiekowych, które mogłyby wskazać, czy istotną rolę odgrywają cechy immunologiczne poszczególnych grup, czy cechy [[fenotyp]]owe samego wirusa, a tym samym wskazać dalsze możliwości leczenia. Prace badawcze wykryły podwyższone [[Miano mikroorganizmów|miano]] wirusa u osób starszych, co może korelować z ciężkim przebiegiem choroby w tych grupach wiekowych. Część naukowców spekuluje, że może to być związane z podwyższoną ekspresją receptora ACE2 u starszych pacjentów<ref>{{Cytuj |autor = Yu Chen, Lanjuan Li |tytuł = SARS-CoV-2: virus dynamics and host response |czasopismo = The Lancet Infectious Diseases |data = 2020-03 |data dostępu = 2020-03-28 |s = S1473309920302358 |doi = 10.1016/S1473-3099(20)30235-8 |url = https://linkinghub.elsevier.com/retrieve/pii/S1473309920302358 |język = en}}</ref>. |
Nie istnieje wiele badań koncentrujących się na przyczynach różnic w zachorowalności i śmiertelności wirusa u różnych płci i w różnych grupach wiekowych, które mogłyby wskazać, czy istotną rolę odgrywają cechy immunologiczne poszczególnych grup, czy cechy [[fenotyp]]owe samego wirusa, a tym samym wskazać dalsze możliwości leczenia. Prace badawcze wykryły podwyższone [[Miano mikroorganizmów|miano]] wirusa u osób starszych, co może korelować z ciężkim przebiegiem choroby w tych grupach wiekowych. Część naukowców spekuluje, że może to być związane z podwyższoną ekspresją receptora ACE2 u starszych pacjentów<ref>{{Cytuj |autor = Yu Chen, Lanjuan Li |tytuł = SARS-CoV-2: virus dynamics and host response |czasopismo = The Lancet Infectious Diseases |data = 2020-03 |data dostępu = 2020-03-28 |s = S1473309920302358 |doi = 10.1016/S1473-3099(20)30235-8 |url = https://linkinghub.elsevier.com/retrieve/pii/S1473309920302358 |język = en}}</ref>. |
||
| ⚫ | Analiza kliniczna pacjentów pokazuje, że grupa krwi może wpływać na ryzyko infekcji [[COVID-19]]. Podobnie jak wykazywały wcześniejsze badania [[SARS]] w czasie epidemii w 2019 roku<ref>{{Cytuj |autor = Yufeng Cheng, Gregory Cheng, C. H. Chui, F. Y. Lau, Paul K. S. Chan |tytuł = ABO Blood Group and Susceptibility to Severe Acute Respiratory Syndrome |czasopismo = JAMA |data = 2005-03-23 |data dostępu = 2020-03-21 |issn = 0098-7484 |wolumin = 293 |numer = 12 |s = 1447–1451 |doi = 10.1001/jama.293.12.1450-c |url = https://jamanetwork.com/journals/jama/fullarticle/200582 |język = en}}</ref>, na podstawie wyników badań 2173 pacjentów chorych na COVID-19 z trzech szpitali w [[Wuhan]] i [[Shenzhen]] uważa się, że [[Grupy krwi|grupa krwi]] A zwiększa ryzyko choroby zakaźnej COVID-19, podczas gdy grupa krwi 0 je zmniejsza (lecz nie eliminuje całkowicie)<ref>{{Cytuj |autor = Jiao Zhao, Yan Yang, Han-Ping Huang, Dong Li, Dong-Feng Gu |tytuł = Relationship between the ABO Blood Group and the COVID-19 Susceptibility |czasopismo = medRxiv |data = 2020-03-16 |data dostępu = 2020-03-21 |s = 2020.03.11.20031096 |doi = 10.1101/2020.03.11.20031096 |url = https://www.medrxiv.org/content/10.1101/2020.03.11.20031096v1 |język = en}}</ref>. |
||
=== Zjadliwość i wskaźniki śmiertelności === |
=== Zjadliwość i wskaźniki śmiertelności === |
||
| Linia 360: | Linia 362: | ||
Słabo poznany jest sam proces egzocytozy wirusa. |
Słabo poznany jest sam proces egzocytozy wirusa. |
||
| ⚫ | |||
==== Immunopatologia ==== |
|||
[[Odpowiedź odpornościowa swoista|Swoista odpowiedź immunologiczna]] odgrywa istotną rolę w infekcji SARS-CoV-2<ref>{{Cytuj |autor = Luca Cristiani, Enrica Mancino, Luigi Matera, Raffaella Nenna, Alessandra Pierangeli |tytuł = Will children reveal their secret? The coronavirus dilemma |czasopismo = The European Respiratory Journal |data = 2020-04-02 |data dostępu = 2020-04-09 |issn = 0903-1936 |doi = 10.1183/13993003.00749-2020 |pmid = 32241833 |pmc = 7113798 |url = https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7113798/}}</ref>. Wirus może z jednej strony unikać nadzoru immunologicznego w organizmie<ref>{{Cytuj |autor = Elio Issa, Georgi Merhi, Balig Panossian, Tamara Salloum, Sima Tokajian |tytuł = SARS-CoV-2 and ORF3a: Non-Synonymous Mutations and Polyproline Regions |czasopismo = bioRxiv |data = 2020-03-28 |data dostępu = 2020-04-09 |s = 2020.03.27.012013 |doi = 10.1101/2020.03.27.012013 |url = https://www.biorxiv.org/content/10.1101/2020.03.27.012013v1 |język = en}}</ref>, z drugiej wywołać w ciężkich przypadkach [[Burza cytokin|burzę cytokin]]<ref>{{Cytuj |autor = Savannah F. Pedersen, Ya-Chi Ho |tytuł = SARS-CoV-2: A Storm is Raging |czasopismo = The Journal of Clinical Investigation |data = 2020-03-27 |data dostępu = 2020-04-09 |issn = 1558-8238 |doi = 10.1172/JCI137647 |pmid = 32217834 |url = https://www.ncbi.nlm.nih.gov/pubmed/32217834}}</ref>. |
|||
| ⚫ | |||
SARS-CoV-2 wytwarza co najmniej trzy czynniki wirulencji, które promują rozprzestrzenianie się nowych wirionów z komórek gospodarza i hamują odpowiedź immunologiczną: Nsp1, Nsp3c oraz ORF7a<ref>{{Cytuj |autor = Canrong Wu, Yang Liu, Yueying Yang, Peng Zhang, Wu Zhong |tytuł = Analysis of therapeutic targets for SARS-CoV-2 and discovery of potential drugs by computational methods |czasopismo = Acta Pharmaceutica Sinica B |data = 2020-02 |data dostępu = 2020-03-12 |s = S2211383520302999 |doi = 10.1016/j.apsb.2020.02.008 |url = https://linkinghub.elsevier.com/retrieve/pii/S2211383520302999 |język = en}}</ref>. |
SARS-CoV-2 wytwarza co najmniej trzy czynniki wirulencji, które promują rozprzestrzenianie się nowych wirionów z komórek gospodarza i hamują odpowiedź immunologiczną: Nsp1, Nsp3c oraz ORF7a<ref>{{Cytuj |autor = Canrong Wu, Yang Liu, Yueying Yang, Peng Zhang, Wu Zhong |tytuł = Analysis of therapeutic targets for SARS-CoV-2 and discovery of potential drugs by computational methods |czasopismo = Acta Pharmaceutica Sinica B |data = 2020-02 |data dostępu = 2020-03-12 |s = S2211383520302999 |doi = 10.1016/j.apsb.2020.02.008 |url = https://linkinghub.elsevier.com/retrieve/pii/S2211383520302999 |język = en}}</ref>. |
||
===== Indukcja burzy cytokin ===== |
===== Indukcja burzy cytokin ===== |
||
Spośród białek strukturalnych glikoproteina S (zwłaszcza domena A białka S1 oraz domena wiążąca receptor RBD) i N są najważniejszymi [[Immunogen|immunogenami]]<ref>{{Cytuj |autor = Nisreen M. A. Okba, Marcel A. Muller, Wentao Li, Chunyan Wang, Corine H. GeurtsvanKessel |tytuł = SARS-CoV-2 specific antibody responses in COVID-19 patients |czasopismo = medRxiv |data = 2020-03-20 |data dostępu = 2020-04-09 |s = 2020.03.18.20038059 |doi = 10.1101/2020.03.18.20038059 |url = https://www.medrxiv.org/content/10.1101/2020.03.18.20038059v1 |język = en}}</ref><ref>{{Cytuj |autor = Chen Dong, Ling Ni, Fang Ye, Meng-Li Chen, Yu Feng |tytuł = Characterization of anti-viral immunity in recovered individuals infected by SARS-CoV-2 |czasopismo = medRxiv |data = 2020-03-20 |data dostępu = 2020-04-09 |s = 2020.03.17.20036640 |doi = 10.1101/2020.03.17.20036640 |url = https://www.medrxiv.org/content/10.1101/2020.03.17.20036640v1 |język = en}}</ref>. |
|||
| ⚫ | Główną przyczyną śmierci u pacjentów w przebiegu [[COVID-19]] jest [[Zespół ostrej niewydolności oddechowej|zespół ostrej niewydolności oddechowej (ARDS)]]. Spośród 41 pacjentów zakażonym wirusem SARS-CoV-2 we wczesnej fazie pandemii w Wuhan 6 zmarło właśnie na skutek [[Zespół ostrej niewydolności oddechowej|ARDS]]. Jednym z głównych mechanizmów występujących w ARDS jest niekontrolowana systemowa odpowiedź zapalna w postaci burzy [[Cytokiny|cytokin]] (IFN-α, IFN-γ, IL-1β, IL-6, IL-12, IL-18, IL-33, TNF-α, TGFβ itp.) oraz [[Chemokiny|chemokin]] (CCL2, CCL3, CCL5, CXCL8, CXCL9, CXCL10), co prowadzi w konsekwencji do niewydolności oddechowej oraz niewydolności wielu organów i śmierci<ref name=":6" />. |
||
| ⚫ | Grupa naukowców z [[Melbourne]] pod kierownictwem Katherine Kedzierskiej dokonała studium przypadku pacjentki z łagodnym przebiegiem [[COVID-19]] i przeanalizowała przebieg jej [[Odpowiedź immunologiczna|odpowiedzi immunologicznej]], wskazując na podobieństwa do reakcji immunologicznej organizmu na [[Wirus grypy|wirusa grypy]], będącej przedmiotem wcześniejszych badań tego zespołu. Badacze wykazali podwyższenie poziomu komórek wydzielających swoiste przeciwciała ASC, [[Limfocyty T|limfocytów pomocniczych grudkowych T<sub>FH</sub>]], [[Antygen różnicowania komórkowego|aktywowanych limfocytów CD4<sup>+</sup> i CD8<sup>+</sup>]] wraz z przeciwciałami [[Immunoglobuliny M|IgM]] i [[Immunoglobuliny G|IgG]] wiążącymi [[Koronawirusy|koronawirusa]] SARS-CoV-2. Schemat odpowiedzi immunologicznej może być przydatny przy prognozowaniu przebiegu choroby, opracowaniu szczepionki lub leków łagodzących przebieg choroby. Autorzy postulują jednak kontynuację badań na większej grupie pacjentów o różnych stopniach zaostrzenia choroby COVID-19<ref>{{Cytuj |autor = Irani Thevarajan, Thi H. O. Nguyen, Marios Koutsakos, Julian Druce, Leon Caly |tytuł = Breadth of concomitant immune responses prior to patient recovery: a case report of non-severe COVID-19 |czasopismo = Nature Medicine |data = 2020-03-16 |data dostępu = 2020-03-21 |issn = 1546-170X |s = 1–3 |doi = 10.1038/s41591-020-0819-2 |url = https://www.nature.com/articles/s41591-020-0819-2 |język = en}}</ref>. |
||
== Odpowiedź immunologiczna == |
|||
Chińscy naukowcy wykazali, że infekcja wirusem SARS-CoV-2 wywołuje w organizmie aktywację [[Antygen różnicowania komórkowego|limfocytów T CD4<sup>+</sup>]], które następnie różnicują się w komórki [[Limfocyty Th|limfocytów Th1]] i wydzielają [[Czynnik stymulujący tworzenie kolonii granulocytów i makrofagów|czynnik GM-CSF]]. [[Cytokiny]] te indukują inicjujące procesy zapalne [[monocyt]]y CD14<sup>+</sup>CD16<sup>+</sup> z wysoką ekspresją [[Cytokiny|cytokin]] [[Interleukina 6|IL-6]], co przyspiesza rozwój zapalenia płuc i odgrywa rolę w zwiększeniu ryzyka śmiertelności. Mając na uwadze, że u pacjentów z ciężkim przebiegiem [[COVID-19]] zaobserwowano dużą liczbę nacieków zapalnych w płucach, naukowcy sugerują, że wspomniane wcześniej nieprawidłowe patogenne komórki [[Limfocyty Th|Th1]] i monocyty zapalne mogą dostać się w ogromnej liczbie do [[Krążenie małe|krążenia płucnego]] i odgrywać szkodliwą rolę immunologiczną, prowadząc do zaburzeń czynności płuc, a nawet śmierci klinicznej. Z tego powodu autorzy badania sugerują, że przeciwciała monoklonalne skierowane przeciw [[Czynnik stymulujący tworzenie kolonii granulocytów i makrofagów|GM-CSF]] lub [[IL-6]] mogą skutecznie blokować burzę zapalną i stanowić obiecującą terapię pacjentów z ostrym przebiegiem COVID-19<ref>{{Cytuj |autor = Yonggang Zhou, Binqing Fu, Xiaohu Zheng, Dongsheng Wang, Changcheng Zhao |tytuł = Pathogenic T cells and inflammatory monocytes incite inflammatory storm in severe COVID-19 patients |czasopismo = National Science Review |data = 2020-03-13 |data dostępu = 2020-03-21 |issn = 2095-5138 |s = nwaa041 |doi = 10.1093/nsr/nwaa041 |url = https://academic.oup.com/nsr/advance-article/doi/10.1093/nsr/nwaa041/5804736 |język = en}}</ref>. |
Chińscy naukowcy wykazali, że infekcja wirusem SARS-CoV-2 wywołuje w organizmie aktywację [[Antygen różnicowania komórkowego|limfocytów T CD4<sup>+</sup>]], które następnie różnicują się w komórki [[Limfocyty Th|limfocytów Th1]] i wydzielają [[Czynnik stymulujący tworzenie kolonii granulocytów i makrofagów|czynnik GM-CSF]]. [[Cytokiny]] te indukują inicjujące procesy zapalne [[monocyt]]y CD14<sup>+</sup>CD16<sup>+</sup> z wysoką ekspresją [[Cytokiny|cytokin]] [[Interleukina 6|IL-6]], co przyspiesza rozwój zapalenia płuc i odgrywa rolę w zwiększeniu ryzyka śmiertelności. Mając na uwadze, że u pacjentów z ciężkim przebiegiem [[COVID-19]] zaobserwowano dużą liczbę nacieków zapalnych w płucach, naukowcy sugerują, że wspomniane wcześniej nieprawidłowe patogenne komórki [[Limfocyty Th|Th1]] i monocyty zapalne mogą dostać się w ogromnej liczbie do [[Krążenie małe|krążenia płucnego]] i odgrywać szkodliwą rolę immunologiczną, prowadząc do zaburzeń czynności płuc, a nawet śmierci klinicznej. Z tego powodu autorzy badania sugerują, że przeciwciała monoklonalne skierowane przeciw [[Czynnik stymulujący tworzenie kolonii granulocytów i makrofagów|GM-CSF]] lub [[IL-6]] mogą skutecznie blokować burzę zapalną i stanowić obiecującą terapię pacjentów z ostrym przebiegiem COVID-19<ref>{{Cytuj |autor = Yonggang Zhou, Binqing Fu, Xiaohu Zheng, Dongsheng Wang, Changcheng Zhao |tytuł = Pathogenic T cells and inflammatory monocytes incite inflammatory storm in severe COVID-19 patients |czasopismo = National Science Review |data = 2020-03-13 |data dostępu = 2020-03-21 |issn = 2095-5138 |s = nwaa041 |doi = 10.1093/nsr/nwaa041 |url = https://academic.oup.com/nsr/advance-article/doi/10.1093/nsr/nwaa041/5804736 |język = en}}</ref>. |
||
| ⚫ | Główną przyczyną śmierci u pacjentów w przebiegu [[COVID-19]] jest [[Zespół ostrej niewydolności oddechowej|zespół ostrej niewydolności oddechowej (ARDS)]]. Spośród 41 pacjentów zakażonym wirusem SARS-CoV-2 we wczesnej fazie pandemii w Wuhan 6 zmarło właśnie na skutek [[Zespół ostrej niewydolności oddechowej|ARDS]]. Jednym z głównych mechanizmów występujących w ARDS jest niekontrolowana systemowa odpowiedź zapalna w postaci burzy [[Cytokiny|cytokin]] (IFN-α, IFN-γ, IL-1β, IL-6, IL-12, IL-18, IL-33, TNF-α, TGFβ itp.) oraz [[Chemokiny|chemokin]] (CCL2, CCL3, CCL5, CXCL8, CXCL9, CXCL10), co prowadzi w konsekwencji do niewydolności oddechowej oraz niewydolności wielu organów i śmierci<ref name=":6" />. |
||
| ⚫ | |||
===== Przeciwciała SARS-CoV a SARS-CoV-2 ===== |
|||
| ⚫ | Grupa naukowców z [[Melbourne]] pod kierownictwem Katherine Kedzierskiej dokonała studium przypadku pacjentki z łagodnym przebiegiem [[COVID-19]] i przeanalizowała przebieg jej [[Odpowiedź immunologiczna|odpowiedzi immunologicznej]], wskazując na podobieństwa do reakcji immunologicznej organizmu na [[Wirus grypy|wirusa grypy]], będącej przedmiotem wcześniejszych badań tego zespołu. Badacze wykazali podwyższenie poziomu komórek wydzielających swoiste przeciwciała ASC, [[Limfocyty T|limfocytów pomocniczych grudkowych T<sub>FH</sub>]], [[Antygen różnicowania komórkowego|aktywowanych limfocytów CD4<sup>+</sup> i CD8<sup>+</sup>]] wraz z przeciwciałami [[Immunoglobuliny M|IgM]] i [[Immunoglobuliny G|IgG]] wiążącymi [[Koronawirusy|koronawirusa]] SARS-CoV-2. Schemat odpowiedzi immunologicznej może być przydatny przy prognozowaniu przebiegu choroby, opracowaniu szczepionki lub leków łagodzących przebieg choroby. Autorzy postulują jednak kontynuację badań na większej grupie pacjentów o różnych stopniach zaostrzenia choroby COVID-19<ref>{{Cytuj |autor = Irani Thevarajan, Thi H. O. Nguyen, Marios Koutsakos, Julian Druce, Leon Caly |tytuł = Breadth of concomitant immune responses prior to patient recovery: a case report of non-severe COVID-19 |czasopismo = Nature Medicine |data = 2020-03-16 |data dostępu = 2020-03-21 |issn = 1546-170X |s = 1–3 |doi = 10.1038/s41591-020-0819-2 |url = https://www.nature.com/articles/s41591-020-0819-2 |język = en}}</ref>. |
||
| ⚫ | Podczas testów na komórkach zwierzęcych (u myszy) badania wykazały, że [[Przeciwciała monoklonalne|przeciwciała monoklonalne mAb]] mogą rozpoznawać fragmenty glikoproteiny S wirusa SARS-CoV-2<ref name=":15">{{Cytuj |autor = Zhiqiang Zheng, Vanessa M. Monteil, Sebastian Maurer-Stroh, Chow Wenn Yew, Carol Leong |tytuł = Monoclonal antibodies for the S2 subunit of spike of SARS-CoV cross-react with the newly-emerged SARS-CoV-2 |czasopismo = bioRxiv |data = 2020-03-07 |data dostępu = 2020-03-21 |s = 2020.03.06.980037 |doi = 10.1101/2020.03.06.980037 |url = https://www.biorxiv.org/content/10.1101/2020.03.06.980037v1 |język = en}}</ref><ref>{{Cytuj |autor = Kuo-Ming Lip, Shuo Shen, Xiaoming Yang, Choong-Tat Keng, Aihua Zhang |tytuł = Monoclonal Antibodies Targeting the HR2 Domain and the Region Immediately Upstream of the HR2 of the S Protein Neutralize In Vitro Infection of Severe Acute Respiratory Syndrome Coronavirus |czasopismo = Journal of Virology |data = 2006-01-15 |data dostępu = 2020-03-21 |issn = 0022-538X |wolumin = 80 |numer = 2 |s = 941–950 |doi = 10.1128/JVI.80.2.941-950.2006 |pmid = 16378996 |url = https://jvi.asm.org/content/80/2/941 |język = en}}</ref>. W środowisku laboratoryjnym zastosowano cztery mysie [[Przeciwciała monoklonalne|przeciwciała mAb]]: 2B2, 1A9, 4B12 i 1G10 na komórkach COS-7. Wyniki pokazały, że wspomniane cztery [[przeciwciała monoklonalne]] swoiste wobec [[SARS]] mogą reagować krzyżowo także z białkiem S (aa1048-1206) wirusa SARS-CoV-2<ref name=":15" />. Natomiast w osobnym eksperymencie zaobserwowano jedynie umiarkowaną reakcję krzyżową pomiędzy serum z przeciwciałami T62 specyficznymi wobec wirusa SARS, a wirusem SARS-CoV-2, co sugeruje, że przejście infekcji SARS nie chroni w pełni przed infekcją wirusem SARS-CoV-2, i vice versa<ref name=":41" />. |
||
| ⚫ | Analiza kliniczna pacjentów pokazuje, że grupa krwi może wpływać na ryzyko infekcji [[COVID-19]]. Podobnie jak wykazywały wcześniejsze badania [[SARS]] w czasie epidemii w 2019 roku<ref>{{Cytuj |autor = Yufeng Cheng, Gregory Cheng, C. H. Chui, F. Y. Lau, Paul K. S. Chan |tytuł = ABO Blood Group and Susceptibility to Severe Acute Respiratory Syndrome |czasopismo = JAMA |data = 2005-03-23 |data dostępu = 2020-03-21 |issn = 0098-7484 |wolumin = 293 |numer = 12 |s = 1447–1451 |doi = 10.1001/jama.293.12.1450-c |url = https://jamanetwork.com/journals/jama/fullarticle/200582 |język = en}}</ref>, na podstawie wyników badań 2173 pacjentów chorych na COVID-19 z trzech szpitali w [[Wuhan]] i [[Shenzhen]] uważa się, że [[Grupy krwi|grupa krwi]] A zwiększa ryzyko choroby zakaźnej COVID-19, podczas gdy grupa krwi 0 je zmniejsza (lecz nie eliminuje całkowicie)<ref>{{Cytuj |autor = Jiao Zhao, Yan Yang, Han-Ping Huang, Dong Li, Dong-Feng Gu |tytuł = Relationship between the ABO Blood Group and the COVID-19 Susceptibility |czasopismo = medRxiv |data = 2020-03-16 |data dostępu = 2020-03-21 |s = 2020.03.11.20031096 |doi = 10.1101/2020.03.11.20031096 |url = https://www.medrxiv.org/content/10.1101/2020.03.11.20031096v1 |język = en}}</ref>. |
||
== Leczenie == |
== Leczenie == |
||
Wersja z 20:05, 9 kwi 2020
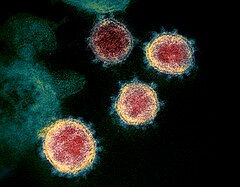 Wirus SARS-CoV-2 (obraz z mikroskopu elektronowego) | |
| Systematyka | |
| Grupa |
Grupa IV ((+)ssRNA) |
|---|---|
| Rząd | |
| Rodzina |
Coronaviridae |
| Rodzaj | |
| Cechy wiralne | |
| Skrót |
SARS-CoV-2 |
| Kwas nukleinowy |
RNA |
| Nagi kwas nukleinowy |
Tak |
| Rezerwuar | |
| Wywoływane choroby | |
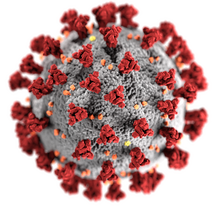
Wirus SARS-CoV-2 (od ang. Severe acute respiratory syndrome coronavirus 2)[a][1][2] – wirus należący do grupy koronawirusów, z pojedynczą nicią o dodatniej polaryzacji ssRNA(+)[3], który wywołuje ostrą chorobę układu oddechowego – COVID-19. Jest to siódmy znany chorobotwórczy dla ludzi gatunek z tej grupy wirusów[4].
Pierwsze przypadki zakażeń ludzi tym wirusem odnotowano pod koniec 2019 roku w mieście Wuhan w środkowych Chinach[5][6]. Pierwsze zakażenie miało miejsce w dniu 17 listopada 2019 r., zarażonym był 55-letni mężczyzna, mieszkaniec Wuhan[7][8]. Istnieją przypuszczenia, że źródłem, z którego wirus mógł się rozprzestrzenić, był targ rybny Huanan[9][10], gdzie sprzedawane były zarówno dzikie zwierzęta, jak i owoce morza[11][12][13]. Wirus przenosi się drogą kropelkową, a u niektórych chorych wywołuje ciężkie zapalenie płuc i ARDS wymagający zastosowania wentylacji mechanicznej[14][15].
Odkrycie
Zachorowania powodowane niezidentyfikowanym wirusem rozpoczęły się w listopadzie 2019[16]. Odnotowano wówczas grupę pacjentów cierpiących na zapalenie płuc nieznanej etiologii. Zachorowania te udało się powiązać z miejscem sprzedaży owoców morza w Wuhan w chińskiej prowincji Hubei[4], gdzie handlowano też żywymi zwierzętami[17]. Ostatniego dnia roku Chińskie Centrum Kontroli i Prewencji Chorób (CCDC, od ang. Chinese Center for Disease Control and Prevention; chiń. 中国疾病预防控制中心) wysłało tam zespół mający wspomóc władze lokalne. Przeprowadzono dochodzenie epidemiologiczne mające ukazać przyczynę zachorowań[4]. Udało się wykluczyć koronawirusy powodujące SARS i MERS, wirusy wywołujące grypę ludzką i ptasią, a także inne popularne wirusy powodujące infekcje dróg oddechowych[17]. 7 stycznia 2020 potwierdzono oficjalnie, że zachorowania powoduje nowy wirus z grupy koronawirusów, nazwany roboczo 2019-nCoV[18], a 5 dni później dysponowano już jego zsekwencjonowanym genomem[17]. W nabłonku dróg oddechowych trzech pacjentów udało się bowiem wykryć i wyizolować wirusa, którego genom cechował się dużym podobieństwem (identyczne w 85%) do beta-koronawirusów linii B izolowanych wcześniej z nietoperzy – bat SARS-like CoV (bat-SL-CoVZC45, MG772933.1)[4][19].
Budowa molekularna

SARS-CoV-2 jest wirusem osłonkowym, którego genom stanowi jednoniciowe RNA o polarności dodatniej[20]. Każdy wirion SARS-CoV-2 ma kształt zasadniczo kulisty, choć nieco pleomorficzny, o średnicy 60–140 nm. Otoczony jest wyraźnymi wypustkami (ang. spikes) o długości 9–12 nm, nadającymi mu wygląd podobny do korony słonecznej[4]. Na dzień 13 marca 2020 roku przeprowadzono przynajmniej 75 kompletnych analiz genomu izolatów wirusa SARS-CoV-2[21]. Długość genomu wynosi od 29867 do 29903 nukleotydów[21] (dokładna liczba zależy od źródła i wynosi przykładowo 29891[20] lub 29903[22]), co czyni go, podobnie jak w przypadku innych koronawirusów, jednym z największych wirusów RNA, zarówno pod względem długości genomu, jak i rozmiaru wirionu[23].
Genom SARS-CoV-2 koduje białka niestrukturalne (ang. nonstructural proteins lub nsp; potrzebne do replikacji), strukturalne oraz pomocnicze (ang. accessory proteins)[24][25][26]. Podobnie jak inne koronawirusy, SARS-CoV-2 posiada cztery białka strukturalne[23][27]:
- S (ang. spike) – białko fuzyjne lub glikoproteina powierzchniowa – odpowiedzialne za interakcję z receptorem na powierzchni komórek;
- E (ang. envelope) – białko płaszcza – odpowiedzialne m.in. za formowanie wirionów;
- M (ang. membrane) – białko błonowe lub membranowe – główne białko macierzy wirusa;
- N (ang. nucleocapsid) – białko nukleokapsydu – pełniące funkcję ochronną dla dużej cząsteczki RNA oraz uczestniczące w modyfikacji procesów komórkowych i replikacji wirusa.
Białko N utrzymuje genom RNA, a białka S, E i M tworzą razem otoczkę wirusa. Białko S jest odpowiedzialne za łączenie z błoną komórki gospodarza[27]. Glikoproteinę S funkcjonalnie różnicuje się na podjednostki S1 i S2. Podjednostka S1 pośredniczy w wiązaniu z receptorem powierzchniowym komórki gospodarza, a podjednostka S2 pośredniczy w fuzji z jej błoną komórkową i następnie wirus dostaje się do komórki poprzez endocytozę[28].
Porównanie białka S genomu SARS-CoV-2 z dwoma innymi izolatami wirusów pochodzących od nietoperzy: bat-SL-CoVZC45 i bat-SL-CoVZXC21 pokazuje niską, 75% zgodność. Szczególnie duże różnice wykazuje sekwencja nukleotydowa kodująca podjednostkę S1 w SARS-CoV-2 (zgodność w 68%), co czyni ją bardziej zbliżoną do odpowiedniej sekwencji nukleotydowej SARS-CoV. Można na tej podstawie wywnioskować, że SARS-CoV i nowy koronawirus wykorzystują ten sam receptor komórkowy, enzym konwertazy angiotensyny 2 (ACE2)[28]. Zostało to udowodnione eksperymentalnie, jak opisano w dalszej sekcji patogeneza.
W porównaniu z SARS zaobserwowano obecność czterech dodatkowych insercji reszt aminokwasowych na granicy pomiędzy podjednostkami S1 i S2[29].
| Proteina | Masa molowa [kDa] | Podobieństwo do SARS-CoV | Opis funkcji |
|---|---|---|---|
| Nsp1 | 19,8 | 91,1% | Tłumienie odpowiedzi przeciwwirusowej w organizmie gospodarza |
| Nsp2 | 70,5 | 82,9% | |
| Nsp3 | 217,3 | 86,5% | Kompleks białek Nsp3-Nsp4-Nsp6 bierze udział w replikacji wirusa |
| Nsp4 | 56,2 | 90,8% | Kompleks białek Nsp3-Nsp4-Nsp6 bierze udział w replikacji wirusa |
| Nsp5 | 33,8 | 98,7% | Główna proteaza (Mpro lub 3CLpro) |
| Nsp6 | 33,0 | 94,8% | Kompleks białek Nsp3-Nsp4-Nsp6 bierze udział w replikacji wirusa |
| Nsp7 | 9,2 | 100,0% | Kompleks białek Nsp7-Nsp8 jest częścią polimerazy RNA |
| Nsp8 | 21,9 | 99,0% | Kompleks białek Nsp7-Nsp8 jest częścią polimerazy RNA |
| Nsp9 | 12,4 | 98,2% | Wiązanie ssRNA |
| Nsp10 | 14,8 | 99,3% | Istotne dla aktywności metylotransferazy Nsp16 |
| Nsp11 | 1,3 | 92,3% | Peptyd krótki |
| Nsp12 | 106,7 | 98,3% | Polimeraza RNA |
| Nsp13 | 66,9 | 100,0% | Helikaza/trifosfataza |
| Nsp14 | 59,8 | 98,7% | Egzonukleaza typu 3'→5' |
| Nsp15 | 38,8 | 95,7% | Endorybonukleaza specyficzna wobec urydyny |
| Nsp16 | 33,3 | 98,0% | Metylotransferaza czapeczki RNA |
| S | 141,2 | 87,0% | Białko fuzyjne, pośredniczy w fuzji z receptorem powierzchniowym |
| Orf3a | 31,1 | 85,1% | Aktywuje inflamasom NLRP3 |
| Orf3b | 6,5 | 9,5% | |
| E | 8,4 | 96,1% | Białko płaszcza, biorące udział w morfogenezie i składaniu filamentów wirusowych |
| M | 25,1 | 96,4% | Białko błonowe, dominujący składnik otoczki |
| Orf6 | 7,3 | 85,7% | Antagonista IFN typu I |
| Orf7a | 13,7 | 90,2% | Wywołuje indukowaną przez wirusa apoptozę |
| Orf7b | 5,2 | 84,1% | |
| Orf8 | 13,8 | 45,3% | |
| N | 45,6 | 94,3% | Białko (fosfoproteina) nukleokapsydu, związane z genomem RNA wirusa |
| Orf9b | 10,8 | 84,7% | Antagonista IFN typu I |
| Orf9c | 8,0 | 78,1% | |
| Orf10 | 4,4 | – |
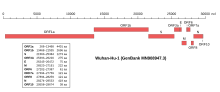
Filogenetyka
Sekwencje koronawirusa SARS-CoV-2 wykazują podobieństwa do beta-koronawirusów znalezionych u nietoperzy[31]. Wirus ten jest jednak genetycznie odmienny od innych koronawirusów, jak koronawirus związany z SARS i koronawirus związany z MERS[32].
Wyizolowano i opisano pięć genomów nowego koronawirusa, w tym[33]:
- BetaCoV/Wuhan/IVDC-HB-01/2019
- BetaCoV/Wuhan/IVDC-HB-04/2020
- BetaCoV/Wuhan/IVDC-HB-05/2019
- BetaCoV/Wuhan/WIV04/2019
- BetaCoV/Wuhan/IPBCAMS-WH-01/2019.
| Identyfikator genomu NCBI | MN908947 |
|---|---|
| Rozmiar genomu | 30473 zasady |
| Rok ukończenia | 2020 |
Zalecany stopień bezpieczeństwa biologicznego (BSL) podczas prac z wirusem
Wirus SARS-CoV-2 został zakwalifikowany do grupy 3 ryzyka mikrobiologicznego.
Światowa Organizacja Zdrowia zaleca przeprowadzanie prac z żywym wirusem, który może się namnażać (na przykład hodowli wirusów, izolacji wirusów, testów neutralizacji) w laboratoriach o minimum 3 stopniu bezpieczeństwa biologicznego z przepływem powietrza ukierunkowanym do wewnątrz (BSL-3).
Z kolei laboratoryjne prace diagnostyczne z preparatami inaktywowanymi (bez możliwości replikacji wirusa), takie jak na przykład sekwencjonowanie białek wirusa lub testy molekularne amplifikacji kwasów nukleinowych (NAAT) powinny być przeprowadzane w laboratoriach o minimum 2 stopniu bezpieczeństwa biologicznego (BSL-2)[34][35].
Objawy infekcji

Opisano serię przypadków osób zainfekowanych wirusem w Chinach. Pacjenci zgłaszali gorączkę i kaszel. Po czterech dniach u jednego z nich kaszel się nasilił, za to gorączka spadła. Z pierwszych trzech opisanych przypadków dwóch wyzdrowiało, stan jednego się pogorszył. Doszło do konieczności użycia wentylacji mechanicznej; leczenie zakończyło się zgonem[4].
Badania krwi często wykazywały małą liczbę białych krwinek (leukopenia i limfopenia)[17].
Wirulencja i patogeneza
Dane wskazują, że w porównaniu z wirusami SARS i MERS wirus SARS-CoV-2 jest mniej wirulentny ze znacznie niższymi wskaźnikami śmiertelności, za to dużo bardziej zakaźny. Eliminacja nowego wirusa następuje najprawdopodobniej szybciej niż SARS i MERS, chociaż dane dotyczące przypadków krytycznych są w tym kontekście niejednoznaczne[36][37].
Charakterystyka zakażeń wywoływanych przez wirus SARS-CoV-2
Wirus SARS-CoV-2 może kolonizować i atakować[38][39][40][41][42][43][44][45][46][47][48][49][36]:
- układ oddechowy – powodując w ciężkich przypadkach ostre atypowe zapalenie płuc oraz zespół ostrej niewydolności oddechowej;
- układ nerwowy – wywołując objawy neurologiczne, jak ból głowy, nudności, uczucie splątania, zaburzenia świadomości, a w ciężkich przypadkach choroby naczyniowo-mózgowe. W przypadku wirusa SARS istnieją badania in vivo mówiące również o możliwości infekcji pnia mózgu[50];
- układ pokarmowy – powodując między innymi biegunkę i wymioty. Badania wykazały obecność wirusa w kale;
- układ moczowy – wywołując powikłania takie jak ostre uszkodzenie nerek. Badania wykazały obecność wirusa w moczu pacjentów;
- układ krwionośny – przyczyniając się do ostrej niewydolności serca.
Wirus może indukować nadmierną odpowiedź immunologiczną w organizmie, co przejawia się zwiększeniem poziomów cytokin IL2, IL7, IL10, G-CSF, IP10, MCP1, MIP1A i TNFα[51]. Zobacz również sekcję odpowiedź immunologiczna.
W skrajnych przypadkach SARS-CoV-2 może doprowadzić do infekcji ogólnoustrojowej (sepsy), a RNA wirusa dostać się do krwi (wiremia).
Infekcja wirusem jest niespecyficzna i może przebiegać bezobjawowo lub z łagodnymi objawami, a pierwotnym szlakiem zakażenia jest najczęściej układ oddechowy. Czynniki kolonizacji nie są jednak do końca poznane i wymagają dalszych badań[52][53].
Drogi szerzenia infekcji
20 stycznia 2020 r. Narodowa Komisja Zdrowia Chin ogłosiła, że możliwe jest przekazywanie wirusa z człowieka na człowieka[54][55], zwłaszcza gdy dwie osoby są ze sobą w bliskim kontakcie (odległość mniejsza niż 1,8 m[56] lub odległość mniejsza niż 1–2 m[57]). Uważa się, że rozprzestrzenia się on podobnie jak inne patogeny chorób układu oddechowego drogą kropelkową[14][56][57][58]. Do tej pory wirus został wykryty w wydzielinie z nosa i gardła, w plwocinie, moczu, stolcu, cieczy łzowej i krwi[59][60][61][62].
Singapurscy naukowcy zalecają traktowanie kału jako zakaźnego w oparciu o wykrycie wirusa w kale oraz zakaźnego wirusa SARS-CoV w ściekach z dwóch chińskich szpitali podczas pandemii SARS 2002/2003. Aby wykluczyć możliwość infekcji fekalno-oralnej oprócz infekcji kropelkowej i inhalacyjnej, konieczne są dalsze badania zarówno dróg wydalania wirusa przez pacjenta, jak i potencjalnych zanieczyszczeń środowiskowych[63]. W badaniu na niewielkiej grupie osób wykazano, że wysokie miano wirusa na tekstyliach i powierzchniach przedmiotów można wykryć w sypialniach i łazienkach osób zakażonych koronawirusem nawet metodą PCR. Ograniczeniem badania był brak hodowli wirusów w celu wykazania ich żywotności, zatem zakaźność nie została ostatecznie udowodniona, ale autorzy badania zakładają na podstawie swoich wstępnych wyników, że prawdopodobne jest przeniesienie wirusa przez zakażone przedmioty[64]. Również nowsze badania SARS-CoV-2 wykazują obecność wirusa w kale, co sugeruje możliwość jego replikacji w przewodzie pokarmowym[65].
Na podstawie analiz ilościowych badań RT-PCR nosowej części gardła (nasopharynx) chińscy naukowcy wywnioskowali, że wirus, podobnie jak grypa, może być przenoszony również drogą wziewną (powietrzną)[66][67]. U 17 badanych pacjentów z objawami choroby miano wirusa w nosie było wyższe niż w gardle, a występowanie szczególnie w górnych drogach oddechowych odróżnia SARS-CoV-2 od wirusa wywołującego SARS[66]. Testy pacjentów z obszaru Monachium nie wykazały wirulencji z próbek kału pomimo wykrycia RNA wirusa. Miano wirusa w nosogardzieli było jednak 1000 razy wyższe niż w znanych wcześniej chorobach wywołanych koronawirusami, takimi jak SARS i MERS[68]. Dowodzi to, że wirus replikuje się już w górnych drogach oddechowych, chociaż dopiero w głębszych obszarach płuc może wchodzić w fuzję z komórkami gospodarza i namnażać się poprzez wiązanie z receptorem błonowym ACE-2 (konwertaza angiotensyny 2; kininaza II) dzięki domenie wiążącej receptor (ang. Receptor Binding Domain) zakodowanej w białku fuzyjnym S (ang. Spike) wirusa SARS-CoV-2. Rozszerzenie tropizmu tkankowego do obszarów gardła prowadzi do założenia, że wirus zawiera proteazę furynopodobną, która jest niezbędna na etapie cięcia enzymatycznego (ang. Cleaving) w celu wejścia do komórki gospodarza i następnie replikacji w niej[69][70][71].
Nie jest jasne, czy transmisja wirusa odbywa się poprzez kontakt z zanieczyszczonymi powierzchniami i przedmiotami[56]. Analiza 22 badań dotyczących przeżywalności koronawirusów istotnych z medycznego punktu widzenia (takich jak SARS-CoV i MERS-CoV) na powierzchniach pokazuje, że wirusy te mogą utrzymywać się na powierzchniach materiałów wykonanych z metalu, szkła lub tworzywa sztucznego do dziewięciu dni w temperaturze pokojowej. Pozostają jednak zakaźne przez średnio cztery do pięciu dni. Jednocześnie mogą one zostać inaktywowane odpowiednimi środkami dezynfekującymi. Według naukowców zaangażowanych we wspomniane badania odkrycia te można zastosować do SARS-CoV-2[72][73].
Temperatura wpływa na aktywność i przeżywalność wirusa SARS-CoV-2, który jest wysoce stabilny w temperaturze 4 °C i może w niej przetrwać znacznie dłużej niż 14 dni. W temperaturze pokojowej (22 °C) i wyższej przeżywalność wirusa nie przekracza 14 dni, a w temperaturze 70 °C czas inaktywacji wirusa jest zredukowany do 5 minut. W temperaturze pokojowej wirus SARS-CoV-2 jest wyjątkowo stabilny w szerokim zakresie pH, wynoszącym od 3 do 10[74].
Mydło inaktywuje wirusa poprzez niszczenie jego otoczki lipidowej[75][76]. Badania potwierdziły również skuteczność środków dezynfekujących na bazie alkoholu w walce z SARS-CoV-2[77].
W badaniu z udziałem dziewięciu pacjentek, które były zakażone SARS-CoV2 w ostatnim trymestrze ciąży, każde z dziewięciorga dzieci okazało się wolne od infekcji wirusem po urodzeniu przez cesarskie cięcie. Na tej podstawie autorzy badania stwierdzili, że wirus nie przenosi się w łonie matki[78]. Do 6 lutego 2020 roku chińskie instytucje zdrowia publicznego zarejestrowały tylko dziewięcioro niemowląt, u których wykazano dowody na obecność wirusa. Autorzy badania za możliwą przyczynę tej niewielkiej liczby wskazują stosunkowo dużą liczbę przypadków skąpoobjawowych wśród dzieci, a także niedostateczny system raportowania przypadków[79].
Ponieważ SARS-CoV-2 jest uznawany za wirus odzwierzęcy, przypuszcza się, że możliwa jest transmisja pomiędzy niektórymi zwierzętami i ludźmi[14][15][80]. Jednocześnie Światowa Organizacja Zdrowia stwierdziła, że nie ma wystarczających dowodów na to, że zwierzęta domowe, takie jak koty czy psy, mogą zarazić się koronawirusem[81]. Dyskusyjny jest pojedynczy przypadek z 28 lutego 2020 roku „lekko pozytywnego” wyniku testu na COVID-19 u psa w Hongkongu, przy czym w oświadczeniu wydanym przez Ministerstwo Rolnictwa, Rybołówstwa i Rezerwatów Przyrody Hongkongu (ang. Hong Kong Agriculture, Fisheries and Conservation Department) oświadczono o braku dowodów na możliwość infekcji COVID-19 u zwierząt domowych czy możliwości przeniesienia infekcji ze zwierzęcia domowego na człowieka[82].
Ze względu na rozmiar (od 60 do 140 nm) wirus jest przepuszczany przez maski z filtrami FFP1, P1,FFP2, P2, N95, N99, FFP3, P3, N100 (filtracja wszystkich cząstek o średnicy 0,3 mikrona lub większych)[83].
Zaraźliwość (współczynnik reprodukcji wirusa R0)
Istnieje wiele obliczeń współczynnika reprodukcji wirusa R0, które wykorzystują różnorodne metody kalkulacji.
Uwzględniając dostępne publikacje, różne raporty szacują wskaźnik R0 w zakresie 1,4 do 6,4, lecz wszyscy uznają, że współczynnik reprodukcji wirusa jest R0 > 1[36][84][85][86][87][88][89][90][91][92].
Raport Europejskiego Centrum Zapobiegania i Kontroli Chorób z 12 marca 2020 roku szacuje współczynnik reprodukcji wirusa R0 = 2-3[93]. Wydaje się to pokrywać z innymi szacunkami[36][84][85][86][87][88][89][90][91][92].
Przyjęty w innym badaniu stochastyczny model dynamiki transmisji użyty do obliczenia współczynnika reprodukcji wirusa zależnego od czasu Rt sugeruje zmienność transmisji wirusa w czasie i jej zmniejszenie dzięki wprowadzeniu restrykcji w podróżach i ograniczeniu kontaktu pomiędzy ludźmi[94].
Zakaźność
Najważniejsze statystyki zakaźności przedstawiono poniżej:
- wirus może infekować wszystkie grupy populacji[95];
- mediana wieku wynosi 47 lat[96]'
- wiek stanowi istotny czynnik prognostyczny zachorowalności; 80% stanowią osoby w wieku 15–59 lat, zachorowalność jest mniejsza wśród dzieci[96];
- płeć męska jest bardziej zagrożona wirusem, ok. 56% przypadków to mężczyźni[96]; wynik ten jest zgodny z charakterystyką wirusów SARS i MERS; naukowcy spekulują, że wynika to z roli hormonów oraz chromosomów i ich wpływu na odpowiedź immunologiczną[97][95];
- na ciężki przebieg infekcji wirusem SARS-CoV-2 narażeni są dorośli w każdym wieku[98].
Poniższe statystyki pokazują zakaźność wirusa według wieku i płci na podstawie łącznej liczby przypadków z Włoch, Hiszpanii i Chin według danych dostępnych na dzień 25 marca 2020 roku[99][100][101].
| Grupa wiekowa | 0-9 | 10-19 | 20-29 | 30-39 | 40-49 | 50-59 | 60-69 | 70-79 | 80+ |
|---|---|---|---|---|---|---|---|---|---|
| Liczba zakażeń | 0,7% | 0,9% | 5,7% | 11,1% | 15,4% | 20,0% | 18,1% | 15,2% | 12,8% |
Nie istnieje wiele badań koncentrujących się na przyczynach różnic w zachorowalności i śmiertelności wirusa u różnych płci i w różnych grupach wiekowych, które mogłyby wskazać, czy istotną rolę odgrywają cechy immunologiczne poszczególnych grup, czy cechy fenotypowe samego wirusa, a tym samym wskazać dalsze możliwości leczenia. Prace badawcze wykryły podwyższone miano wirusa u osób starszych, co może korelować z ciężkim przebiegiem choroby w tych grupach wiekowych. Część naukowców spekuluje, że może to być związane z podwyższoną ekspresją receptora ACE2 u starszych pacjentów[102].
Analiza kliniczna pacjentów pokazuje, że grupa krwi może wpływać na ryzyko infekcji COVID-19. Podobnie jak wykazywały wcześniejsze badania SARS w czasie epidemii w 2019 roku[103], na podstawie wyników badań 2173 pacjentów chorych na COVID-19 z trzech szpitali w Wuhan i Shenzhen uważa się, że grupa krwi A zwiększa ryzyko choroby zakaźnej COVID-19, podczas gdy grupa krwi 0 je zmniejsza (lecz nie eliminuje całkowicie)[104].
Zjadliwość i wskaźniki śmiertelności
Do marca 2020 roku z powodu zakażenia wirusem SARS-CoV-2 zmarło znacznie więcej osób niż z powodu zakażenia wirusami SARS i MERS łącznie. Mimo niższej śmiertelności w porównaniu z poprzednimi wirusami, na wynik ten wpływa wyższa zakaźność wirusa[105][36].
Śmiertelność wirusa SARS-CoV-2 szacuje się na 3,4%[106][36].
Na uwagę zasługuje wyjątkowo niskie prawdopodobieństwo śmiertelności u dzieci poniżej 9 lat, a także wyższy wskaźnik śmiertelności u mężczyzn (4,7%) niż u kobiet (2,8%)[107][108]. 22 marca 2020 roku poinformowano o pierwszym przypadku śmiertelności u niemowlęcia (Illinois)[109].
Mutageneza
Z perspektywy etiologii wirulencja patogenu czasami zwiększa się podczas przeskoku na nowego gospodarza. Inne badania pokazują natomiast, że wirulencja pasożytów zmniejsza się podczas długotrwałych interakcji pasożytów z jednym gospodarzem[110]. Koronawirusy mogą jednak dokonywać mutacji w celu lepszej adaptacji do organizmów ludzkich i zwiększenia wirulencji, tak jak to historycznie miało miejsce w latach 2002–2004 w przypadku SARS[111][112][113]. Drobne mutacje i rekombinacje są naturalne u koronawirusów i stanowią część ich naturalnego cyklu życiowego. Ponieważ wirusy te wykorzystują podatną na błędy replikacji RNA polimerazę, ich genom ma skłonność do gromadzenia mutacji podczas każdego cyklu kopiowania[112][114]. Naturalna selekcja odrzuca zmiany niekorzystne, a promuje zmiany, które dają wirusom korzyści ewolucyjne[113]. Z powyższych powodów istotne jest zrozumienie ryzyka mutacji wirusa SARS-CoV-2 i ich wpływu na wirulencję. Dostępne są wyniki wstępnych badań w tym zakresie[114][115][113][116][117][118][119].
W okresie od grudnia 2019 do lutego 2020 zaobserwowano 8 drobnych mutacji aminokwasów w badaniach 55 genomów, co jednak nie przełożyło się na duże zmiany funkcjonalne w zachowaniu wirusa SARS-CoV-2[113]. Alarmujące są zmiany w obrębie glikoproteiny S w położeniu 28144 (obszar genu ORF8)[114][113]. Inna analiza szczepów wirusa z różnych części świata opublikowana 17 marca 2020 roku wykazała mutacje w 10 z 244 badanych szczepów. Zaobserwowano zmiany w domenie wiążącej receptor białka S, co przypuszczalnie wiąże się z dalszą adaptacją wirusa do organizmów ludzkich[116][118]. Istnieją również dowody, że wirus SARS-CoV-2 ma już przynajmniej 2 podtypy genetyczne[120].
Ponieważ nie ma jasności wśród naukowców, czy wirus może ewoluować w kierunku większej zjadliwości i pandemiczności, istotne są dalsze badania porównawcze[117][118][120].
Okres inkubacji
Okres inkubacji określa czas od zakażenia wirusem do momentu wystąpienia choroby i wynosi do 14 dni[96]. Istnieją pojedyncze dowody, że wirus jest zakaźny podczas inkubacji, co odróżniałoby go od SARS[121][122][123], chociaż nie ma ogólnej zgodności naukowców, jaka jest rola pacjentów asymptomatycznych i przed-symptomatycznych w transmisji wirusa[124][125][126]. Równolegle badacze odkryli, że miano wirusa u pacjentów osiąga najwyższy poziom w pierwszym tygodniu choroby, a następnie stopniowo opada w drugim tygodniu, co sugerowałoby, że wczesne etapy choroby są najbardziej transmisyjne[127].
Patogeneza
Mechanizmy fuzji komórkowej i replikacji
Białko fuzyjne S wirusa (ang. Spike) odgrywa kluczową rolę w procesie wnikania wirusa do komórek gospodarza[128].
Eksperymenty modelowania z użyciem białka S, bazujące na przewidywaniu struktury białek (ang. protein structure prediction), bardzo wcześnie sugerowały, że SARS-CoV-2 posiada odpowiednie powinowactwo do białka receptorowego ACE2 (enzymu konwertaza angiotensyny 2), które może zostać wykorzystane jako mechanizm wnikania do komórek gospodarza[129]. Do 22 stycznia 2020 r. grupa naukowców pracująca nad pełnym genomem wirusa w Chinach oraz grupa naukowców stosująca metody genetyki odwrotnej w Stanach Zjednoczonych wykazały niezależnie i eksperymentalnie, że ACE2 może działać jako receptor dla SARS-CoV-2[130][131][132][133][134][28]. Potwierdziły to dalsze prace[29][135]. Badania wykazały, że SARS-CoV-2 posiada większe powinowactwo do ludzkiego enzymu ACE2 niż wcześniejszy szczep wirusa SARS[136]. W eksperymencie z komórkami HeLa, które zawierały enzym ACE2 ludzi, nietoperzy z gatunku Rhinolophus sinicus, łaskunów, świń i myszy, SARS-CoV-2 był w stanie wykorzystać odpowiednie białko receptorowe ACE2 w celu wniknięcia do komórki. Wirus wyjątkowo nie był w stanie wykorzystać mysiego ACE2 ani łączyć się z komórkami HeLa, które nie tworzyły ACE2. Eksperyment wykazał również, że SARS-CoV-2 nie korzysta z innych receptorów typowych dla innych koronawirusów, takich jak aminopeptydaza N (APN) czy dipeptylopeptydaza IV (DPP4)[137]. Kolejne badania wykazały, że SARS-CoV-2 przedostaje się do komórek 293/hACE2 głównie poprzez endocytozę. Enzym PIKfyve, TPC2 i katepsyna L (lecz nie B) odgrywają krytyczną rolę w procesie ich połączenia[135].
Oprócz białka ACE2 istotnym czynnikiem patogenezy CoV-2 jest enzym seryna 2 (TMPRSS2), który obok ACE2 został przez naukowców uznany za kluczowy w procesie wnikania do komórek gospodarza. Proteaza TMPRSS2 aktywuje proces fuzji komórkowej z białkiem S wirusa SARS-CoV-2 oraz indukuje zależną od aktywności receptora formację syncytium. Mimo że trypsyna jest w stanie również aktywować glikoproteinę S i wyindukować dużą formację syncytium, wirus SARS-CoV-2 może najprawdopodobniej bez udziału trypsyny utworzyć syncytium niezależną od proteazy, lecz zależną od aktywności receptora[135]. Sugeruje się, że blokowanie TMPRSS2 może być kluczową strategią leczenia pacjentów z COVID-19[138][139][140].
Niezależne zespoły naukowców z Chin oraz Francji i Kanady, a w dalszej kolejności innych zespołów wykazały obecność pomiędzy podjednostkami S1 i S2 wirusa CoV-2 genów furynopodobnych[69][141][142][143][29][135]. Sugeruje to możliwość wykorzystania enzymu furyny (ang. furin) do aktywacji procesu fuzji osłonki wirusa i komórek organizmu. Podczas gdy glikoproteina S1 odpowiada za pierwszy etap wnikania wirusa do komórki, glikoproteina S2 może być wykorzystywana podczas drugiej fazy fuzji osłonki wirusa oraz komórki gospodarza[144]. Wspomniana mutacja genu w obrębie glikoproteiny S1/S2 nie jest obecna w betakoronawirusach linii B, lecz jest podobna do genów występujących w innych wirusach (HIV, Ebola) oraz betakoronawirusach linii A (HKU1, HCoV-OC43) i C (MERS-CoV)[69]. Prawdopodobnie powoduje to w porównaniu z SARS zwiększenie tropizmu tkankowego oraz zwiększenie transmisyjności i patogeniczności wirusa[29]. Niniejsze odkrycie oznacza, że sposób infekcji wirusa SARS-CoV-2 może być odmienny i groźniejszy niż w przypadku SARS[145]. Mechanizm mediacji procesu fuzji z komórkami gospodarza za pomocą furyny może implikować potencjalną strategię przeciwwirusową opartą na hamowaniu tego enzymu[69][142][141][146][143].
14 marca 2020 chińska grupa badaczy przedstawiła publikację, a której stwierdziła, że SARS-CoV-2 może wykorzystywać receptor białkowy CD147 jako dodatkową drogę inwazji komórek gospodarza. W badaniach in vitro naukowcy zaobserwowali, że przeciwciała monoklonalne skierowane przeciw CD147 (lek przeciwmalaryczny meplazumab) blokuje interakcję pomiędzy receptorem CD147 a glikoproteiną S wirusa SARS-CoV-2[147][148]. Lek meplazumab aktualnie poddawany jest ocenie klinicznej[149]. Wymagane są jednak dalsze badania w celu potwierdzenia tego odkrycia i opracowania skutecznego leczenia.
Replikacja wirusa

Reasumując, aktualnie udowodniono, że glikoproteina S1 wirusa SARS-COV-2 łączy się z receptorem ACE2 znajdującym się w wielu tkankach, głównie płucach, nerkach oraz w jelicie cienkim, oraz że proces wewnątrzkomórkowej fuzji wymaga aktywacji glikoproteiny S2 za pomocą cięcia enzymatycznego przez proteinę TMPRSS2[150]. Dalszych badań wymaga jednak wciąż udział innych protein (np. furyna, CD147) w procesie fuzji, co postulują naukowcy[145][151][152][153].
W powyżej opisany sposób wirion wnika do komórki poprzez endocytozę i uwalnia RNA do cytoplazmy[135][154].
Po uwolnieniu wirusowego RNA do cytoplazmy komórki gospodarza, realizowana jest synteza poliprotein 1a/1ab (pp1a/pp1ab), produkowanych przez białka ORF1a i ORF1b[80][155][156]. Transkrypcja realizowana jest przez kompleks RCT, zorganizowany w pęcherzyki z podwójną błoną, i poprzez syntezę sekwencji subgenomowego RNA (sgRNA)[80]. Zakończenie transkrypcji występuje w sekwencjach regulatorowych, zlokalizowanych między otwartymi ramkami odczytu (ORF), które działają jako matryce do produkcji subgenomowych mRNA. Wszystkie białka strukturalne (N, S, M, E) i pomocnicze są translowane z sgRNA koronawirusa. Szesnaście białek niestrukturalnych powstaje poprzez cięcie za pomocą proteaz 3CLpro, Mpro i jednej lub dwóch proteaz z rodziny papainy[80][150].
Nowo uformowane glikoproteiny płaszcza (E) są wstawiane do otoczki siateczki śródplazmatycznej lub aparatu Golgiego i przez połączenie genomowego RNA i białka nukleokapsydu (N) powstaje nukleokapsyd. Następnie cząstki wirusa łączą się w kompartment ERGIC (ang. endoplasmic reticulum-Golgi intermediate compartment). W końcu od wewnątrz komórki następuje fuzja pęcherzyków zawierających cząstki wirusa z błoną komórkową (egzocytoza) w celu uwolnienia nowego wirionu[154][155].
Słabo poznany jest sam proces egzocytozy wirusa.
Immunopatologia
Swoista odpowiedź immunologiczna odgrywa istotną rolę w infekcji SARS-CoV-2[157]. Wirus może z jednej strony unikać nadzoru immunologicznego w organizmie[158], z drugiej wywołać w ciężkich przypadkach burzę cytokin[159].
Osłabienie układu odpornościowego
SARS-CoV-2 wytwarza co najmniej trzy czynniki wirulencji, które promują rozprzestrzenianie się nowych wirionów z komórek gospodarza i hamują odpowiedź immunologiczną: Nsp1, Nsp3c oraz ORF7a[160].
Indukcja burzy cytokin
Spośród białek strukturalnych glikoproteina S (zwłaszcza domena A białka S1 oraz domena wiążąca receptor RBD) i N są najważniejszymi immunogenami[161][162].
Grupa naukowców z Melbourne pod kierownictwem Katherine Kedzierskiej dokonała studium przypadku pacjentki z łagodnym przebiegiem COVID-19 i przeanalizowała przebieg jej odpowiedzi immunologicznej, wskazując na podobieństwa do reakcji immunologicznej organizmu na wirusa grypy, będącej przedmiotem wcześniejszych badań tego zespołu. Badacze wykazali podwyższenie poziomu komórek wydzielających swoiste przeciwciała ASC, limfocytów pomocniczych grudkowych TFH, aktywowanych limfocytów CD4+ i CD8+ wraz z przeciwciałami IgM i IgG wiążącymi koronawirusa SARS-CoV-2. Schemat odpowiedzi immunologicznej może być przydatny przy prognozowaniu przebiegu choroby, opracowaniu szczepionki lub leków łagodzących przebieg choroby. Autorzy postulują jednak kontynuację badań na większej grupie pacjentów o różnych stopniach zaostrzenia choroby COVID-19[163].
Chińscy naukowcy wykazali, że infekcja wirusem SARS-CoV-2 wywołuje w organizmie aktywację limfocytów T CD4+, które następnie różnicują się w komórki limfocytów Th1 i wydzielają czynnik GM-CSF. Cytokiny te indukują inicjujące procesy zapalne monocyty CD14+CD16+ z wysoką ekspresją cytokin IL-6, co przyspiesza rozwój zapalenia płuc i odgrywa rolę w zwiększeniu ryzyka śmiertelności. Mając na uwadze, że u pacjentów z ciężkim przebiegiem COVID-19 zaobserwowano dużą liczbę nacieków zapalnych w płucach, naukowcy sugerują, że wspomniane wcześniej nieprawidłowe patogenne komórki Th1 i monocyty zapalne mogą dostać się w ogromnej liczbie do krążenia płucnego i odgrywać szkodliwą rolę immunologiczną, prowadząc do zaburzeń czynności płuc, a nawet śmierci klinicznej. Z tego powodu autorzy badania sugerują, że przeciwciała monoklonalne skierowane przeciw GM-CSF lub IL-6 mogą skutecznie blokować burzę zapalną i stanowić obiecującą terapię pacjentów z ostrym przebiegiem COVID-19[164].
Główną przyczyną śmierci u pacjentów w przebiegu COVID-19 jest zespół ostrej niewydolności oddechowej (ARDS). Spośród 41 pacjentów zakażonym wirusem SARS-CoV-2 we wczesnej fazie pandemii w Wuhan 6 zmarło właśnie na skutek ARDS. Jednym z głównych mechanizmów występujących w ARDS jest niekontrolowana systemowa odpowiedź zapalna w postaci burzy cytokin (IFN-α, IFN-γ, IL-1β, IL-6, IL-12, IL-18, IL-33, TNF-α, TGFβ itp.) oraz chemokin (CCL2, CCL3, CCL5, CXCL8, CXCL9, CXCL10), co prowadzi w konsekwencji do niewydolności oddechowej oraz niewydolności wielu organów i śmierci[128].
Przeciwciała SARS-CoV a SARS-CoV-2
Podczas testów na komórkach zwierzęcych (u myszy) badania wykazały, że przeciwciała monoklonalne mAb mogą rozpoznawać fragmenty glikoproteiny S wirusa SARS-CoV-2[165][166]. W środowisku laboratoryjnym zastosowano cztery mysie przeciwciała mAb: 2B2, 1A9, 4B12 i 1G10 na komórkach COS-7. Wyniki pokazały, że wspomniane cztery przeciwciała monoklonalne swoiste wobec SARS mogą reagować krzyżowo także z białkiem S (aa1048-1206) wirusa SARS-CoV-2[165]. Natomiast w osobnym eksperymencie zaobserwowano jedynie umiarkowaną reakcję krzyżową pomiędzy serum z przeciwciałami T62 specyficznymi wobec wirusa SARS, a wirusem SARS-CoV-2, co sugeruje, że przejście infekcji SARS nie chroni w pełni przed infekcją wirusem SARS-CoV-2, i vice versa[135].
Leczenie
Światowa Organizacja Zdrowia wprowadziła tymczasowy termin „ostra choroba układu oddechowego 2019-nCoV” na określenie choroby wywołanej przez wirusa[167]. Nazwa ta później została zmieniona na COVID-19 (choroba koronawirusowa 2019, ang. Coronavirus Disease 2019[168]). Obecnie nie istnieje leczenie przyczynowe, zalecane jest leczenie objawowe[11]. Nie istnieje obecnie żadna przebadana szczepionka przeciwko temu wirusowi.
Poniżej opisano leczenie eksperymentalne oraz leki w trakcie badań.
W celu lepszego monitorowania i porównywania wyników badań najbardziej obiecujących eksperymentalnych leków przeciw wirusowi SARS-CoV-2 i chorobie COVID-19 w wielu krajach Światowa Organizacja Zdrowia wraz z partnerami podjęła w marcu 2020 roku współpracę na globalnym projektem „SOLIDARITY trial”, polegającym na koordynacji badań leków na całym świecie[169][170].
Szczepionka
Dotychczas nie wyprodukowano szczepionki przeciwko wirusowi SARS-CoV-2, chociaż od początku wybuchu pandemii różne ośrodki badawcze intensywnie dążą do jej opracowania. Ponieważ SARS-CoV-2 używa tego samego receptora, ACE2, jak SARS, możliwe jest wykorzystanie wyników wcześniejszych badań nad SARS[80].
W trwających pracach nad szczepionkami próbuje się wytworzyć szczepionki typu[171]:
- Szczepionka inaktywowana, zawierająca inaktywowane lub martwe wirusy – ma na celu wywołanie szybkiej odpowiedzi immunologicznej organizmu ludzkiego na nową infekcję COVID-19.
- Szczepionka podjednostkowa, zawierająca fragmenty wirusa, ma na celu uwrażliwienie układu odpornościowego na określone podjednostki wirusa. W przypadku wirusa SARS-CoV-2, badania skupiają się na glikoproteinie S, która łączy się z receptorem ACE2.
- Szczepionka oparta na kwasach nukleinowych RNA lub DNA, zawierająca zsyntetyzowany odcinek mRNA wirusa, który koduje białko na które reaguje układ immunologiczny[172].
Szczepionki wymagają badań dotyczących bezpieczeństwa i skuteczności ich stosowania[173]. Po uzyskaniu szczepionki proces badania bezpieczeństwa i skuteczności trwa około 18 miesięcy[174].
Zobacz też: Badania nad szczepionką SARS-CoV-2 (ang.).
Leki przeciwwirusowe
Niektóre istniejące środki przeciwwirusowe, które są stosowane na przykład przeciwko MERS-CoV i HIV, mogą być skuteczne przy zakażeniu SARS-CoV-2[175][176][177][178]. Należą do nich inhibitory proteazy, takie jak indynawir, sakwinawir, lopinawir / rytonawir oraz interferon beta i remdesiwir[179][180]. Narodowa Komisja Zdrowia ChRL zaleca łączenie inhibitorów proteazy HIV lopinawiru i rytonawiru oraz rybawiriny z wziewnym interferonem alfa[181]. Badania z wykorzystaniem komórek Vero E6 wykazały, że w porównaniu z wirusem SARS nowy wirus SARS-CoV-2 wykazuje znacznie większą wrażliwość na terapię interferonami typu I, co skutkuje ograniczeniem jego replikacji[182]. W Stanach Zjednoczonych zastosowano, zaliczany do klasy analogów nukleotydów, lek remdesiwir, który został opracowany w leczeniu choroby wirusowej Ebola. Potwierdzono skuteczność remdesiwiru in vitro[183][184]. Obecnie w Stanach Zjednoczonych, Chinach i Włoszech trwa 3 faza badań klinicznych, sprawdzających skuteczność tego leku – publikacja wyników spodziewana jest do końca kwietnia 2020 roku[185][186][187][188][189]. Według doniesień mediów, pierwszy pacjent w Hiszpanii zarażony wirusem SARS-CoV-2 po terapii lopinawirem / rytonawirem powrócił do pełni zdrowia[190][191]. Należy zauważyć, że istnieją wyniki badań na 199 pacjentach w Chinach, które nie wykazały zadowalającej skuteczności lopinawiru/rytonawiru, jednak autorzy podkreślili krytyczny stan większości badanych pacjentów i fakt późnego wdrożenia leczenia[192]. Jak podała Polska Agencja Prasowa 13 marca 2020 roku, polscy pacjenci hospitalizowani w Wojewódzkim Szpitalu Specjalistycznym im. J. Gromkowskiego we Wrocławiu będą mieli możliwość leczenia lekami na HIV i malarię. Ze względu na eksperymentalny charakter leczenia, prof. Krzysztof Simon, ordynator szpitala, poinformował, że uzyskał zgodę komisji bioetycznej, a leki będą podawane za zgodą pacjenta[193].
Chińskie Centrum Kontroli i Prewencji Chorób testuje istniejące metody leczenia zapalenia płuc pod kątem skuteczności w leczeniu zapalenia płuc związanego z SARS-CoV-2[194]. Oprócz remdesiwiru aktualnie przeprowadzane są w Chinach testy fawipirawiru i chlorochiny (leku stosowanego przeciwko malarii) z udziałem ludzi[195][196]. Badania kliniczne remdesiwiru odbywają się w Chinach i innych krajach. Fawipirawir – lek przeciwko wirusowi grypy – jest testowany na 70 pacjentach w Shenzhen, a 16 lutego 2020 roku został dopuszczony w Chinach na 5 lat pod nową nazwą Favilavir jako eksperymentalny lek przeciw COVID-19 i natychmiast wdrożony do produkcji[197]. Chlorochina została przetestowana na ponad 100 pacjentach w szpitalach w Pekinie oraz w prowincji Guangdong i znajduje się od 20 lutego 2020 roku wraz z umifenowirem (nazwa handlowa Arbidol) – dopuszczonym w Rosji i Chinach lekiem przeciwgrypowym – na liście leków zalecanych[198]. W eksperymencie klinicznym 27 lutego 2020 roku została potwierdzona skuteczność chlorochiny[199]. Pierwsze wyniki zostały opublikowane w czasopiśmie BioScienceTrends, gdzie uznano leczenie chlorochiną jako „bardziej skuteczne” niż leczenie z użyciem placebo[200]. Od 13 marca 2020 roku decyzją Urzędu Rejestracji Produktów Leczniczych, Wyrobów Medycznych i Produktów Biobójczych lek z cholorochiną został dopuszczony do leczenia wspomagającego w leczeniu wirusa w Polsce[201][202]. Produkuje go polska firma Adamed pod nazwą Arechin.
W badaniu 138 pacjentów w Chinach zastosowanie środka przeciwwirusowego oseltamiwir nie wykazało znaczących efektów leczniczych[203].
Zobacz też: Badania istniejących leków w kontekście choroby COVID-19 (ang.).
Zapobieganie burzy cytokin
Burza cytokin, zagrażająca życiu reakcja immunologiczna, może być powikłaniem choroby COVID-19, wywoływanej przez SARS-CoV-2.
Istnieją dowody, że hydroksycholorochina posiada właściwości hamujące burzę cytokin[204]. Niektórzy naukowcy proponują jako potencjalną terapię łączenie hydroksychlorochiny z nitazoksanidem w celu zwiększenia skuteczności[205].
Narodowa Komisja Zdrowia ChRL na podstawie badań na małych grupach osób włączyła tocilizumab do wytycznych w zakresie leczenia pacjentów[206][207]. Lek ten jest przeciwciałem monoklonalnym przeciw IL-6. Aktualnie znajduje się w 2 fazie badań klinicznych we Włoszech po wykazaniu pozytywnych efektów u osób z ciężkim przebiegiem choroby[208][209][210]. W Stanach Zjednoczonych lek funkcjonuje pod nazwą Actemra i został dopuszczony przez FDA do leczenia innych chorób[211][212].
Terapia przeciwciałami
Obiecującą potencjalną terapią ciężkich przypadków zakażenia może być leczenie specjalnie spreparowanym osoczem krwi, zawierającym swoiste przeciwciała zwalczające wirusa SARS-CoV-2 pozyskane od ozdrowieńców po COVID-19 (infekcji wywołanej tym gatunkiem koronawirusa)[213][214][215]. Skuteczność tego rodzaju immunizacji została ogłoszona przez chiński koncern National Biotec Group[216][217][218].
Inne
Inhibitory TMPRSS2
Grupa niemieckich naukowców pod przewodnictwem Christiana Drostena przedstawiła w publikacji[138], że enzym TMPRSS2 (seryna 2), wymagany do rozmnażania wirusa, może być skutecznie hamowany za pomocą leku stosowanego w leczeniu przewlekłego zapalenia trzustki. Lek Camostat jest dopuszczony w Japonii i może zostać poddany bezpośrednio eksperymentom klinicznym.
Innym inhibitorem TMPRSS2 jest dostępna powszechnie bromheksyna[219][205], która jest poddawana klinicznej ocenie skuteczności leczenia u pacjentów z chorobą COVID-19[220].
Proteina Ly6e
Interesujące pod kątem potencjalnego leczenia SARS-CoV-2 są odkrycia naukowców pod przewodnictwem Johna Schogginsa z University of Texas Southwestern, które następnie zostały podjęte i kontynuowane przez grupę naukowców pod przewodnictwem Stephanie Pfaender ze Szwajcarii[221]. Grupa Johna Schogginsa odkryła, że ludzkie białko Ly6e zwiększa wirulencję (wnikanie i mnożenie) niektórych wirusów osłonkowych RNA, takich jak HIV-1, wirusa Gorączki Zachodniego Nilu, wirusa dengi czy wirusa Zika. Paradoksalnie, podczas testów na myszach naukowcy zauważyli, że po usunięciu genu Ly6e zwierzęta stały się niezwykle podatne na infekcje różnego rodzaju koronawirusami, co sugerowało odwrotny wpływ białka Ly6e na ten rodzaj wirusów[222]. Potwierdziły to badania grupy Stephanie Phaender, która wykazała in vitro oraz in vivo na myszach, że Ly6e silnie powstrzymuje infekcje wywołane różnymi typami koronawirusów, takimi jak HCoV-229E, MERS-CoV, SARS-CoV czy SARS-CoV-2, jednocześnie wywołując odwrotny efekt (tj. sprzyjając infekcji) podczas badania wirusów grypy typu A, flawiwirusów i HIV-1. W szczególności Ly6e powstrzymywała mediowaną przez glikoproteinę S fuzję osłonki wirusa z komórkami gospodarza. Wykazano również, że Ly6e bezpośrednio chroni limfocyty B i komórki dendrytyczne przed infekcją CoV u myszy. Podejście terapeutyczne naśladujące mechanizm działania Ly6e może zapewnić pierwszą linię obrony przed infekcjami koronawirusowymi[223].
Inhibitory proteazy Mpro
Podstawą do poszukiwania leku przeciw wirusowi SARS-CoV-2 są badania grupy naukowców, w skład której wchodzi Marcin Drąg z Politechniki Wrocławskiej. Badacze, opierając się na doświadczeniach z wirusem SARS, zidentyfikowali enzym Mpro (lub 3CLpro, czyli główną proteazę), który jest kluczowy w procesie cięcia białek wirusa i jego replikacji. Autorzy sugerują, że zahamowanie tego enzymu jest kluczową formą terapii przeciwwirusowej[224]. Podobne stanowisko zajął zespół naukowców z Niemiec, którzy w późniejszej publikacji potwierdzają znaczenie proteazy Mpro w leczeniu[225]. Obydwie grupy badaczy podkreślają, że ponieważ proteaza ta nie występuje w organizmie człowieka, potencjalny lek będący jej inhibitorem cechowałby się brakiem skutków ubocznych i małą toksycznością dla człowieka[224][225]. Jak poinformowały media, badacze z Politechniki Śląskiej, University of Alberta z Kanady i Politechniki w Turynie utworzyły międzynarodowy zespół Tunnelling Group, badający enzym Mpro i potencjalne leki[226][227]. Zespół naukowców z Chińskiej Akademii Nauk wykorzystując metody teoretyczne 3D, bazujące na integracji technik modelowania homologicznego struktury Mpro, dokowania molekularnego cząsteczek wybranych leków i obliczeniach energii wiązania wskazał nelfinavir jako potencjalny inhibitor głównej proteazy (Mpro) wirusa SARS-CoV-2[228].
Procedura triażu i leczenia pacjentów w Wuhan
W centrum medycznej segregacji w Wuhan schemat diagnostyki i leczenia pacjentów z podejrzeniem zakażenia zakłada stosowanie przeciwwirusowego umifenowiru i antybiotykoterapię linezolidem, nemonoksacyną lub fluorochinolonami. Leczenie antybiotykami jest uzasadnione oczekiwaniem wtórnego zakażenia bakteryjnego tkanki płucnej uszkodzonej przez infekcję wirusową. Autorzy podkreślają znaczenie rozpoczęcia terapii przed potwierdzeniem diagnozy za pomoca testu RT-PCR, na podstawie wyników badań przedmiotowych, laboratoryjnych i diagnostycznych. Pacjenci bez obniżonej liczby limfocytow we krwi, bez wirusowego zapalenia płuc, bez duszności i bez zmniejszenia saturacji krwi tlenem poniżej 93% powinni zostać wypisani do opieki domowej bez badania RT-PCR. Dla tych pacjentów przeznaczona jest doustna azytromycyna lub amoksycylina. Pacjenci z zapaleniem płuc o innej niż CoV-2 patogenezie powinni być leczeni systematycznie i, jeśli to możliwe, w warunkach ambulatoryjnych. Kobiety w ciąży i osoby powyżej 65 roku życia powinny być traktowane podczas leczenia hospitalizacyjnego jako grupy szczególnego ryzyka. Autorzy badania uzasadniają swoje podejście przeciążeniem systemu opieki zdrowotnej w Wuhan. Leczenie jak największej liczby pacjentów w domach wiąże się z ryzykiem, ale jest konieczne w celu równoczesnego leczenia wielu przypadków krytycznych[229].
Profilaktyka
Zalecenia profilaktyczne:
- Często myć ręce, ciepłą wodą i mydłem, szczególnie przed jedzeniem, po wydmuchaniu nosa, kaszlu lub kichaniu, przez co najmniej 30 sekund, i po wydmuchaniu nosa, kaszlu lub kichaniu szczególnie po kontakcie z osobami chorującymi lub ich otoczeniem. Kran zakręcić przez chusteczkę. Gdy mydło i woda nie są łatwo dostępne, należy dezynfekować ręce z co najmniej 60% objętościowym alkoholem.
- Unikać dotykania nosa, oczu, lub ust nieumytymi rękami.
- Unikać bliskiego kontaktu z chorymi wykazującymi choroby układu oddechowego.
- Wystarczająco długo gotować mięso, ryby i owoce morza.
- Zabezpieczać się w czasie kontaktu ze zwierzętami hodowlanymi oraz zwierzętami dzikimi.
- Zasłaniać nos i usta chusteczką higieniczną podczas kichania lub kasłania.
- W placówkach opieki zdrowotnej należy wzmocnić standardowe praktyki zapobiegania i kontroli zakażeń w szpitalach, zwłaszcza na oddziałach ratunkowych[230][231][232][233][234]
- Nosić sprzęt indywidualnej ochrony układu oddechowego (na przykład półmaski filtrujące) chroniące usta i nos[235].
Jednym ze środków prewencyjnych jest kwarantanna osób zakażonych wirusem, mająca na celu ograniczenia rozprzestrzeniania się wirusa[236].

Pojawienie się wirusa zwróciło uwagę na wirusy odzwierzęce[237]. Przeniesieniu się wirusa sprzyjają takie obecne w Chinach czynniki, jak wzrost liczebności populacji, wylesianie, urbanizacja, zmiana klimatu, aspekty związane z produkcją żywności i trzymaniem zwierząt, a w końcu mutacja. Po poprzednich epidemiach zoonoz wirusowych rząd Chin podjął działania przeciwdziałające powtórzeniu się sytuacji[238]. Li i współpracownicy zwracają uwagę na ograniczony charakter dotychczas podjętych działań mających zapobiec przenoszeniu się chorób wirusowych z innych gatunków zwierząt na człowieka[237].
Epidemiologia
Ze względu na dużą epidemiologię nowego wirusa 11 marca 2020 roku Światowa Organizacja Zdrowia sklasyfikowała rozprzestrzenianie się wirusa SARS-CoV-2 jako pandemię[239].
Dezinformacja
Według szeregu źródeł od stycznia 2020 prowadzona jest operacja dezinformacyjna wykorzystująca koronawirus SARS-CoV-2. Prowadzona jest w internecie na platformach społecznościowych oraz przez prokremlowskie media. W założeniach jest identyczna do operacji dezinformacyjnej z lat 80. XX wieku prowadzonej przez ZSRR (KGB) – Operation INFEKTION – w której m.in. przedstawiano ludzki wirus niedoboru odporności (HIV) jako rzekomą „amerykańską broń biologiczną” (zob. „operation infektion” w dezinformacja: historia)[240][241][242][243][244][245][246][247].
3 marca 2020 potwierdzono, że „system szybkiego ostrzegania i monitorowania poważnych przypadków dezinformacji” Unii Europejskiej został zastosowany celem przeciwdziałania dezinformacji wykorzystującej koronawirus SARS-CoV-2, chorobę COVID-19 i szerzenie się zakażeń wirusem SARS-CoV-2[248]. Wiceprzewodnicząca Komisji Europejskiej Věra Jourová poinformowała, że narzędzie to zostało użyte do wymiany wiedzy między państwami członkowskimi UE, a także partnerami G7 na temat dezinformacji „pochodzącej ze źródeł zewnętrznych”[248]. Dzień wcześniej doszło do spotkania w którym uczestniczyli przedstawiciele m.in. Google, Facebook, Twitter i Microsoft, aby omówić sposoby ograniczenia przepływu dezinformacji online dotyczącej koronawirusa[248]. Według oświadczenia Jourovej: „Wszyscy uczestnicy spotkania potwierdzili, że wykryli różne rodzaje dezinformacji lub fake news w Internecie i podjęli szereg działań w celu przeciwdziałania. Przykłady dezinformacji obejmują fałszywe środki zaradcze, które mają rzekomo na celu zaradzenie zagrożeniu i mogą stanowić zagrożenie dla zdrowia (np. picie wybielacza w celu zabicia wirusa lub wkładanie octu do nozdrzy, aby uniknąć zarażenia) lub mistyfikację na temat pochodzenia wirusa oraz sposobu i prędkości, z jaką się rozprzestrzenia”[248].
9 marca 2020 brytyjski rząd utworzył jednostkę ds. przeciwdziałania dezinformacji, z uwagi na działalność grupy państw „wykorzystujących kryzys koronawirusa”[249][250]. Eksperci mają m.in. współpracować z firmami prowadzącymi media społecznościowe w celu ochrony Wielkiej Brytanii przed fałszywymi informacjami[249][250]. Brytyjska narodowa służba zdrowia (NHS) współpracuje m.in. z Twitterem w celu zawieszania fałszywych kont[251]. Niektóre z tych kont np. udają profile szpitali i usiłują wprowadzać niedokładne informacje na temat liczby przypadków zakażeń[251].
Według szefa sztabu obrony Kanady gen. Jonathana Vance występują oznaki internetowych działań dezinformacyjnych zmierzających do zasiania paniki z powodu COVID-19[251]. Będą również podejmowane wysiłki ze strony aktorów państwowych i niepaństwowych (przestrzeni informacyjnej), aby dyskredytować kroki państw podejmowane w związku z zakażeniami wirusem SARS-CoV-2[251].
Zobacz też
- koronawirusy
- pandemia COVID-19
- pandemia COVID-19 w Polsce
- wirus SARS – koronawirus odpowiedzialny za pandemię z lat 2002–2003
Uwagi
- ↑ Przed 11 lutego 2020 stosowana była nazwa tymczasowa: wirus 2019-nCoV.
Przypisy
- ↑ Alexander E. Gorbalenya, Severe acute respiratory syndrome-related coronavirus – The species and its viruses, a statement of the Coronavirus Study Group, „bioRxiv”, 2020, DOI: 10.1101/2020.02.07.937862 (ang.).
- ↑ Alexander E. Gorbalenya i inni, The species Severe acute respiratory syndrome-related coronavirus. Classifying 2019-nCoV and naming it SARS-CoV-2, „Nature Microbiology”, 2020, s. 1–9, DOI: 10.1038/s41564-020-0695-z, ISSN 2058-5276 [dostęp 2020-03-20].
- ↑ Charles Patrick Davis, Medical Definition of Wuhan Coronavirus [online], MedicineNet, 22 stycznia 2020 [dostęp 2020-01-24] (ang.).
- ↑ a b c d e f Na Zhu i inni, A Novel Coronavirus from Patients with Pneumonia in China, 2019, „The New England Journal of Medicine”, 2020, DOI: 10.1056/NEJMoa2001017, PMID: 31978945 (ang.).
- ↑ New-type coronavirus causes pneumonia in Wuhan: expert [online], Xinhuanet [dostęp 2020-01-23].
- ↑ 中国疾病预防控制中心 [online], Chinese Center for Disease Control and Prevention [dostęp 2020-01-23].
- ↑ Coronavirus: China’s first confirmed Covid-19 case traced back to November 17 [online], scmp.com, 13 marca 2020 [dostęp 2020-03-21] (ang.).
- ↑ The first COVID-19 case originated on November 17, according to Chinese officials searching for 'patient zero' [online], businessinsider.com, 13 marca 2020 [dostęp 2020-03-21] (ang.).
- ↑ Jon Cohen, Dennis Normile, New SARS-like virus in China triggers alarm, „Science”, 367 (6475), 2020, s. 234–235, DOI: 10.1126/science.367.6475.234, PMID: 31949058 (ang.).
- ↑ Jane Parry, China coronavirus: cases surge as official admits human to human transmission, „British Medical Journal”, 368, 2020, DOI: 10.1136/bmj.m236, PMID: 31959587 (ang.).
- ↑ a b 2019 Novel Coronavirus (2019-nCoV), Wuhan, China [online], Centers for Disease Control and Prevention, 28 stycznia 2020 [dostęp 2020-01-29] (ang.).
- ↑ Grace Ren, Mystery Virus in Wuhan Identified As Novel Coronavirus; Researchers Still Searching For Animal Host [online], Health Policy Watch, 13 stycznia 2020 (ang.).
- ↑ Ewen Callaway, David Cyranoski, Why snakes probably aren’t spreading the new China virus, „Nature”, 2020, DOI: 10.1038/d41586-020-00180-8 (ang.).
- ↑ a b c Informacja dot. przypadków zachorowań na zapalenie płuc wywołane nowym typem koronawirusa 2019-nCoV w Chinach. [online], Wojewódzka Stacja Sanitarno-Epidemiologiczna w Warszawie, 22 stycznia 2020 [dostęp 2020-01-27].
- ↑ a b 新型冠状病毒问答 [online], New South Wales Ministry of Health [dostęp 2020-01-27] (chiń.).
- ↑ https://www.scmp.com/news/china/society/article/3074991/coronavirus-chinas-first-confirmed-covid-19-case-traced-back, dostęp 2020-03-21, 17.23.
- ↑ a b c d David S. Hui i inni, The continuing 2019-nCoV epidemic threat of novel coronaviruses to global health – The latest 2019 novel coronavirus outbreak in Wuhan, China, „International Journal of Infectious Diseases”, 91, 2020, s. 264–266, DOI: 10.1016/j.ijid.2020.01.009, PMID: 31953166 (ang.).
- ↑ Novel Coronavirus (2019-nCoV) [online], World Health Organization [dostęp 2020-01-26] (ang.).
- ↑ Pochodzenia wirusa... analiza danych, wyniki ciekawe [online], openin.pl [dostęp 2020-02-13].
- ↑ a b Weilong Shang i inni, The outbreak of SARS-CoV-2 pneumonia calls for viral vaccines, „npj Vaccines”, 5 (1), 2020, s. 1–3, DOI: 10.1038/s41541-020-0170-0, ISSN 2059-0105 [dostęp 2020-03-13] (ang.).
- ↑ a b txid2697049 – Nucleotide – NCBI [online], www.ncbi.nlm.nih.gov [dostęp 2020-03-13] (ang.).
- ↑ Severe acute respiratory syndrome coronavirus 2 isolate Wuhan-Hu-1, complete genome [online], 11 lutego 2020 [dostęp 2020-03-13] (ang.).
- ↑ a b Krzysztof Pyrć, Ludzkie koronawirusy [PDF], „Postępy Nauk Medycznych”.
- ↑ Naina Barretto i inni, The Papain-Like Protease of Severe Acute Respiratory Syndrome Coronavirus Has Deubiquitinating Activity, „Journal of Virology”, 79 (24), 2005, s. 15189–15198, DOI: 10.1128/JVI.79.24.15189-15198.2005, ISSN 0022-538X, PMID: 16306590 [dostęp 2020-03-24] (ang.).
- ↑ Proteins of Coronavirus SARS-CoV-2 and related drugs in HOMCOS [online].
- ↑ Stanley Perlman, Jason Netland, Coronaviruses post-SARS: update on replication and pathogenesis, „Nature Reviews Microbiology”, 7 (6), 2009, s. 439–450, DOI: 10.1038/nrmicro2147, ISSN 1740-1526 [dostęp 2020-03-24] (ang.).
- ↑ a b Canrong Wu i inni, Analysis of therapeutic targets for SARS-CoV-2 and discovery of potential drugs by computational methods, „Acta Pharmaceutica Sinica B”, 2020, S2211383520302999, DOI: 10.1016/j.apsb.2020.02.008 [dostęp 2020-03-13] (ang.).
- ↑ a b c Roujian Lu i inni, Genomic characterisation and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding, „The Lancet”, 395 (10224), 2020, s. 565–574, DOI: 10.1016/S0140-6736(20)30251-8 [dostęp 2020-03-12] (ang.).
- ↑ a b c d Alexandra C. Walls i inni, Structure, Function, and Antigenicity of the SARS-CoV-2 Spike Glycoprotein, „Cell”, 2020, DOI: 10.1016/j.cell.2020.02.058, ISSN 0092-8674 [dostęp 2020-03-30].
- ↑ David E. Gordon i inni, A SARS-CoV-2-Human Protein-Protein Interaction Map Reveals Drug Targets and Potential Drug-Repurposing, „bioRxiv”, 2020, 2020.03.22.002386, DOI: 10.1101/2020.03.22.002386 [dostęp 2020-03-30] (ang.).
- ↑ Coronavirus [online], World Health Organization [dostęp 2020-01-23] (ang.).
- ↑ Phylogeny of SARS-like betacoronaviruses including novel coronavirus from Wuhan using data generated by the Shanghai Public Health Clinical Center & School of Public Health, the National Institute for Viral Disease Control and Prevention, the Institute of Pathogen Biology, and the Wuhan Institute of Virology shared via GISAID [online], Nextstrain [dostęp 2020-01-23] (ang.).
- ↑ Novel 2019 coronavirus genome [online], Virological, 11 stycznia 2020 [dostęp 2020-01-23] (ang.).
- ↑ National laboratories [online], www.who.int [dostęp 2020-03-30] (ang.).
- ↑ Laboratory biosafety guidance related to coronavirus disease 2019 (COVID-19) [online], www.who.int [dostęp 2020-03-30] (ang.).
- ↑ a b c d e f Yan-Rong Guo i inni, The origin, transmission and clinical therapies on coronavirus disease 2019 (COVID-19) outbreak – an update on the status, „Military Medical Research”, 7 (1), 2020, s. 11, DOI: 10.1186/s40779-020-00240-0, ISSN 2054-9369, PMID: 32169119, PMCID: PMC7068984 [dostęp 2020-03-26].
- ↑ Jun Chen i inni, Clinical progression of patients with COVID-19 in Shanghai, China, „Journal of Infection”, 2020, DOI: 10.1016/j.jinf.2020.03.004, ISSN 0163-4453 [dostęp 2020-03-26] (ang.).
- ↑ RKI - Coronavirus SARS-CoV-2 – Antworten auf häufig gestellte Fragen zum Coronavirus SARS-CoV-2 [online], www.rki.de [dostęp 2020-03-26].
- ↑ Jasper Fuk-Woo Chan i inni, A familial cluster of pneumonia associated with the 2019 novel coronavirus indicating person-to-person transmission: a study of a family cluster, „The Lancet”, 395 (10223), 2020, s. 514–523, DOI: 10.1016/S0140-6736(20)30154-9 [dostęp 2020-03-26] (ang.).
- ↑ CDC, Coronavirus Disease 2019 (COVID-19) – Symptoms [online], Centers for Disease Control and Prevention, 20 marca 2020 [dostęp 2020-03-26] (ang.).
- ↑ Nanshan Chen i inni, Epidemiological and clinical characteristics of 99 cases of 2019 novel coronavirus pneumonia in Wuhan, China: a descriptive study, „The Lancet”, 395 (10223), 2020, s. 507–513, DOI: 10.1016/S0140-6736(20)30211-7 [dostęp 2020-03-26] (ang.).
- ↑ Chaolin Huang i inni, Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China, „The Lancet”, 395 (10223), 2020, s. 497–506, DOI: 10.1016/S0140-6736(20)30183-5 [dostęp 2020-03-26] (ang.).
- ↑ Chih-Cheng Lai i inni, Severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) and coronavirus disease-2019 (COVID-19): The epidemic and the challenges, „International Journal of Antimicrobial Agents”, 55 (3), 2020, s. 105924, DOI: 10.1016/j.ijantimicag.2020.105924 [dostęp 2020-03-26] (ang.).
- ↑ Ying-Ying Zheng i inni, COVID-19 and the cardiovascular system, „Nature Reviews Cardiology”, 2020, s. 1–2, DOI: 10.1038/s41569-020-0360-5, ISSN 1759-5010 [dostęp 2020-03-26] (ang.).
- ↑ David S. Hui i inni, The continuing 2019-nCoV epidemic threat of novel coronaviruses to global health – The latest 2019 novel coronavirus outbreak in Wuhan, China, „International Journal of Infectious Diseases”, 91, 2020, s. 264–266, DOI: 10.1016/j.ijid.2020.01.009 [dostęp 2020-03-26] (ang.).
- ↑ Ling Mao i inni, Neurological Manifestations of Hospitalized Patients with COVID-19 in Wuhan, China: a retrospective case series study, 25 lutego 2020, DOI: 10.1101/2020.02.22.20026500 [dostęp 2020-03-26] (ang.).
- ↑ Zi Yue Zu i inni, Coronavirus Disease 2019 (COVID-19): A Perspective from China, „Radiology”, 2020, s. 200490, DOI: 10.1148/radiol.2020200490, ISSN 0033-8419 [dostęp 2020-03-26] (ang.).
- ↑ Yang Liu i inni, Viral dynamics in mild and severe cases of COVID-19, „The Lancet Infectious Diseases”, 2020, S1473309920302322, DOI: 10.1016/S1473-3099(20)30232-2 [dostęp 2020-03-26] (ang.).
- ↑ WHO, Clinical management of severe acute respiratory infection when novel coronavirus (2019-nCoV) infection is suspected [PDF].
- ↑ Yan-Chao Li, Wan-Zhu Bai, Tsutomu Hashikawa, The neuroinvasive potential of SARS-CoV2 may play a role in the respiratory failure of COVID-19 patients, „Journal of Medical Virology”, 2020, DOI: 10.1002/jmv.25728, ISSN 1096-9071, PMID: 32104915 [dostęp 2020-03-26].
- ↑ Dandan Wu, Xuexian O. Yang, TH17 responses in cytokine storm of COVID-19: An emerging target of JAK2 inhibitor Fedratinib, „Journal of Microbiology, Immunology and Infection”, 2020, S1684118220300657, DOI: 10.1016/j.jmii.2020.03.005 [dostęp 2020-03-26] (ang.).
- ↑ Didier Raoult i inni, Coronavirus infections: Epidemiological, clinical and immunological features and hypotheses, „Cell Stress”, 2020, DOI: 10.15698/cst2020.04.216, ISSN 2523-0204, PMCID: PMC7064018 [dostęp 2020-03-26].
- ↑ RKI - Coronavirus SARS-CoV-2 – SARS-CoV-2 Steckbrief zur Coronavirus-Krankheit-2019 (COVID-19) [online], www.rki.de [dostęp 2020-03-26].
- ↑ Nicky Phillips, Smriti Mallapaty, David Cyranoski, How quickly does the Wuhan virus spread?, „Nature”, 2020, d41586–020–00146-w, DOI: 10.1038/d41586-020-00146-w, ISSN 0028-0836 [dostęp 2020-03-11] (ang.).
- ↑ Zhangkai J. Cheng, Jing Shan, 2019 Novel coronavirus: where we are and what we know, „Infection”, 2020, DOI: 10.1007/s15010-020-01401-y, ISSN 0300-8126 [dostęp 2020-03-11] (ang.).
- ↑ a b c CDC CDC, Coronavirus Disease 2019 (COVID-19) – Transmission [online], Centers for Disease Control and Prevention, 4 marca 2020 [dostęp 2020-03-11] (ang.).
- ↑ a b RKI - Coronavirus SARS-CoV-2 – Antworten auf häufig gestellte Fragen zum Coronavirus SARS-CoV-2 [online], www.rki.de [dostęp 2020-03-11].
- ↑ Beschluss 1/2020 des ABAS vom 19.2.2020 und Begründung zur vorläufigen Einstufung des Virus SARS-CoV-2 in Risikogruppe 3 und Empfehlungen zu nicht gezielten Tätigkeiten (Labordiagnostik) und gezielten Tätigkeiten mit SARS-CoV-2. [pdf].
- ↑ Jasper Fuk-Woo Chan i inni, A familial cluster of pneumonia associated with the 2019 novel coronavirus indicating person-to-person transmission: a study of a family cluster, „The Lancet”, 395 (10223), 2020, s. 514–523, DOI: 10.1016/S0140-6736(20)30154-9 [dostęp 2020-03-11] (ang.).
- ↑ Michelle L. Holshue i inni, First Case of 2019 Novel Coronavirus in the United States, „The New England Journal of Medicine”, 382 (10), 2020, s. 929–936, DOI: 10.1056/NEJMoa2001191, ISSN 0028-4793 [dostęp 2020-03-11] (ang.).
- ↑ Jianhua Xia i inni, Evaluation of coronavirus in tears and conjunctival secretions of patients with SARS-CoV-2 infection, „Journal of Medical Virology”, 2020, jmv.25725, DOI: 10.1002/jmv.25725, ISSN 0146-6615 [dostęp 2020-03-11] (ang.).
- ↑ Yu Chen, Lanjuan Li, SARS-CoV-2: virus dynamics and host response, „The Lancet Infectious Diseases”, 2020, S1473309920302358, DOI: 10.1016/S1473-3099(20)30235-8 [dostęp 2020-03-26] (ang.).
- ↑ Charleen Yeo, Sanghvi Kaushal, Danson Yeo, Enteric involvement of coronaviruses: is faecal–oral transmission of SARS-CoV-2 possible?, „The Lancet Gastroenterology & Hepatology”, 2020, S2468125320300480, DOI: 10.1016/S2468-1253(20)30048-0 [dostęp 2020-03-11] (ang.).
- ↑ Sean Wei Xiang Ong i inni, Air, Surface Environmental, and Personal Protective Equipment Contamination by Severe Acute Respiratory Syndrome Coronavirus 2 (SARS-CoV-2) From a Symptomatic Patient, „Journal of the American Medical Association”, 2020, DOI: 10.1001/jama.2020.3227, ISSN 0098-7484 [dostęp 2020-03-14] (ang.).
- ↑ Yongjian Wu i inni, Prolonged presence of SARS-CoV-2 viral RNA in faecal samples, „The Lancet Gastroenterology & Hepatology”, 5 (5), 2020, s. 434–435, DOI: 10.1016/s2468-1253(20)30083-2, ISSN 2468-1253 [dostęp 2020-04-09].
- ↑ a b Lirong Zou i inni, SARS-CoV-2 Viral Load in Upper Respiratory Specimens of Infected Patients, „The New England Journal of Medicine”, 2020, NEJMc2001737, DOI: 10.1056/NEJMc2001737, ISSN 0028-4793 [dostęp 2020-03-11] (ang.).
- ↑ Neuartiges Virus SARS-CoV-2 (bislang 2019-nCoV) durch den ABAS in Risikogruppe 3 eingestuft und Empfehlungen zur Labordiagnostik [online], BAuA: Federal Institute for Occupational Safety and Health.
- ↑ Roman Woelfel i inni, Clinical presentation and virological assessment of hospitalized cases of coronavirus disease 2019 in a travel-associated transmission cluster, 8 marca 2020, DOI: 10.1101/2020.03.05.20030502 [dostęp 2020-03-11] (ang.).
- ↑ a b c d B. Coutard i inni, The spike glycoprotein of the new coronavirus 2019-nCoV contains a furin-like cleavage site absent in CoV of the same clade, „Antiviral Research”, 176, 2020, s. 104742, DOI: 10.1016/j.antiviral.2020.104742 [dostęp 2020-03-11] (ang.).
- ↑ Z. Galen Wo, Sequence Analysis Indicates that 2019-nCoV Virus Contains a Putative Furin Cleavage Site at the Boundary of S1 and S2 Domains of Spike Protein, 18 lutego 2020, DOI: 10.31219/osf.io/nkcrf [dostęp 2020-03-11].
- ↑ Roman Wölfel i inni, Virological assessment of hospitalized patients with COVID-2019, „Nature”, 2020, s. 1–10, DOI: 10.1038/s41586-020-2196-x, ISSN 1476-4687 [dostęp 2020-04-09] (ang.).
- ↑ G. Kampf i inni, Persistence of coronaviruses on inanimate surfaces and their inactivation with biocidal agents, „Journal of Hospital Infection”, 104 (3), 2020, s. 246–251, DOI: 10.1016/j.jhin.2020.01.022 [dostęp 2020-03-11] (ang.).
- ↑ Wie lang Coronaviren auf Flächen überleben und wie man sie inaktiviert [online], idw-online.de [dostęp 2020-03-11] (niem.).
- ↑ Alex Chin i inni, Stability of SARS-CoV-2 in different environmental conditions, „medRxiv”, 2020, 2020.03.15.20036673, DOI: 10.1101/2020.03.15.20036673 [dostęp 2020-03-28] (ang.).
- ↑ Ed Yong, Why the Coronavirus Has Been So Successful [online], The Atlantic, 20 marca 2020 [dostęp 2020-03-21] (ang.).
- ↑ WHO potwierdza: w wodzie koronawirusa nie ma [online], Inżynieria.com [dostęp 2020-03-21].
- ↑ Annika Kratzel i inni, Efficient inactivation of SARS-CoV-2 by WHO-recommended hand rub formulations and alcohols, „bioRxiv”, 2020, 2020.03.10.986711, DOI: 10.1101/2020.03.10.986711 [dostęp 2020-03-21] (ang.).
- ↑ Huijun Chen i inni, Clinical characteristics and intrauterine vertical transmission potential of COVID-19 infection in nine pregnant women: a retrospective review of medical records, „The Lancet”, 395 (10226), 2020, s. 809–815, DOI: 10.1016/S0140-6736(20)30360-3 [dostęp 2020-03-11] (ang.).
- ↑ Min Wei i inni, Novel Coronavirus Infection in Hospitalized Infants Under 1 Year of Age in China, „Journal of the American Medical Association”, 2020, DOI: 10.1001/jama.2020.2131, ISSN 0098-7484, PMID: 32058570, PMCID: PMC7042807 [dostęp 2020-03-11] (ang.).
- ↑ a b c d e f Marco Cascella i inni, Features, Evaluation and Treatment Coronavirus (COVID-19), „StatPearls”, Treasure Island (FL): StatPearls Publishing, 2020, PMID: 32150360 [dostęp 2020-04-06].
- ↑ Myth busters [online], www.who.int [dostęp 2020-03-11] (ang.).
- ↑ Detection of low level of COVID-19 virus in pet dog [online], www.info.gov.hk [dostęp 2020-03-11].
- ↑ COVID-19 a praca lekarza dentysty [online], www.termedia.pl [dostęp 2020-03-29].
- ↑ a b Ying Liu i inni, The reproductive number of COVID-19 is higher compared to SARS coronavirus, „Journal of Travel Medicine”, 27 (2), 2020, DOI: 10.1093/jtm/taaa021, ISSN 1195-1982 [dostęp 2020-03-26] (ang.).
- ↑ a b Ewen Callaway i inni, The coronavirus pandemic in five powerful charts, „Nature”, 579, 2020, s. 482–483, DOI: 10.1038/d41586-020-00758-2 [dostęp 2020-03-26] (ang.).
- ↑ a b WHO, Coronavirus (COVID-19) – selected bibliographic references 18-02-2020 [PDF].
- ↑ a b Chih-Cheng Lai i inni, Severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) and coronavirus disease-2019 (COVID-19): The epidemic and the challenges, „International Journal of Antimicrobial Agents”, 55 (3), 2020, s. 105924, DOI: 10.1016/j.ijantimicag.2020.105924, ISSN 0924-8579 [dostęp 2020-03-26] (ang.).
- ↑ a b Ed Yong, The Deceptively Simple Number Sparking Coronavirus Fears [online], The Atlantic, 28 stycznia 2020 [dostęp 2020-03-26] (ang.).
- ↑ a b Shi Zhao i inni, Preliminary estimation of the basic reproduction number of novel coronavirus (2019-nCoV) in China, from 2019 to 2020: A data-driven analysis in the early phase of the outbreak, „International Journal of Infectious Diseases”, 92, 2020, s. 214–217, DOI: 10.1016/j.ijid.2020.01.050, ISSN 1201-9712 [dostęp 2020-03-26] (ang.).
- ↑ a b Jonathan M. Read i inni, Novel coronavirus 2019-nCoV: early estimation of epidemiological parameters and epidemic predictions, „medRxiv”, 2020, 2020.01.23.20018549, DOI: 10.1101/2020.01.23.20018549 [dostęp 2020-03-26] (ang.).
- ↑ a b COVID-19 reports [online], Imperial College London [dostęp 2020-03-26] (ang.).
- ↑ a b Biao Tang i inni, Estimation of the Transmission Risk of 2019-nCov and Its Implication for Public Health Interventions (2019-nCov的传播风险估计及其对公共卫生干预的意义), Rochester, NY, 24 stycznia 2020 [dostęp 2020-03-26] (ang.).
- ↑ Novel coronavirus disease 2019 (COVID-19) pandemic: increased transmission in the EU/EEA and the UK – sixth update [PDF].
- ↑ Adam J Kucharski i inni, Early dynamics of transmission and control of COVID-19: a mathematical modelling study, „The Lancet Infectious Diseases”, 2020, S1473309920301444, DOI: 10.1016/S1473-3099(20)30144-4 [dostęp 2020-03-26] (ang.).
- ↑ a b Zhangkai J. Cheng, Jing Shan, 2019 Novel coronavirus: where we are and what we know, „Infection”, 2020, DOI: 10.1007/s15010-020-01401-y, ISSN 0300-8126 [dostęp 2020-03-26] (ang.).
- ↑ a b c d RKI - Coronavirus SARS-CoV-2 – SARS-CoV-2 Steckbrief zur Coronavirus-Krankheit-2019 (COVID-19) [online], www.rki.de [dostęp 2020-03-26].
- ↑ Why Some COVID-19 Cases Are Worse than Others [online], The Scientist Magazine® [dostęp 2020-03-26] (ang.).
- ↑ CDCMMWR, Severe Outcomes Among Patients with Coronavirus Disease 2019 (COVID-19) – United States, February 12–March 16, 2020, „MMWR. Morbidity and Mortality Weekly Report”, 69, 2020, DOI: 10.15585/mmwr.mm6912e2, ISSN 0149-2195 [dostęp 2020-03-26] (ang.).
- ↑ a b Sorveglianza integrata COVID-19: i principali dati nazionali [online].
- ↑ a b The Novel Coronavirus Pneumonia Emergency Response Epidemiology Team, The Epidemiological Characteristics of an Outbreak of 2019 Novel Coronavirus Diseases (COVID-19) – China, 2020, „China CDC Weekly”, 2 (8), 1 lutego 2020, s. 113–122, ISSN 2096-7071 [dostęp 2020-03-26] (ang.).
- ↑ a b Ministerio de Sanidad, Consumo y Bienestar Social - Profesionales - Situación actual Coronavirus [online], www.mscbs.gob.es [dostęp 2020-03-26].
- ↑ Yu Chen, Lanjuan Li, SARS-CoV-2: virus dynamics and host response, „The Lancet Infectious Diseases”, 2020, S1473309920302358, DOI: 10.1016/S1473-3099(20)30235-8 [dostęp 2020-03-28] (ang.).
- ↑ Yufeng Cheng i inni, ABO Blood Group and Susceptibility to Severe Acute Respiratory Syndrome, „Journal of the American Medical Association”, 293 (12), 2005, s. 1447–1451, DOI: 10.1001/jama.293.12.1450-c, ISSN 0098-7484 [dostęp 2020-03-21] (ang.).
- ↑ Jiao Zhao i inni, Relationship between the ABO Blood Group and the COVID-19 Susceptibility, „medRxiv”, 2020, 2020.03.11.20031096, DOI: 10.1101/2020.03.11.20031096 [dostęp 2020-03-21] (ang.).
- ↑ Thomas R. Frieden, Christopher T. Lee, Early Release - Identifying and Interrupting Superspreading Events–Implications for Control of Severe Acute Respiratory Syndrome Coronavirus 2 - Volume 26, Number 6–June 2020 - Emerging Infectious Diseases journal – CDC, DOI: 10.3201/eid2606.200495 [dostęp 2020-03-26] (ang.).
- ↑ WHO Director-General’s opening remarks at the media briefing on COVID-19 – 3 March 2020 [online], www.who.int [dostęp 2020-03-26] (ang.).
- ↑ Coronavirus Age, Sex, Demographics (COVID-19) – Worldometer [online], www.worldometers.info [dostęp 2020-03-26] (ang.).
- ↑ Qing Cao i inni, SARS-CoV-2 infection in children: Transmission dynamics and clinical characteristics, „Journal of the Formosan Medical Association = Taiwan Yi Zhi”, 119 (3), 2020, s. 670–673, DOI: 10.1016/j.jfma.2020.02.009, ISSN 0929-6646, PMID: 32139299 [dostęp 2020-03-30].
- ↑ Public Health Officials Announce 296 New Cases of Coronavirus Disease | IDPH [online], www.dph.illinois.gov [dostęp 2020-03-30].
- ↑ Wenjie Yang i inni, Clinical characteristics and imaging manifestations of the 2019 novel coronavirus disease (COVID-19):A multi-center study in Wenzhou city, Zhejiang, China, „Journal of Infection”, 80 (4), 2020, s. 388–393, DOI: 10.1016/j.jinf.2020.02.016, ISSN 0163-4453 [dostęp 2020-03-26] (ang.).
- ↑ David Peña-Otero i inni, Are we ready for the new coronavirus?, „Archivos de Bronconeumología (English Edition)”, 2020, DOI: 10.1016/j.arbr.2020.02.003, ISSN 1579-2129 [dostęp 2020-03-26] (ang.).
- ↑ a b Jieliang Chen, Pathogenicity and transmissibility of 2019-nCoV–A quick overview and comparison with other emerging viruses, „Microbes and Infection”, 22 (2), Special issue on the new coronavirus causing the COVID-19 outbreak, 2020, s. 69–71, DOI: 10.1016/j.micinf.2020.01.004, ISSN 1286-4579 [dostęp 2020-03-26] (ang.).
- ↑ a b c d e Chung-I. Wu, Mu-ming Poo, Moral imperative for the immediate release of 2019-nCoV sequence data, „National Science Review”, DOI: 10.1093/nsr/nwaa030 [dostęp 2020-03-26] (ang.).
- ↑ a b c Nathan D. Grubaugh, Mary E. Petrone, Edward C. Holmes, We shouldn’t worry when a virus mutates during disease outbreaks, „Nature Microbiology”, 5 (4), 2020, s. 529–530, DOI: 10.1038/s41564-020-0690-4, ISSN 2058-5276 [dostęp 2020-03-26] (ang.).
- ↑ Chen Wang i inni, A novel coronavirus outbreak of global health concern, „The Lancet”, 395 (10223), 2020, s. 470–473, DOI: 10.1016/S0140-6736(20)30185-9 [dostęp 2020-03-26] (ang.).
- ↑ a b The Chinese SARS Molecular Epidemiology Consortium, Molecular Evolution of the SARS Coronavirus During the Course of the SARS Epidemic in China, „Science”, 303 (5664), 2004, s. 1666–1669, DOI: 10.1126/science.1092002, ISSN 0036-8075, PMID: 14752165 [dostęp 2020-03-26] (ang.).
- ↑ a b Junxian Ou i inni, RBD mutations from circulating SARS-CoV-2 strains enhance the structural stability and human ACE2 affinity of the spike protein, 17 marca 2020, DOI: 10.1101/2020.03.15.991844 [dostęp 2020-03-26] (ang.).
- ↑ a b c Tung Phan, Genetic diversity and evolution of SARS-CoV-2, „Infection, Genetics and Evolution”, 81, 2020, s. 104260, DOI: 10.1016/j.meegid.2020.104260, ISSN 1567-1348 [dostęp 2020-03-26] (ang.).
- ↑ Thomas R. Frieden, Christopher T. Lee, Early Release - Identifying and Interrupting Superspreading Events–Implications for Control of Severe Acute Respiratory Syndrome Coronavirus 2 - Volume 26, Number 6–June 2020 - Emerging Infectious Diseases journal – CDC, DOI: 10.3201/eid2606.200495 [dostęp 2020-03-26] (ang.).
- ↑ a b Thomas R. Frieden, Christopher T. Lee, Early Release - Identifying and Interrupting Superspreading Events–Implications for Control of Severe Acute Respiratory Syndrome Coronavirus 2 - Volume 26, Number 6–June 2020 - Emerging Infectious Diseases journal – CDC, DOI: 10.3201/eid2606.200495 [dostęp 2020-03-26] (ang.).
- ↑ Roman Woelfel i inni, Clinical presentation and virological assessment of hospitalized cases of coronavirus disease 2019 in a travel-associated transmission cluster, „medRxiv”, 2020, 2020.03.05.20030502, DOI: 10.1101/2020.03.05.20030502 [dostęp 2020-03-28] (ang.).
- ↑ Peng Li i inni, Transmission of COVID-19 in the terminal stage of incubation period: a familial cluster, „International Journal of Infectious Diseases”, 2020, DOI: 10.1016/j.ijid.2020.03.027, ISSN 1201-9712 [dostęp 2020-03-28] (ang.).
- ↑ Guang Zeng i inni, Infectivity of severe acute respiratory syndrome during its incubation period, „Biomedical and environmental sciences: BES”, 22 (6), 2009, s. 502–510, DOI: 10.1016/S0895-3988(10)60008-6, ISSN 0895-3988, PMID: 20337224 [dostęp 2020-03-28].
- ↑ CDC, Coronavirus Disease 2019 (COVID-19) [online], Centers for Disease Control and Prevention, 11 lutego 2020 [dostęp 2020-03-28] (ang.).
- ↑ Jane Qiu, Covert coronavirus infections could be seeding new outbreaks, „Nature”, 2020, DOI: 10.1038/d41586-020-00822-x [dostęp 2020-03-28] (ang.).
- ↑ Kai Kupferschmidt, Study claiming new coronavirus can be transmitted by people without symptoms was flawed, „Science | AAAS”, 2020, DOI: 10.1126/science.abb1524 [dostęp 2020-03-28] (ang.).
- ↑ Yu Chen, Lanjuan Li, SARS-CoV-2: virus dynamics and host response, „The Lancet Infectious Diseases”, 2020, S1473309920302358, DOI: 10.1016/S1473-3099(20)30235-8 [dostęp 2020-03-28] (ang.).
- ↑ a b Xiaowei Li i inni, Molecular immune pathogenesis and diagnosis of COVID-19, „Journal of Pharmaceutical Analysis”, 2020, S2095177920302045, DOI: 10.1016/j.jpha.2020.03.001 [dostęp 2020-03-12] (ang.).
- ↑ Xintian Xu i inni, Evolution of the novel coronavirus from the ongoing Wuhan outbreak and modeling of its spike protein for risk of human transmission, „Science China Life Sciences”, 63 (3), 2020, s. 457–460, DOI: 10.1007/s11427-020-1637-5, ISSN 1674-7305 [dostęp 2020-03-12] (ang.).
- ↑ Peng Zhou i inni, A pneumonia outbreak associated with a new coronavirus of probable bat origin, „Nature”, 579 (7798), 2020, s. 270–273, DOI: 10.1038/s41586-020-2012-7, ISSN 0028-0836 [dostęp 2020-03-12] (ang.).
- ↑ Michael Letko, Vincent Munster, Functional assessment of cell entry and receptor usage for lineage B β-coronaviruses, including 2019-nCoV, 22 stycznia 2020, DOI: 10.1101/2020.01.22.915660 [dostęp 2020-03-12] (ang.).
- ↑ Michael Letko, Andrea Marzi, Vincent Munster, Functional assessment of cell entry and receptor usage for SARS-CoV-2 and other lineage B betacoronaviruses, „Nature Microbiology”, 2020, DOI: 10.1038/s41564-020-0688-y, ISSN 2058-5276 [dostęp 2020-03-12] (ang.).
- ↑ NEJM Journal Watch: Summaries of and commentary on original medical and scientific articles from key medical journals [online], www.jwatch.org [dostęp 2020-03-12].
- ↑ Lisa E. Gralinski, Vineet D. Menachery, Return of the Coronavirus: 2019-nCoV, „Viruses”, 12 (2), 2020, s. 135, DOI: 10.3390/v12020135, ISSN 1999-4915 [dostęp 2020-03-12] (ang.).
- ↑ a b c d e f Xiuyuan Ou i inni, Characterization of spike glycoprotein of SARS-CoV-2 on virus entry and its immune cross-reactivity with SARS-CoV, „Nature Communications”, 11 (1), 2020, s. 1–12, DOI: 10.1038/s41467-020-15562-9, ISSN 2041-1723 [dostęp 2020-03-30] (ang.).
- ↑ Daniel Wrapp i inni, Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation, „Science”, 2020, eabb2507, DOI: 10.1126/science.abb2507, ISSN 0036-8075 [dostęp 2020-03-12] (ang.).
- ↑ Peng Zhou i inni, A pneumonia outbreak associated with a new coronavirus of probable bat origin, „Nature”, 579 (7798), 2020, s. 270–273, DOI: 10.1038/s41586-020-2012-7, ISSN 0028-0836 [dostęp 2020-03-12] (ang.).
- ↑ a b Markus Hoffmann i inni, SARS-CoV-2 Cell Entry Depends on ACE2 and TMPRSS2 and Is Blocked by a Clinically Proven Protease Inhibitor, „Cell”, 2020, S0092867420302294, DOI: 10.1016/j.cell.2020.02.052 [dostęp 2020-03-12] (ang.).
- ↑ Charlotte Harrison, Coronavirus puts drug repurposing on the fast track, „Nature Biotechnology”, 2020, DOI: 10.1038/d41587-020-00003-1 [dostęp 2020-03-12] (ang.).
- ↑ Thailand Medical News, Coronavirus Drug Research: German Researchers Identify Japanese Drug, Camostat Mesylate That Could Be Repurposed To Treat Covid-19 – Thailand Medical News [online], Coronavirus Drug Research: German Researchers Identify Japanese Drug, Camostat Mesylate That Could Be Repurposed To Treat Covid-19 – Thailand Medical News [dostęp 2020-03-12] (ang.).
- ↑ a b ChinaXiv.org 中国科学院科技论文预发布平台 [online], www.chinaxiv.org [dostęp 2020-03-12].
- ↑ a b Thailand Medical News, BREAKING! LatestCoronavirus Research Reveals That The Virus Has Mutated Gene Similar To HIV and Is 1,000 Times More Potent. - Thailand Medical News [online], BREAKING! LatestCoronavirus Research Reveals That The Virus Has Mutated Gene Similar To HIV and Is 1,000 Times More Potent. – Thailand Medical News [dostęp 2020-03-12] (ang.).
- ↑ a b Jennifer Harcourt i inni, Early Release - Severe Acute Respiratory Syndrome Coronavirus 2 from Patient with 2019 Novel Coronavirus Disease, United States - Volume 26, Number 6–June 2020 - Emerging Infectious Diseases journal – CDC, DOI: 10.3201/eid2606.200516 [dostęp 2020-03-12] (ang.).
- ↑ How Furin and ACE2 Interact with the Spike on SARS-CoV-2 [online], Assay Genie [dostęp 2020-03-12] (ang.).
- ↑ a b Shuai Xia i inni, Inhibition of SARS-CoV-2 infection (previously 2019-nCoV) by a highly potent pan-coronavirus fusion inhibitor targeting its spike protein that harbors a high capacity to mediate membrane fusion, 12 marca 2020, DOI: 10.1101/2020.03.09.983247 [dostęp 2020-04-07] (ang.).
- ↑ Koronawirus zakaża skuteczniej niż SARS? Nowe fakty ws. wirusa z Chin [online], www.rmf24.pl [dostęp 2020-03-12].
- ↑ Ke Wang i inni, SARS-CoV-2 invades host cells via a novel route: CD147-spike protein, „bioRxiv”, 2020, 2020.03.14.988345, DOI: 10.1101/2020.03.14.988345 [dostęp 2020-03-26] (ang.).
- ↑ Coronavirus Information | IUPHAR/BPS Guide to PHARMACOLOGY [online], www.guidetopharmacology.org [dostęp 2020-03-26] (ang.).
- ↑ Clinical Study of Anti-CD147 Humanized Meplazumab for Injection to Treat With 2019-nCoV Pneumonia – Full Text View – ClinicalTrials.gov [online], clinicaltrials.gov [dostęp 2020-03-26] (ang.).
- ↑ a b Leila Mousavizadeh, Sorayya Ghasemi, Genotype and phenotype of COVID-19: Their roles in pathogenesis, „Journal of Microbiology, Immunology and Infection”, 2020, DOI: 10.1016/j.jmii.2020.03.022, ISSN 1684-1182 [dostęp 2020-04-06] (ang.).
- ↑ Soeren Lukassen i inni, SARS-CoV-2 receptor ACE2 and TMPRSS2 are predominantly expressed in a transient secretory cell type in subsegmental bronchial branches, „bioRxiv”, 2020, 2020.03.13.991455, DOI: 10.1101/2020.03.13.991455 [dostęp 2020-04-06] (ang.).
- ↑ Markus Hoffmann i inni, SARS-CoV-2 Cell Entry Depends on ACE2 and TMPRSS2 and Is Blocked by a Clinically Proven Protease Inhibitor, „Cell”, 2020, S0092867420302294, DOI: 10.1016/j.cell.2020.02.052, PMID: 32142651, PMCID: PMC7102627 [dostęp 2020-04-06] (ang.).
- ↑ David H. Brann i inni, Non-neural expression of SARS-CoV-2 entry genes in the olfactory epithelium suggests mechanisms underlying anosmia in COVID-19 patients, „bioRxiv”, 2020, 2020.03.25.009084, DOI: 10.1101/2020.03.25.009084 [dostęp 2020-04-06] (ang.).
- ↑ a b Christian A. Devaux i inni, New insights on the antiviral effects of chloroquine against coronavirus: what to expect for COVID-19?, „International Journal of Antimicrobial Agents”, 2020, DOI: 10.1016/j.ijantimicag.2020.105938, ISSN 0924-8579, PMID: 32171740, PMCID: PMC7118659 [dostęp 2020-04-07].
- ↑ a b Xiaowei Li i inni, Molecular immune pathogenesis and diagnosis of COVID-19, „Journal of Pharmaceutical Analysis”, 2020, DOI: 10.1016/j.jpha.2020.03.001, ISSN 2095-1779 [dostęp 2020-04-06] (ang.).
- ↑ Edyta Abramczuk i inni, Niepandemiczne koronawirusy człowieka [PDF], „Postępy mikrobiologii”.
- ↑ Luca Cristiani i inni, Will children reveal their secret? The coronavirus dilemma, „The European Respiratory Journal”, 2020, DOI: 10.1183/13993003.00749-2020, ISSN 0903-1936, PMID: 32241833, PMCID: PMC7113798 [dostęp 2020-04-09].
- ↑ Elio Issa i inni, SARS-CoV-2 and ORF3a: Non-Synonymous Mutations and Polyproline Regions, „bioRxiv”, 2020, 2020.03.27.012013, DOI: 10.1101/2020.03.27.012013 [dostęp 2020-04-09] (ang.).
- ↑ Savannah F. Pedersen, Ya-Chi Ho, SARS-CoV-2: A Storm is Raging, „The Journal of Clinical Investigation”, 2020, DOI: 10.1172/JCI137647, ISSN 1558-8238, PMID: 32217834 [dostęp 2020-04-09].
- ↑ Canrong Wu i inni, Analysis of therapeutic targets for SARS-CoV-2 and discovery of potential drugs by computational methods, „Acta Pharmaceutica Sinica B”, 2020, S2211383520302999, DOI: 10.1016/j.apsb.2020.02.008 [dostęp 2020-03-12] (ang.).
- ↑ Nisreen M.A. Okba i inni, SARS-CoV-2 specific antibody responses in COVID-19 patients, „medRxiv”, 2020, 2020.03.18.20038059, DOI: 10.1101/2020.03.18.20038059 [dostęp 2020-04-09] (ang.).
- ↑ Chen Dong i inni, Characterization of anti-viral immunity in recovered individuals infected by SARS-CoV-2, „medRxiv”, 2020, 2020.03.17.20036640, DOI: 10.1101/2020.03.17.20036640 [dostęp 2020-04-09] (ang.).
- ↑ Irani Thevarajan i inni, Breadth of concomitant immune responses prior to patient recovery: a case report of non-severe COVID-19, „Nature Medicine”, 2020, s. 1–3, DOI: 10.1038/s41591-020-0819-2, ISSN 1546-170X [dostęp 2020-03-21] (ang.).
- ↑ Yonggang Zhou i inni, Pathogenic T cells and inflammatory monocytes incite inflammatory storm in severe COVID-19 patients, „National Science Review”, 2020, nwaa041, DOI: 10.1093/nsr/nwaa041, ISSN 2095-5138 [dostęp 2020-03-21] (ang.).
- ↑ a b Zhiqiang Zheng i inni, Monoclonal antibodies for the S2 subunit of spike of SARS-CoV cross-react with the newly-emerged SARS-CoV-2, „bioRxiv”, 2020, 2020.03.06.980037, DOI: 10.1101/2020.03.06.980037 [dostęp 2020-03-21] (ang.).
- ↑ Kuo-Ming Lip i inni, Monoclonal Antibodies Targeting the HR2 Domain and the Region Immediately Upstream of the HR2 of the S Protein Neutralize In Vitro Infection of Severe Acute Respiratory Syndrome Coronavirus, „Journal of Virology”, 80 (2), 2006, s. 941–950, DOI: 10.1128/JVI.80.2.941-950.2006, ISSN 0022-538X, PMID: 16378996 [dostęp 2020-03-21] (ang.).
- ↑ Novel Coronavirus (2019-nCoV) Situation Report – 10 [online], Światowa Organizacja Zdrowia [dostęp 2020-02-01] (ang.).
- ↑ Coronavirus disease 2019 (COVID-19) Situation Report – 31. who.int. [dostęp 2020-02-20]. (ang.).
- ↑ Kai Kupferschmidt, Jon Cohen, WHO launches global megatrial of the four most promising coronavirus treatments, „Science | AAAS”, 22 marca 2020 [dostęp 2020-03-24] (ang.).
- ↑ WHO Director-General’s opening remarks at the Mission briefing on COVID-19 – 19 March 2020 [online], www.who.int [dostęp 2020-03-24] (ang.).
- ↑ Wen-Hsiang Chen i inni, The SARS-CoV-2 Vaccine Pipeline: an Overview, „Current Tropical Medicine Reports”, 2020, DOI: 10.1007/s40475-020-00201-6, ISSN 2196-3045 [dostęp 2020-03-24] (ang.).
- ↑ Weronika Bartosik, Szczepionki mRNA – obiecująca alternatywa [online].
- ↑ Michelle Roberts, Coronavirus: US volunteers test first vaccine, „BBC News”, 17 marca 2020 [dostęp 2020-03-24] (ang.).
- ↑ Trwają prace nad szczepionką na koronawirusa SARS-CoV-2 [online], pulsmedycyny.pl [dostęp 2020-03-24].
- ↑ Case of the Index Patient Who Caused Tertiary Transmission of COVID-19 Infection in Korea: the Application of Lopinavir/Ritonavir for the Treatment of COVID-19 Infected Pneumonia Monitored by Quantitative RT-PCR., „Journal of Korean Medical Science”, 35 (6), 2020, e79, DOI: 10.3346/jkms.2020.35.e79, PMID: 32056407, PMCID: PMC7025910 [dostęp 2020-02-23] (ang.).
- ↑ Zhijian Xu i inni, Nelfinavir was predicted to be a potential inhibitor of 2019-nCov main protease by an integrative approach combining homology modelling, molecular docking and binding free energy calculation, „bioRxiv”, 2020, 2020.01.27.921627, DOI: 10.1101/2020.01.27.921627 [dostęp 2020-03-11] (ang.).
- ↑ Jon Cohen, Can an anti-HIV combination or other existing drugs outwit the new coronavirus?, „Science | AAAS”, 2020, DOI: 10.1126/science.abb0659 [dostęp 2020-03-11] (ang.).
- ↑ Xin Liu, Xiu-Jie Wang, Potential inhibitors for 2019-nCoV coronavirus M protease from clinically approved medicines, „bioRxiv”, 2020, 2020.01.29.924100, DOI: 10.1101/2020.01.29.924100 [dostęp 2020-03-11] (ang.).
- ↑ Catharine I. Paules, Hilary D. Marston, Anthony S. Fauci, Coronavirus Infections–More Than Just the Common Cold, „JAMA]”, 323 (8), 2020, s. 707–708, DOI: 10.1001/jama.2020.0757, ISSN 0098-7484 [dostęp 2020-03-11] (ang.).
- ↑ 上海药物所和上海科技大学联合发现一批可能对新型肺炎有治疗作用的老药和中药----中国科学院 [online], www.cas.cn [dostęp 2020-03-11].
- ↑ Liying Dong, Shasha Hu, Jianjun Gao, Discovering drugs to treat coronavirus disease 2019 (COVID-19), „Drug Discoveries & Therapeutics”, 14 (1), 2020, s. 58–60, DOI: 10.5582/ddt.2020.01012 [dostęp 2020-03-21].
- ↑ Kumari G. Lokugamage, Craig Schindewolf, Vineet D. Menachery, SARS-CoV-2 sensitive to type I interferon pretreatment, „bioRxiv”, 2020, 2020.03.07.982264, DOI: 10.1101/2020.03.07.982264 [dostęp 2020-03-21] (ang.).
- ↑ Wen-Chien Ko i inni, Arguments in favour of remdesivir for treating SARS-CoV-2 infections, „International Journal of Antimicrobial Agents”, 2020, s. 105933, DOI: 10.1016/j.ijantimicag.2020.105933 [dostęp 2020-03-21] (ang.).
- ↑ Manli Wang i inni, Remdesivir and chloroquine effectively inhibit the recently emerged novel coronavirus (2019-nCoV) in vitro, „Cell Research”, 30 (3), 2020, s. 269–271, DOI: 10.1038/s41422-020-0282-0, ISSN 1001-0602, PMID: 32020029, PMCID: PMC7054408 [dostęp 2020-03-21] (ang.).
- ↑ Drug meant for Ebola may also work against coronaviruses: Understanding how drugs work is an important step in developing new treatments for COVID-19 [online], ScienceDaily [dostęp 2020-03-11] (ang.).
- ↑ Fu-Sheng Wang, Chao Zhang, What to do next to control the 2019-nCoV epidemic?, „The Lancet”, 395 (10222), 2020, s. 391–393, DOI: 10.1016/S0140-6736(20)30300-7, ISSN 0140-6736, PMID: 32035533 [dostęp 2020-03-11] (ang.).
- ↑ Guangdi Li, Erik De Clercq, Therapeutic options for the 2019 novel coronavirus (2019-nCoV), „Nature Reviews Drug Discovery”, 19 (3), 2020, s. 149–150, DOI: 10.1038/d41573-020-00016-0, ISSN 1474-1776 [dostęp 2020-03-21] (ang.).
- ↑ BMJ Best Practice. COVID-19. [PDF].
- ↑ AIFA e Gilead annunciano che l’Italia è tra i Paesi che testeranno l’antivirale remdesivir per il trattamento del COVID-19 [online], aifa.gov.it [dostęp 2020-03-21] (wł.).
- ↑ Yaron Steinbuch, Coronavirus patient in Spain reportedly recovers after being treated with HIV drug [online], New York Post, 5 marca 2020 [dostęp 2020-03-14] (ang.).
- ↑ Connor Boyd, Coronavirus patient 'recovers after being treated with a HIV drug’ [online], Mail Online, 5 marca 2020 [dostęp 2020-03-14].
- ↑ Bin Cao i inni, A Trial of Lopinavir–Ritonavir in Adults Hospitalized with Severe Covid-19, „The New England Journal of Medicine”, 0 (0), 2020, null, DOI: 10.1056/NEJMoa2001282, ISSN 0028-4793, PMID: 32187464 [dostęp 2020-03-24].
- ↑ Wrocław: pacjenci zakażeni koronawirusem będą leczeni lekami m.in. na HIV i malarię | Polska Agencja Prasowa SA [online], www.pap.pl [dostęp 2020-03-14].
- ↑ China CDC developing novel coronavirus vaccine - Xinhua | English.news.cn [online], www.xinhuanet.com [dostęp 2020-03-11].
- ↑ 马驰, Drugs with potential against coronavirus in human trials - Chinadaily.com.cn [online], www.chinadaily.com.cn [dostęp 2020-03-11].
- ↑ Jianjun Gao, Zhenxue Tian, Xu Yang, Breakthrough: Chloroquine phosphate has shown apparent efficacy in treatment of COVID-19 associated pneumonia in clinical studies, „Bioscience Trends”, 2020, DOI: 10.5582/bst.2020.01047, ISSN 1881-7823, PMID: 32074550 [dostęp 2020-03-11].
- ↑ 陈子琰, Potential coronavirus drug approved for marketing - Chinadaily.com.cn [online], www.chinadaily.com.cn [dostęp 2020-03-11].
- ↑ 蒋成龙, Progress seen in epidemic fight - Chinadaily.com.cn [online], www.chinadaily.com.cn [dostęp 2020-03-11].
- ↑ Wiener Zeitung Online, Coronavirus: Zaghafte Erfolge bei Medikamentensuche [online], Österreich Politik – Nachrichten – Wiener Zeitung Online [dostęp 2020-03-11] (niem.).
- ↑ Apotheke Adhoc, Malariamittel gegen Coronavirus [online], APOTHEKE ADHOC [dostęp 2020-03-11] (niem.).
- ↑ Komunikat dla profesjonalistów medycznych | Adamed [online], www.adamed.com [dostęp 2020-03-13].
- ↑ Informacja w sprawie druków informacyjnych do produktu Arechin (Chloroquini phosphas), 250 mg, tabletki | Urząd Rejestracji Produktów Leczniczych, Wyrobów Medycznych i Produktów Biobójczych [online], www.urpl.gov.pl [dostęp 2020-03-14].
- ↑ Dawei Wang i inni, Clinical Characteristics of 138 Hospitalized Patients With 2019 Novel Coronavirus–Infected Pneumonia in Wuhan, China, „JAMA]”, 323 (11), 2020, s. 1061–1069, DOI: 10.1001/jama.2020.1585, ISSN 0098-7484 [dostęp 2020-03-24] (ang.).
- ↑ Xueting Yao i inni, In Vitro Antiviral Activity and Projection of Optimized Dosing Design of Hydroxychloroquine for the Treatment of Severe Acute Respiratory Syndrome Coronavirus 2 (SARS-CoV-2), „Clinical Infectious Diseases”, 2020, ciaa237, DOI: 10.1093/cid/ciaa237, ISSN 1058-4838 [dostęp 2020-03-24] (ang.).
- ↑ a b Srivatsan Padmanabhan, Potential dual therapeutic approach against SARS-CoV-2/COVID-19 with Nitazoxanide and Hydroxychloroquine [PDF], DOI: 10.13140/RG.2.2.28124.74882.
- ↑ China approves use of Roche drug in battle against coronavirus complications, „Reuters”, 4 marca 2020 [dostęp 2020-03-24] (ang.).
- ↑ ChinaXiv.org 中国科学院科技论文预发布平台 [online], chinaxiv.org [dostęp 2020-03-24].
- ↑ Azioni intraprese per favorire la ricerca e l’accesso ai nuovi farmaci per il trattamento del COVID-19 [online], aifa.gov.it [dostęp 2020-03-24] (wł.).
- ↑ 3 patients get better on arthritis drug - English [online], ANSA.it, 13 marca 2020 [dostęp 2020-03-24] (ang.).
- ↑ Coronavirus, via libera dell’Aifa al farmaco anti-artrite efficace su 3 pazienti e a un antivirale: test in 5 centri [online], www.ilmessaggero.it [dostęp 2020-03-24] (wł.).
- ↑ National Center for Biotechnology Information i inni, SUMMARY, Canadian Agency for Drugs and Technologies in Health, sierpień 2015 [dostęp 2020-03-24] (ang.).
- ↑ China turns Roche arthritis drug Actemra against COVID-19 in new treatment guidelines [online], FiercePharma [dostęp 2020-03-24] (ang.).
- ↑ 陈子琰, Plasma therapy could lead to development of vaccine for novel coronavirus pneumonia - Chinadaily.com.cn [online], www.chinadaily.com.cn [dostęp 2020-03-11].
- ↑ 陈子琰, More plasma needed from recovered coronavirus patients, experts say - Chinadaily.com.cn [online], www.chinadaily.com.cn [dostęp 2020-03-11].
- ↑ Arturo Casadevall, Liise-anne Pirofski, The convalescent sera option for containing COVID-19, „Journal of Clinical Investigation”, 2020, 10.1172/JCI138003, DOI: 10.1172/JCI138003, ISSN 0021-9738 [dostęp 2020-03-21] (ang.).
- ↑ China develops convalescent plasma therapy for COVID-19 patients- China.org.cn [online], www.china.org.cn [dostęp 2020-03-11].
- ↑ Nowy oręż w walce z koronawirusem – osocze wyleczonych pacjentów [online], www.focus.pl [dostęp 2020-03-11].
- ↑ Koronawirus w Chinach: Osocze wyleczonych leczy chorych [online], www.rp.pl [dostęp 2020-03-11].
- ↑ Robert Phillips, TMPRSS2 promotes metastasis through proteolysis, „Nature Reviews Urology”, 11 (10), 2014, s. 546–546, DOI: 10.1038/nrurol.2014.238, ISSN 1759-4820 [dostęp 2020-03-31] (ang.).
- ↑ Evaluating the Efficacy and Safety of Bromhexine Hydrochloride Tablets Combined With Standard Treatment/ Standard Treatment in Patients With Suspected and Mild Novel Coronavirus Pneumonia (COVID-19) – Full Text View – ClinicalTrials.gov [online], clinicaltrials.gov [dostęp 2020-03-31] (ang.).
- ↑ Białko powstrzymujące koronawirusy powstaje w ludzkim organizmie. Szansa na opracowanie leku [online], KopalniaWiedzy.pl [dostęp 2020-03-14].
- ↑ Katrina B. Mar i inni, LY6E mediates an evolutionarily conserved enhancement of virus infection by targeting a late entry step, „Nature Communications”, 9 (1), 2018, s. 3603, DOI: 10.1038/s41467-018-06000-y, ISSN 2041-1723, PMID: 30190477, PMCID: PMC6127192 [dostęp 2020-03-14] (ang.).
- ↑ Stephanie Pfaender i inni, LY6E impairs coronavirus fusion and confers immune control of viral disease, 7 marca 2020, DOI: 10.1101/2020.03.05.979260 [dostęp 2020-03-14] (ang.).
- ↑ a b Wioletta Rut i inni, Substrate specificity profiling of SARS-CoV-2 Mpro protease provides basis for anti-COVID-19 drug design, „bioRxiv”, 2020, 2020.03.07.981928, DOI: 10.1101/2020.03.07.981928 [dostęp 2020-03-24] (ang.).
- ↑ a b Linlin Zhang i inni, Crystal structure of SARS-CoV-2 main protease provides a basis for design of improved α-ketoamide inhibitors, „Science”, 2020, DOI: 10.1126/science.abb3405, ISSN 0036-8075, PMID: 32198291 [dostęp 2020-03-24] (ang.).
- ↑ Politechnika Śląska w międzynarodowym projekcie badającym kluczowy enzym koronawirusa [online], PolskieRadio24.pl [dostęp 2020-03-24].
- ↑ Nauka w Polsce PAP, Politechnika Śląska w międzynarodowym projekcie badającym kluczowy enzym wirusa SARS-CoV-2 [online], Zdrowie i medycyna Wprost, 24 marca 2020 [dostęp 2020-03-24].
- ↑ Zhijian Xu i inni, Nelfinavir was predicted to be a potential inhibitor of 2019-nCov main protease by an integrative approach combining homology modelling, molecular docking and binding free energy calculation, „bioRxiv”, 2020, 2020.01.27.921627, DOI: 10.1101/2020.01.27.921627 [dostęp 2020-03-28] (ang.).
- ↑ Jinnong Zhang i inni, Therapeutic and triage strategies for 2019 novel coronavirus disease in fever clinics, „The Lancet Respiratory Medicine”, 8 (3), 2020, e11–e12, DOI: 10.1016/S2213-2600(20)30071-0 [dostęp 2020-03-11] (ang.).
- ↑ Coronavirus infection: Prevention and risks [online], Public Health Agency of Canada, Government of Canada, 16 stycznia 2020 [dostęp 2020-01-28].
- ↑ Coronavirus [online], Centers for Disease Control and Prevention, 28 stycznia 2020 [dostęp 2020-01-28] (ang.).
- ↑ Advice for public [online], World Health Organization [dostęp 2020-01-28] (ang.).
- ↑ Koronawirus – przyczyny, objawy i leczenie choroby z Chin [online], Medme.pl, 22 stycznia 2020 [dostęp 2020-01-28].
- ↑ Coronavirus disease 2019 (COVID-19) Situation Report – 35. who.int. [dostęp 2020-02-24]. (ang.).
- ↑ WPROST.pl, Będzie obowiązek zasłaniania twarzy. Minister Szumowski przedstawił nowe zasady [online], Zdrowie i medycyna Wprost, 9 kwietnia 2020 [dostęp 2020-04-09] (pol.).
- ↑ PAPSylwia Wysocka, Kwarantanna ponad 270 migrantów i załogi statku organizacji pozarządowej we Włoszech [online], portalmorski.pl, 24 lutego 2020 [dostęp 2020-02-26].
- ↑ a b Hong-Ying Li i inni, A qualitative study of zoonotic risk factors among rural communities in southern China., „International Health”, Oxford University Press, 2020, DOI: 10.1093/inthealth/ihaa001, PMID: 32040190 (ang.).
- ↑ Quan Liu, Lili Cao, Xing-Quan Zhu, Major emerging and re-emerging zoonoses in China: a matter of global health and socioeconomic development for 1.3 billion., „Int J Infect Dis”, 25, 2014, s. 65–72, DOI: 10.1016/j.ijid.2014.04.003, PMID: 24858904 (ang.).
- ↑ WHO Director-General’s opening remarks at the media briefing on COVID-19 – 11 March 2020 [online], www.who.int [dostęp 2020-03-12] (ang.).
- ↑ Stara, dobra szkoła KGB w roli „odgrzewanego kotleta”, czyli amerykańskie pochodzenie koronawirusa [online], cyberdefence.pl, 31 stycznia 2020.
- ↑ EUvsDisinfo: Analyses and disinformation cases – Coronavirus [online], EUvsDisinfo [dostęp 2020-03-03] (ang.).
- ↑ Rosja prowadzi kampanię dezinformacyjną w sprawie koronawirusa [online], polskieradio.pl, 23 lutego 2020.
- ↑ US says Russia behind huge coronavirus disinformation campaign [online], dtnext.in, 22 lutego 2020 (ang.).
- ↑ Millions of tweets peddled conspiracy theories about coronavirus in other countries, an unpublished U.S. report says [online], washingtonpost.com, 29 lutego 2020 (ang.).
- ↑ Roughly 2 million tweets spread dangerous misinformation and hoaxes about the coronavirus, according to an unpublished State Department report [online], businessinsider.com, 1 marca 2020 (ang.).
- ↑ Coronavirus: Fake news is spreading fast [online], bbc.com, 26 lutego 2020 (ang.).
- ↑ Dezinformacja napędza lęk przed koronawirusem. Stoją za nia Rosjanie [online], newsweek.pl, 25 lutego 2020.
- ↑ a b c d EU Rapid Alert System used amid coronavirus disinformation campaign [online], euroactiv.com, 4 marca 2020 (ang.).
- ↑ a b UK Government sets up counter-disinformation unit [online], dailymail.co.uk, 9 marca 2020.
- ↑ a b Coronavirus: Unit set up to counter false claims [online], bbc.co.uk, 9 marca 2020.
- ↑ a b c d Murray Brewster, Send in the trolls: Canada braces for an online disinformation assault on COVID-19 [online], CBC, 14 marca 2020.
