Wikiprojekt:Tłumaczenie artykułów/Enzymy

Enzymy - to białkowe katalizatory przyspieszające specyficzne reakcje chemiczne wskutek obniżenia energii aktywacji potrzebnej do przebiegu tych reakcji[1], rodzaj biokatalizatorów. Właściwie wszystkie przemiany chemiczne związane z funkcjonowaniem organizmów żywych (a także wirusów i podobnych) wymagają współudziału enzymów, by zapewnić wystarczającą wydajność reakcji. Enzymy, z racji tego, że są wysoce specyficzne wobec substratów i katalizują zaledwie kilka reakcji spośród wielu możliwych, definiują procesy metaboliczne i biochemiczne związane z funkcjonowaniem organizmów żywych. Znane enzymy katalizują przebieg co najmniej 4000 różnych reakcji biochemicznych[2]. Nauka o enzymach i ich działaniu to enzymologia.
Jak wszystkie katalizatory, enzymy działają na zasadzie obniżenia energii aktywacji (Ea lub ΔG‡) reakcji chemicznej, przyspieszając w ten sposób przebieg reakcji (patrz: Struktury i mechanizmy działania). Większość reakcji enzymatycznych (tj. z udziałem enzymów) przebiega miliony razy szybciej niż ich niekatalizowane enzymatycznie odpowiedniki. Jednym z najszybszych znanych enzymów jest anhydraza węglanowa, która w ciągu sekundy może uwodnić 106 cząsteczek dwutlenku węgla[3], z kolei jeden z najwolniejszych - lizozym, katalizuje 0,5 reakcji na sekundę (1 co 2 sekundy)[4]. Jak wszystkie katalizatory, również enzymy nie zużywają się w trakcie przebiegu reakcji, a także nie wpływają na ich równowagę. Jakkolwiek enzymy różnią się od zwykłych katalizatorów, przejawiając znacznie większą specyficzność substratową. Aktywność enzymatyczna może być zatrzymana lub obniżona przez inne cząsteczki - inhibitory. Z kolei aktywatory enzymatyczne to cząsteczki zwiększające aktywność enzymów. Wiele leków i trucizn jest inhibitorami enzymów. Ponadto aktywność enzymów zależy od parametrów fizyko-chemicznych środowiska reakcji, jak: temperatura, pH, siła jonowa, obecność niektórych jonów i innych.
Jakkolwiek wszystkie enzymy to białka, nie wszystkie reakcje biochemiczne są katalizowane przez nie. Znane są cząsteczki RNA - rybozymy, które także biorą udział w biochemicznej katalizie niektórych reakcji[5]. Także niektóre sztucznie stworzone cząsteczki, zwane sztucznymi enzymami, przejawiają podobną do enzymatycznej aktywność katalityczną[6].
Niektóre enzymy znalazły zastosowanie przemysłowe (patrz: Zastosowanie przemysłowe), m.in. w przemyśle spożywczym czy chemii leków. Wiele produktów używanych w gospodarstwach domowych zawiera enzymy w celu podniesienia wydajności ich działania, jak proszki do prania czy enzymatyczne wywabiacze do plam. Enzymy są także powszechnie używane we współczesnych naukach biologicznych i medycznych oraz w diagnostyce medycznej.
Etymologia nazwy i historia odkrycia
[edytuj | edytuj kod]
Na przełomie XVIII i XIX wieku obserwowano już trawienie in vitro mięsa przez wydzieliny żołądka[7] oraz rozkład skrobi do cukrów prostych przez ekstrakty roślinne lub ślinę. Jakkolwiek, mechanizmy stojące za tymi procesami nie były znane[8].
W XIX wieku, podczas studiów nad fermentacją cukrów do alkoholu przeprowadzaną przez drożdże, Louis Pasteur doszedł do wniosku, że reakcja ta jest katalizowana przez "siłę życiową" zawartą w drożdżach, którą nazwał "fermentem". Podejrzewano, że może być ona aktywna tylko w żywych organizmach. Pasteur odnotował, że "fermentacja alkoholowa jest procesem powiązanym z życiem i organizacją komórek drożdżowych, a nie ze śmiercią czy rozkładem komórek"[9].
W 1878 niemiecki fizjolog Wilhelm Kühne, by opisać te procesy, ukuł termin enzym, który pochodzi z greckiego ενζυμον i oznacza "w zaczynie". Później słowo enzym było używane w odniesieniu do substancji nieożywionych, jak chociażby pepsyna, a słowo ferment w odniesieniu do aktywności chemicznej wykazywanej przez organizmy żywe. W języku polskim oba terminy były stosowane wymiennie, przy czym ten ostatni jest obecnie archaiczny[10].
W 1897 Eduard Buchner rozpoczął badania nad zdolnością ekstraktów drożdżowych do fermentacji cukrów przy nieobecności żywych komórek. Po szeregów eksperymentów przeprowadzonych na Uniwersytecie Humboldta w Berlinie, odkrył on, że taka fermentacja jest możliwa[11]. Nazwał on enzym przeprowadzający taką fermentację sacharozy - "zymazą"[12]. W 1907 Eduard Buchner otrzymał Nagrodę Nobla w dziedzinie chemii "za swoje badania biochemiczne i odkrycie fermentacji bez udziału komórek". Zgodnie z nazwą pierwszego enzymu, obecna nomenklatura ich nazewnictwa, przewiduje tworzenie nazwy od reakcji, którą przeprowadzają (patrz: Konwencja nazewnictwa). Zazwyczaj dodaje się przyrostek -aza do nazwy substratu przetwarzanego w reakcji (np. laktaza to enzym przetwarzający laktozę) lub do ogólnej nazwy reakcji (np. polimeraza DNA to enzym syntezujący polimery DNA).

Po udowodnieniu, że enzymy mogą funkcjonować poza komórką żywą, następnym krokiem było opisanie ich natury biochemicznej. Wielu badaczy zaobserwowało, że aktywność enzymatyczna była w jakiś sposób związana z białkami, ale kilku naukowców (w tym laureat Nagrody Nobla Richard Willstätter) dowodziło, że białka nie mogą być jedynymi "nośnikami" aktywności enzymatycznej i same z siebie nie są zdolne do przeprowadzania katalizy. Jednak w 1926, James B. Sumner udowodnił, że enzym ureaza to czyste białko i skrystalizował je. Powtórzył to także w 1937 dla katalazy. Ostateczny wniosek, że enzymy mogą być czystymi białkami, został potwierdzony przez J.H. Northropa i W.M. Stanleya, którzy pracowali nad enzymami trawiennymi - trypsyną i chymotrypsyną. Ta trójka naukowców została za swoje odkrycia uhonorowana w 1946 Nagrodą Nobla w dziedzinie chemii[13].
Odkrycie, że enzymy mogą być wykrystalizowane, pozwoliło na późniejsze badanie ich struktur metodami rentgenografii strukturalnej. Pierwszym enzymem, którego strukturę zbadano w ten sposób, był lizozym, enzym obecny m.in. w łzach, ślinie i białku jaja. Jego struktura została rozwiązana przez grupę Davida Phillipsa i opublikowana w 1965[14]. Uzyskanie modelu struktury lizozymu o tej wysokiej rozdzielczości było początkiem biologii strukturalnej enzymów i zrozumienia mechanizmów ich działania na poziomie molekularnym.
Struktury i mechanizmy działania
[edytuj | edytuj kod]
Enzymy to białka o zróżnicowanej wielkości, od na przykład 62 aminokwasów w łańcuchu i monomerecznej budowie w przypadku tautomerazy 4-oksokrotonianu (4-OT)[15] do ponad 2 500 w zwierzęcej syntazie kwasów tłuszczowych[16]. Aktywność enzymów jest determinowana ich strukturą czwartorzędową (ułożeniem przestrzennym)[17]. Enzymy są zazwyczaj dużo większe od substratów, które przerabiają, ale z kolei zwykle tylko kilka kluczowych aminokwasów jest bezpośrednio zaangażowane w katalizę[18]. Region, który bezpośrednio wiąże i oddziaływuje z substratem, oraz zawiera kluczowe do przebiegu reakcji reszty aminokwasowe, nazywany jest centrum aktywnym. Prócz niego enzymy mogą zawierać miejsca wiązania kofaktorów - niezbędnych do aktywności enzymatycznej lub jej regulacji (patrz: Kofaktory, grupy prostetyczne i koenzymy). Prócz tego enzymy mogą także zawierać dodatkowe miejsca wiązania małych cząsteczek, na przykład pośrednich lub bezpośrednich produktów czy substratów szlaków metabolicznych obsługiwanych przez enzym, które związane mogą dodatkowo regulować aktywność enzymu na zasadzie sprzężenia zwrotnego.
Jak każde białko, enzymy są syntezowane jako długie łańcuchy aminokwasowe, które następnie zwijają się i przybierają odpowiednią strukturę przestrzenną. Indywidualne, zwinięte łańcuchy białkowe, mogą także asocjować w większe kompleksy. Takie enzymy nazywa się wtedy multimerycznymi (wielopodjednostkowymi). W przypadku asocjacji kilku takich samych peptydów (podjednostek) mówi się o homomerach (np. homodimer - kompleks złożony z dwóch jednakowych peptydów), a gdy asocują różne jakościowo podjednostki, o heteromerach (np. heteropentamer - kompleks pięciu różnych łańcuchów peptydowych). Asocjacja podjednostek enzymów może być wymagana by dopełnić nawzajem swoje funkcje, by w ogóle móc katalizować reakcję biochemiczną, lub by obsługiwać wielokrotność tej samej reakcji czy cały ich szereg (odcinek szlaku metabolicznego) [potrzebny przypis].
Specyficzność
[edytuj | edytuj kod]Enzymy charakteryzują się zwykle dużą specyficznością pod względem katalizowanej reakcji, jak i również konwertowanych substratów. Za wysoką specyficzność odpowiada kształt cząsteczeki enzymu dopasowny do substratów geometrycznie, ale także pod względem oddziaływań hydrofobowo-hydrofilowych oraz elektrostatycznych. Enzymy wykazują także wysoki poziom stereospecyficzności, regioselektywności i chemoselektywności[19].
Niektóre z enzymów zaangażowaniach w kopiowanie i ekspresję informacji genetycznej ponad swój wysoki stopień specyficzności i precyzji, wykazują także zdolność działania "korekcyjnego" (ang. reading-proof activity). Przykładem może być polimeraza DNA I, która katalizuje syntezę nici DNA w czasie replikacji i naprawy[20]. Polimeraza ta, nie tylko jest wysoce precyzyjna, ale i zdolna do poprawy ewentualnie zaistniałego błędu (źle wbudowanego nukleotydu), w rezultacie dzięki tym dwóm procesom (precyzyjność syntezy i korekcja błędów), ryzyko popełnienia błędu przez ssacze polimerazy, który nie zostanie zauważony w procesie syntezy, wynosi mniej niż jeden na milion wprowadzonych nukleotydów [3]. Podobne mechanizmy korekcyjne posiadają polimerazy RNA[21], syntetazy aminoacylo tRNA[22] i rybosomy[23].
Z kolei niektóre enzymy uczestniczące w produkcji metabolitów wtórnych, charakteryzują się stosunkowo szerokim zakresem dobierania różnych substratów. Te enzymy, które posiadają szczególną zdolność wiązania różnych substratów, odgrywają bardzo ważną rolę w ewolucji nowych szlaków metabolicznych[24].
Model "klucza i zamka" (ang. "Lock and key" model)
[edytuj | edytuj kod]Enzymy są niezwykle specyficzne wobec swoich substratów. Jak sugerował w roku 1894 Hermann Emil Fischer, zarówno enzym jak i jego substraty są do siebie geometrycznie dopasowane, w taki sposób, że idealnie pasują jeden do drugiego (jak "klucz i zamek")[25]. Model ten wyjaśnia specyficzność enzymów, ale nie wyjaśnia w jaki sposób stabilizowany jest stan przejściowy podczas reakcji enzymatycznej.
Model indukowanego dopasowania (ang. Induced fit model)
[edytuj | edytuj kod]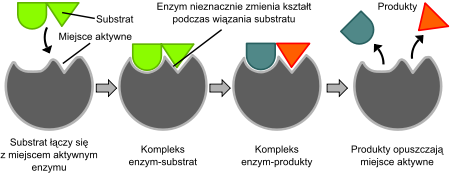
W 1958 Daniel Koshland zasugerował modyfikację modelu "klucza i zamka". Ponieważ enzymy są zwykle dość elastyczne strukturalnie, ich centrum aktywne podlega ciągłym rearanżacjom przestrzennym podczas oddziaływania z substratami[26]. W rezultacie, substrat nie tyle wiąże się do niezmiennego strukturalnie miejsca aktywnego, ale grupy boczne aminokwasów je tworzące podlegają rearanżacjom przestrzennym, ściśle dopasowując swe pozycje do wiązanego substratu, co dopiero umożliwia przeprowadzenie katalizy. W niektórych wypadkach, jak np. glikozydazy, także cząsteczki substratu podlegają lekkim zmianom strukturalnym po wejściu do centrum aktywnego, wzmacniając w ten sposób dopasowanie[27]. Centrum aktywne kontynuuje rearanżację, aż osiągnięte zostanie końcowe stadium dopasowania z substratem[28].
Mechanizmy
[edytuj | edytuj kod]Enzymy mogą na kilka różnych sposobów zmniejszać swobodną energię aktywacji Gibbsa (ΔG‡)[29]:
- Obniżanie energii aktywacji przez tworzenie środowiska, w którym następuje stabilizacja stanu przejściowego (np. zniekształcenie cząsteczki substratu - dzieje się to przez utrwalenie konformacji stanu przejściowego pomiędzy substratem a produktem oraz zniekształcenie przez enzym wiązań w cząsteczce substratu dzięki czemu zmniejsza się suma energii wymaganej do zakończenia przejścia).
- Obniżanie energii stanu przejściowego przez likwidację niefaworyzowanych energetycznie oddziaływań ze środowiskiem, i ich zamianę na faworyzowane energetycznie oddziaływania z centrum aktywnym (np. np oddziaływania elektrostatyczne ładunków o przeciwnych znakach).
- Wykorzystanie alternatywnego szlaku przejścia, np. tymczasowa reakcja substratu do pośredniego kompleksu enzym-substrat (ES), która nie byłaby możliwa w nieobecności enzymu.
- Zmniejszanie entropii reakcji poprzez zabezpieczenie, blokowanie dostarczanych razem substratów w poprawnej orientacji, niezbędnej do zajścia reakcji. Rozpatrując energetykę rekacji enzymatycznej pod kątem jedynie zmiany entalpii (ΔH‡), efekt ten jest zwykle pomijany. Wiąże się to z faktem, że ma on miejsce tylko dla stanu podstawowego reakcji (substraty) i jego wpływ na właściwą katalizę jest znikomo mały[30][31].
Stabilizacja stanu przejściowego
[edytuj | edytuj kod]Enzymy, w obsługiwanych reakcjach, obniżają ΔG‡ (energię aktywacji) poprzez stabilizację stanu przejściowego (ES*). Stabilizacja tego stanu, a więc redukcja energii, jest o wiele większa niż w analogicznej reakcji zachodzącej spontanicznie, bez udziału enzymów, co jest kluczem do jej przyspieszenia. Jest to możliwe poprzez niezwykle efektywne wykorzystanie efektów oddziaływań elektrostatycznych, w szczególności pomiędzy substratami w stanie przejściowym, a otaczającym je środowiskiem. W reakcji niekatalizowanej, kompleks stanu przejściowego oddziałuje z wodą (z otoczenia) w sposób nieoptymalny z energetycznego punktu widzenia, co zmusza system do pokonania dużej bariery energetycznej (energia atywacji). Enzym, zapewniając ściśle dopasowaną "kieszeń" centrum aktywnego, jest w stanie zapewnić optymalne energetycznie oddziaływanie kompleksu stanu przejściowego z otaczającym je środowiskiem - na przykład minimalizując niepotrzebny, niefaworyzowany energetycznie kontakt z wodą i zamieniając go na energetycznie mniej kosztowny kontakt z mającymi odpowiedni ładunek resztami aminokwasowymi swojego centrum aktywnego. Także inne oddziaływania elektrostatyczne są wspomagane przez enzym, głównie poprzez zapewnienie dokładnie pasującego do rozkładu ładunków na powierzchni kompleksu stanu przejściowego otoczenia (aminokwasów centrum aktywnego)[32]. Dodatkowo, także same zbliżenie do siebie substratów, a także zapewnienie donorów/akceptorów elektronów zachodzącej reakcji, obniża koszta energetyczne jej przebiegu, co nie ma miejsca w reakcji zachodzącej spontanicznie w wodzie.
Dynamika enzymów
[edytuj | edytuj kod]Nowe badania pozwoliły głębiej poznać związek między dynamiką enzymów a mechanizmem ich katalizy[33][34][35]. Dynamika wewnętrzna enzymów jest opisywana jako ruch w obrębie cząsteczki (np. aminokwasy, grupy aminokwasów, regiony pętli, alfa-helis, obszary beta-kartek lub nawet całe domeny) tych biocząsteczek, który może zajść w szerokim przedziale czasowym, wahającym się od kilku femtoskund do sekundy. Pozostałe obszary łańcuchów białkowych tworzą rusztowanie dla funkcjonalnych obszarów cząsteczki oraz umożliwiają ich dynamiczne ruchy, wspomagając w ten sposób katalizę[36][37][38][39]. Białkowe ruchy są istotne w obrębie cząsteczki wielu enzymów, ale czy będą to małe i szybkie ruchy czy też duże i powolne zmiany konformacyjne, zależne jest od typu katalizowanej reakcji w którą zaangażowany jest konkretny enzym. Jakkolwiek taka dynamika prawdopodobnie nie ma wpływu na ogólną szybkość katalizy[40].
Modulacja allosteryczna
[edytuj | edytuj kod]Niektóre enzymy, to enzymy allosteryczne, zmieniające swoją konformację w odpowiedzi na związanie efektora (inhibitora lub aktywatora). Modulacja taka może być bezpośrednia; gdy efektor sam wiąże się z enzymem, lub pośrednia; gdy efektor wiąże się z innym białkiem, lub podjednostką białka, która oddziałowuje z enzymem, zmieniając jego aktywność katalityczną.
Kofaktory, grupy prostetyczne i koenzymy
[edytuj | edytuj kod]
Wiele enzymów potrzebuje dodatkowych składników do uaktywnienia czy osiągnięcia pełnej aktywności. Takie niebiałkowe, dodatkowe składniki enzymów, nazywane są kofaktorami[41]. Enzym bez swojego kofaktora to apoenzym, natomiast wraz z kofaktorem, katalitycznie aktywny enzym to holoenzym.
Kofaktory można podzielić na dwie szerokie grupy: grupy prostetyczne to kofaktory silnie, często kowalencyjnie, związane przez cały czas istnienia enzymu. Zwykle są to cząsteczki nieorganiczne i jony metali (np. kompleksy żelazo-siarkowe, jony cynku - Zn2+), ale także małe cząsteczki organiczne (np. flawiny i hem). Z kolei koenzymy to małe, niebiałkowe cząsteczki organiczne, wiążące się z enzymami tylko na czas reakcji, i przenoszące grupy chemiczne pomiędzy poszczególnymi reakcjami[42]. Koenzymy mogą być traktowane jako kosubstraty, ponieważ są wiązane i uwalniane z enzymów jak substraty i produkty oraz biorą bezpośredni udział w reakcji.
Niektóre źródła nazwę kofaktor przypisują cząsteczkom nieorganicznym, podczas gdy organicznie zwane są koenzymami[43][44].
Grupy prostetyczne
[edytuj | edytuj kod]Silnie związane z enzymem grupy prostetyczne występują zwykle w jego miejscu aktywnym i są bezpośrednio zaangażowane w katalizę oraz nie opuszczają miejsca aktywnego podczas reakcji. Na przykład flawinowe i hemowe kofaktory biorą udział w reakcjach redox.
Większość kofaktorów nie jest kowalencyjnie powiązana z enzymem. Jednakże organiczne grupy prostetyczne mogą się wiązać kowalencyjnie np. pirofosforan tiaminy w dehydrogenazie pirogronianowej.
Koenzymy
[edytuj | edytuj kod]
Koenzymy są małymi cząsteczkami organicznymi transportującymi grupy chemiczne pomiędzy różnymi reakcjami (substratami)[45]. Na przykład koenzymy nikotynamidowe, NAD+ lub NADP+, powodują odłączenie dwóch atomów wodoru od zredukowanych substratów przyjmując na siebie jony wodorkowe z równoczesnym uwolnieniem protonu, grupy acetylowe przenoszone są przez koenzym A, grupy: formylowe, metylowe lub metylenowe przenoszone są z udziałem kwasu foliowego, grupa metylowa może też być przenoszona przez S-adenozylometioninę. Niektóre z koenzymów, takich jak: ryboflawina, tiamina i kwas foliowy, są witaminami.
Skoro koenzymy podlegają zmianom chemicznym w wyniku działania enzymów, można je postrzegać jako specjalną klasę substratów (kosubstratów), czy też substratów wtórnych, wspólnych dla wielu różnych enzymów.
Stężenie koenzymów wewnątrz komórki jest utrzymywane na stałym poziomie, dzięki ich ciągłej regeneracji na różnych szlakach biochemicznych. Np. NADPH jest regenerowany na szlaku pentozofosforanowym.
Termodynamika
[edytuj | edytuj kod]
Tak jak wszystkie katalizatory: enzymy nie zmieniają stanu równowagi reakcji chemicznej, a jedynie przyspieszają jego ustalenie. Zazwyczaj w obecności enzymu reakcja zachodzi w kierunku takim samym, w jakim by zachodziła spontanicznie, jedynie wzrasta jej szybkość. Jednak nieobecność enzymu oznacza, że najbardziej wydajnie (najszybciej) będzie zachodzić reakcja najbardziej faworyzowana energetycznie, co nie zawsze oznacza, że z grupy możliwych reakcji jest to ta sama, która byłaby katalizowana przez enzym. Stąd obecność enzymu, z pośród wielu możliwych reakcji, energetycznie wymusza i faworyzuje jedną z nich.
Jest to termodynamicznie możliwe, gdyż enzymy sprzęgają reakcje niekoniecznie wydajne energetycznie (takie, które spontanicznie zachodziłyby niezwykle wolno) z reakcjami wybitnie energetycznie korzystnymi, tak że taka para reakcji, w sumie, jest energetycznie faworyzowana i korzystna. Przykładem może być sprzężenie reakcji hydrolizy ATP z innymi licznymi reakcjami chemicznymi, wymagającymi do zajścia dużej ilości energii.
Enzymy zwiększają szybkość reakcji zachodzącej w obu kierunkach. Na przykład, anhydraza węglanowa katalizuje swoją reakcję dwukierunkowo w zależności od stosunku stężenia substratów i produktów.
Jednak, gdy w danych warunkach szybkość reakcji zachodzącej w konkretną stronę jest bardzo duża (równowaga mocno przesunięta mocno w jedną ze stron), w praktyce oznacza to katalizę nieodwracalną i reakcja zachodzi tylko w tym jednym kierunku.
Kinetyka
[edytuj | edytuj kod]
Kinetyka enzymów opisuje mechanizmy wiązania substratów przez enzymy oraz ich przekształcania w produkty. Dane wykorzystywane do analizy kinetycznej pochodzą z reakcji enzymatycznych przeprowadzonych w kontrolowanych warunkach umożliwiających śledzenie zmieniających się parametrów w czasie.
W 1902 roku Victor Henri[46] zaproponował kwantytatywną teorię kinetyki enzymów, ale wyniki jego badań okazały się nie przydatne ponieważ zaniedbał on wpływ stężenia jonów wodorowych (pH). Dopiero kilka lat później, w 1909 roku, Peter Lauritz Sørensen zdefiniował logarytmiczną skalę pH oraz zaproponował koncepcję roztworów buforowych[47]. Niemiecki chemik Leonor Michaelis i jego kanadyjska współpracowniczka odbywająca staż podoktorski Maud Leonora Menten, powtórzyli wtedy eksperymenty, które przeprowadzał Henri i zaproponowali prosty model kinetyki aktywności enzymatycznej znany jako kinetyka Henri-Michaelis-Menten (znany również jako kinetyka Michaelis-Menten[48]). Ich dzieło rozwinęli później G. E. Briggs i J. B. S. Haldane, których równiania kinetyki aktywności enzymatycznej są do dziś używane w nie zmienionej formie[49].
Głównym założeniem jakie podał Henri, był dwuetapowy przebieg reakcji katalizowanej przez enzymy. W pierwszym etapie substraty wiążą się odwracalnie z enzymem, tworząc kompleks enzym-substrat (ES), nazywany czasem kompleksem Michaelisa. Następnie podczas drugiego etapu reakcji, enzym katalizuje zajście chemicznych zmian substratów i uwalnia produkty.

Enzymy mogą katalizować powyżej kilku milionów reakcji na sekundę, gdzie te same reakcje bez ich obecności mają czasy połowicznej przemiany substratu, tzn. czas w jakim pod nieobecność enzymu, połowa dostępnego substratu zostanie zużyta, kilkanaście rzędów wielkości dłuższy (21 rzędów wielkości to najwyższe znane przyspieszenie reakcji enzymatycznej, z tryliona lat do dziesiatek milisekund[50]). Przykładem jest reakcja katalizowana przez dekarboksylazę orotydyno 5'-monofosforanu. Czas połowicznej przemiany reakcji nieenzymatycznej wynosi 78 mln lat, a czas połowicznej przemiany tej samej reakcji, ale z udziałem dekarboksylazy orotydyno 5'-monofosforanu, wynosi 25 milisekund[51]. Bezpośrednia szybkość z jaką działają enzymy zależy od stężenia substratów w roztworze (dodatkowo szybkość enzymów zależy od ogólnych parametrów fizyko-chemicznych środowiska, tak jak w przypadku aktywności każdego białka, patrz: Aktywność a parametry środowiska). By znaleźć maksymalną szybkość reakcji katalizowanej enzymatycznie, określa się ilość tworzonego produktu w funkcji czasu, przy stopniowym zwiększaniu stężenia substratu, ale jednoczesnym zachowaniu innych warunków, do momentu, w którym dalszy wzrost jego stężenia nie powoduje dalszego przyśpieszania reakcji. Szybkość początkowa w takim eksperymencie (V0) rośnie do wartości maksymalnej, Vmax) i nie przekracza jej mimo dalszego wzrostu stężenia substratu. W chwili osiągnięcia stanu równowagi (reakcji katalizowanej i reakcji do niej przeciwnej), nie obserwujemy zmiany netto stężenia substratów i produktów. Zależność szybkości reakcji od stężenia substratu dla prostej reakcji wg kinetyki Michaelisa-Menten, przy założeniu, że kompleks ES jest koniecznym etapem pośrednim procesu katalitycznego, przedstawia krzywa Michaelisa-Menten. Wysycanie enzymu substratem (zbliżanie się do szybkości maksymalnej) oznacza, że maleje liczba wolnych cząsteczek enzymu, gdyż rośnie ilość tych związanych w kompleksie z substratem (ES). Osiągana asymptotycznie maksymalna szybkość (Vmax) reakcji enzymatycznej, oznacza, że praktycznie wszystkie miejsca aktywne enzymu (wolne enzymy) zostają wysycone substratem, a wówczas suma ilości (stężenie) kompleksów ES jest miarą ilości samego enzymu. Niemniej jednak, Vmax jest tylko jedną stałą kinetyczną opisującą enzym. Inną jest ilość substratu (stężenie) potrzebne do osiągnięcia ustalonej szybkości reakcji. Zwykle używa się do tego celu stałej Michaelisa-Menten (Km), która jest takim stężeniem substratu przy którym szybkość reakcji osiąga połowę swojej maksymalnej wartości. Każdy enzym ma swoją charakterystyczną wartość Km dla danego substratu, która charakteryzuje również siłę wiązania substratu w miejscu aktywnym enzymu. Inną użyteczną stałą jest szybkość katalizy (kcat), która jest liczbą obrotów jednego miejsca aktywnego enzymu w czasie jednej sekundy (liczba reakcji przeprowadzana przez jedno miejsce aktywne w czasie jednej sekundy).
Innymi jednostkami używanymi do opisu aktywności enzymów są: jednostka enzymu (U), czyli taka ilość enzymu, która katalizuje przemianę 1 μmola substratu w ciągi 1 minuty w temperaturze 30 °C i określonym pH oraz katal (kat), czyli ilość enzymu katalizująca przemianę 1 mola substratu w ciągu 1 sekundy. Dodatkowo dla niektórych enzymów używa się jednostek umownych, definiowanych przez zastosowany pomiar aktywności. Na przykład przyrost absorbancji światła o danej długości fali w danej jednostce czasu, gdy wynikiem reakcji enzymatycznej jest barwny produkt. W preparatyce biochemicznej dodatkowo używa się aktywności właściwej, która określa liczbę jednostek enzymu (lub jednostek umownych aktywności) przypadających na 1 mg białka (w danym preparacie)[52].
W sytuacji, gdy stężenie substratu znacznie przewyższa Km, szybkość katalizy jest równa kkat, liczbie obrotów. Jednak w warunkach fizjologicznych, większość enzymów nie jest wysycona substratem, zatem stosunek [S]/Km mieści się w granicach od 0,01 do 1,0. Gdy stężenie substratu ([S]) jest dużo mniejsze od Km ([S<<Km), wówczas szybkość katalizowanej reakcji jest znacznie mniejsza od kkat ponieważ większość miejsc aktywnych nie jest zajęta. Do scharakteryzowania kinetyki enzymów w tych warunkach służy równanie V0=kcat/Km, powstałe z połączenia dwóch innych równań: V0=k2 [ES] oraz [ES]=[E][S] / Km. W przedstawionej sytuacji, gdy [S]<<Km to stężenie wolnego enzymu jest niemal równe całkowitemu stężeniu enzymu. W tych warunkach stosunek kcat/Km jest stałą szybkości oddziaływania S i E i może być użyty jako miara wydajności katalitycznej. Ponieważ stosunek kcat/Km odzwierciedla zarówno powinowactwo chemiczne jak i zdolność katalityczną, jest używany do porównywania różnych enzymów względem siebie lub preferencji jednego enzymu do różnych substratów. W sytuacji, gdy szybkość tworzenia produktu (kkat) jest znacznie szybsza niż szybkość dysocjacji kompleksu ES, wartość kkat/Km zbliża się do wartości stałej tworzenia kompleksu ES. Z tego wynika, że górna granica wartości kkat/Km jest ograniczana przez szybkość tworzenia kompleksu ES, która nie może być większa niż częstość z jaka spotykają sie enzym i jego substrat na skutek dyfuzji. Częstość ta mieści się w granicach od 108 do 109 (M-1 s-1) i jest to jednocześnie górna granica kkat/Km. W tym punkcie, każde zderzenie enzymu z jego substratem będzie powodowało katalizę i szybkość formowania produktu nie jest limitowana przez szybkość reakcji ale przez szybkość dyfuzji. Enzymy posiadające tą własność nazywamy katalitycznie perfekcyjnymi lub kinetycznie perfekcyjnymi. Przykładami takich enzymów są: izomeraza triozofosforanowa, anhydraza węglanowa, esteraza acetylocholinowa, katalaza, fumaraza, β-laktamaza i dysmutaza ponadtlenkowa[3].
Niektóre enzymy działają z kinetyką szybszą od teoretycznej szybkości dyfuzji. Jest to możliwe, gdyż wiązanie substratów przez enzym (krok limitowany przez szybkość dyfuzji) i tworzenie kompleksu ES są wspomagane dodatkowymi efektami niezależnymi od samej dyfuzji. Niektóre białka enzymatyczne aktywnie przeorientowują substraty za pomocą pól elektrycznych generowanych przez cząsteczki (reszty) dipolowe. Inne opierają się na kwantowo-mechanicznym zjawisku tunelowym, za pomocą którego proton lub elektron może pokonać barierę potencjału o energii większej niż energia cząstki, chociaż dla protonu ten model wciąż jest przedmiotem dyskusji[53][54]. Jakkolwiek kwantowo-mechaniczne zjawisko tunelowania zostało zaobserowane w tryptaminie[55]. Sugeruje to, że przynajmniej w niektórych przypadkach, kataliza enzymatyczna to nie "przeskakiwanie przez barierię", a raczej "przebijanie się przez nią".
Aktywność a parametry środowiska
[edytuj | edytuj kod]Ogólna aktywność enzymów, podobnie jak wszystkich białek, jest zależna od parametrów fizyko-chemicznych środowiska: temperatury, pH, siły jonowej i innych. Maksimum aktywności enzymu leży w pewnym optymalnym zakresie danego parametru środowiskowego. W zależności od enzymu, położenie optimum może być różne, a jego zakres szerszy lub węższy. Także ogólny kształt wykresu zależności aktywności danego enzymu od parametru środowiska jest różny dla różnych enzymów, w zależności od ich pochodzenia, budowy itp. Dla czynników środowiska bezpośrednio wpływających na strukturę drugorzędowa białka (i wyższe organizacje struktury), jak temperatura czy pH, charakterystyczny jest gwałtowny spadek aktywności enzymów poza optimum (lub po przekroczeniu go, w przypadku temperatury), związany z denaturacją enzymów. W zależności od enzymu, taka denaturacja może być odwracalna lub nie. Parametry środowiska mogą stanowić podstawę kontroli aktywności enzymów (patrz: Kontrola aktywności)[1].
Temperatura
[edytuj | edytuj kod]
Szybkość reakcji enzymatycznych wzrasta wraz ze wzrostem temperatury. Wiąże się to z podwyższeniem energii kinetycznej systemu (cząstek) i większą częstotliwością ich zderzeń w roztworze. Wielkość wzrostu aktywności enzymatycznej w zależności od temperatury opisuje współczynnik temperaturowy Q10, określający jak zmieni się szybkość reakcji przy wzroście temperatury o 10 °C:
- Q10=v(t+10) / vt
Wpływ temperatury na aktywność enzymów nie jest prostą zależnością. Aktywność rośnie wraz ze wzrostem temperatury, jednakże tylko w takim zakresie temperatury, w którym enzym pozostaje stabilny. Po przekroczeniu temperatury krytycznej, postępuje denaturacja termiczna enzymów i aktywność gwałtownie spada. Przeciętnie szybkość reakcji enzymatycznych wzrasta dwukrotnie przy wzroście o każde 10 °C w zakresie temperatur nie denaturujących struktury enzymu (Q10=2). Zatem także parametr Q10 ma zastosowanie tylko w niedenaturującym zakresie temperatur i jest on charakterystyczny dla danego enzymu, i zależny od energii aktywacji katalizowanej reakcji. W temperaturze optymalnej aktywność enzymu jest największa. Obserwowana temperatura optymalna jest wypadkową dwóch procesów: wzrostu szybkości reakcji związanego ze wzrostem energii kinetycznej oraz zwrotu szybkości denaturacji termicznej enzymu powyżej krytycznej temperatury. Gdy drugi składnik zaczyna przeważać, następuje spadek aktywności. Większość enzymów ma optimum temperaturowe w zakresie 30-45 °C i nieodwracalnie denaturuje oraz traci aktywność w temperaturach wyższych niż 60 °C. Jednak w przypadku organizmów termofilnych (np. bakterii ze źródeł termalnych) ich enzymy zachowują aktywność i osiągają maksymalną szybkość w podwyższonych temperaturach.
Większość enzymów ulega powolnej denaturacji nawet w temperaturach optymalnych i niższych niż krytyczna. Zależy to od natury samego enzymu, stopnia jego oczyszczenia (w preparatach), a także od pH, siły jonowej i pozostałych parametrów. Najwyższa temperatura w której jeszcze nie zachodzi termiczna dezaktywacja enzymu w danych warunkach to tak zwana termostabilność enzymu[52].
pH
[edytuj | edytuj kod]Większość enzymów ma także swoje optymalne pH. Optimum pH, obok optimum temperaturowego, to drugi najważniejszy parametr środowiska charakteryzujący aktywność enzymów. Wpływ pH na aktywność enzymów wiąże się z faktem, że enzymy jako białka posiadają wiele aminokwasów ulegających jonizacji, a aminokwasy centrum aktywnego często mogą pełnić swoją rolę tylko w określonym stanie jonizacji. Jonizacja samych substratów i kompleksów ES także ma swój udział w aktywności. Również oddziaływania enzym-substrat mogą mieć charakter jonowy, zależny od jonizacji. Od stopnia jonizacji pewnych grup zależy także ogólna konformacja cząsteczki enzymu, zapewniająca mu pełną aktywność, a w za kwaśnym czy zbyt zasadowym środowisku enzym ulegnie denaturacji.
Wykres zależności aktywności enzymu od pH ma najczęściej kształt dzwonowaty, czyli enzym ma największą aktywność w swoim optymalnym pH i aktywność ta spada wraz z oddalaniem się od tego punktu (np. trypsyna). Niektóre jednak enzymy mają właściwie stała aktywność w szerokim zakresie pH (np. papaina) lub są wrażliwe tylko na niskie pH, zachowując aktywność w wysokim (jak esteraza cholinowa) lub na odwrót. Zależność aktywności od pH bada się w stałej temperaturze przy maksymalnym wysyceniu enzymu substratem[52].
Kontrola aktywności
[edytuj | edytuj kod]Istnieje kilka głównych szlaków kontroli aktywności enzymów w komórkach:
- Sama produkcja enzymu na etapach translacji, jak i wcześniejszej transkrypcji, podlega ścisłej kontroli i może być zwiększana (uruchamiana) lub zmniejszana (zatrzymana) przez komórkę w odpowiedzi na zmiany w środowisku zewnętrznym komórki, tak samo jak kontrolowana jest w ten sposób produkcja każdego innego białka.
- Sortowanie i rozdzielanie enzymów do docelowych kompartmentów (także poza komórkę) i utrzymywanie ich tam, jest sposobem na kontrolę ich aktywności w przestrzeni. Enzymy działają wtedy na szlakach metabolicznych umiejscowionych w różnych przedziałach komórkowych, nie intereferują z innymi szlakami, a ich aktywność może być także szybko zmieniona poprzez globalną zmianę charakterystyki środowiska kompartmentu w którym się znajdują (np. obniżenie pH w lizosomie). Różne kompleksy enzymatyczne zawarte w różnych przedziałach komórkowych, a co za tym idzie podział syntezy na etapy, jak również oddzielenie rozkładu, umożliwia dokładną kontrolę szlaku metabolicznego na każdym etapie przez różne czynniki[56].
- Aktywność enzymów może być regulowana przez specjalne cząsteczki - inhibitory (patrz: Inhibicja) i aktywatory. Jest to najbardziej bezpośredni sposób modulacji ich aktywności.
- Enzymy mogą być regulowane poprzez ich modyfikacje potranslacyjne. Modyfikacje takie mogą obejmować kowalencyjne zmiany struktury cząsteczki, np. dodawanie dodatkowych grup funkcyjnych: fosforylacja, mirystylacja czy glikozylacja. Przykładowo związanie insuliny z odpowiednim receptorem uwalnia kaskadę fosforylacji wielu enzymów i w rezultacie syntezę glikogenu[57]. Innym przykładem potranslacyjnych modyfikacji enzymów jest modyfikacja łańcucha polipeptydowego enzymu poprzez jego skracanie, przycinanie. Przykładowo chymotrypsyna, proteolityczny enzym trawienny, produkowany w trzustce w formie nieaktywnej, jako chymotrypsynogen jest transportowany w tej formie do żołądka. Tam następuje jego aktywacja poprzez skrócenie łańcucha polipetydowego i uwolnienie aktywnej cząsteczki dojrzałego enzymu. Produkcja enzymu w formie proenzymu zabezpiecza trzustkę i inne tkanki, zanim dotrze do jelita, przed strawieniem. Ten typ nieaktywnego prekursora enzymu zwany jest jako zymogen. Czasami taka modyfikacja jest wieloetapowa, mówi się wtedy o preproenzymach[58]. Przycinanie proenzymu do formy aktywnej zachodzi z udziałem innych enzymów lub czasami sam enzym jest w stanie katalizować reakcję aktywacji swoich form proenzymatycznych[59].
- Niektóre enzymy mogą stać się aktywne (nieaktywne), gdy znajdą się w innym środowisku, np. po przeniesieniu ze środowiska cytoplazmy o właściwościach redukujących do środowiska przestrzeni peryplazmatycznej o właściwościach utleniających, z wysokiego pH do niskiego pH, itp. Przykładem może być hemaglutynina - glikoproteina o właściwościach antygenowych - na powierzchni wirusów grypy, która zmienia konformację, gdy znajdzie się w kwaśnym środowisku pęcherzyka komórkowego gospodarza, powodując aktywację wirusa[60].
Inhibicja
[edytuj | edytuj kod]

Aktywność wielu enzymów może być hamowana przez różne typy inhibitorów. Inhibicja taka może być odwracalna lub nieodwracalna. Ostatnia ma miejsce wtedy, gdy cząsteczki inhibitora wiążą się z enzymem trwale (np. kowalencyjnie), co doprowadza do sytuacji zablokowania aktywności danej cząsteczki enzymu na stałe[3].
Typy inhibicji
[edytuj | edytuj kod]W inhibicji kompetycyjnej inhibitor i substrat współzawodniczą o miejsce aktywne cząsteczki enzymu. Związanie przez enzym cząsteczki inhibitora uniemożliwia zatem związanie substratów (i vice versa) i kompleks enzym-inhibitor (EI) jest enzymatycznie nieaktywny. Zazwyczaj inhibitor kompetycyjny jest strukturalnie bardzo podobny (lub ma podobny motyw bezpośrendnio wiążący się do centrum aktywnego) do prawdziwego substratu dla określonego enzymu. Na przykład metotreksat jest inhibitorem kompetycyjnym dla enzymu reduktazy dihydrofolianu, który katalizuje redukcję dhydrofolianu do tetrahydrofolianu i obie substancje są strukturalnie bardzo zbliżone. Przyłączenie inhibitora uniemożliwia związanie substratu i odwrotnie, dzięki czemu poprzez regulację stężenia inhibitora możliwa jest kontrola szybkości reakcji. Stałą dysocjacji inhibitora kompetycyjnego można opisać równaniem:
W inhibicji kompetycyjnej maksymalna szybkość reakcji (Vmax) nie zmienia się i może być osiągnięta poprzez zwiększenie stężenia substratów, które przezwycięży inhibicję. Zmianie natomiast ulega pozorna wartość Km, nowa wartość Km, zwana Kmapp (pozorna stała Michaelisa) jest równa:
gdzie [I] - stężenie inhibitora, Ki stała dysocjacji dla kompleksu enzym-inhibitor
Zatem wzrost [I] pociąga za sobą wzrost Kmapp.
Inhibicja akompetycyjna
[edytuj | edytuj kod]W inhibicji akompetycyjnej inhibitor nie może się wiązać do wolnego enzymu, a jedynie do kompleksu enzym-substrat (ES). Utworzony kompleks enzym-inhibitor-substrat (EIS) jest enzymatycznie nieaktywny i reakcja nie może być kontynuowana. Ten typ inhibicji jest dość rzadki i może dotyczyć niektórych enzymów multimerycznych (wielopodjednostkowych).
Inhibitory niekompetycyjne mogą się wiązać do wolnego enzymu, jednak nigdy do jego miejsca aktywnego, wobec tego nie konkurują z substratami, które także mogą się przyłączyć do powstałego kompleksu. Oba możliwe kompleksy, enzym-inhibitor (EI) i enzym-inhibitor-substrat (EIS) są enzymatycznie nieaktywne. Ponieważ wiązanie inhibitora jest całkowicie niezależne od substratu, co oznacza, że większe stężenie substratu nie wpływa na obniżenie oddziaływań z inhibitorem (w przeciwieństwie do inhibicji kompetycyjnej), obie wartości opisujące reakcję enzymatyczną: Km i Vmax, zmieniają się.
Inhibicja mieszana
[edytuj | edytuj kod]Ten typ inhibicji przypomina inhibicję niekompetycyjną, z wyjątkiem występowania kompleksu enzym-inhibitor-substrat (EIS), mającego znikomą aktywność enzymatyczną.
Inhibicja nieodwracalna
[edytuj | edytuj kod]Inhibitory nieodwracalne reagują z enzymem, wiążac się kowalencyjnie, a zatem nieodracalnie, do jego łańcuchów białkowych. Taka inhibicja trwale unieczynnia daną cząsteczkę enzymu. Do inhibitorów tego typu należy eflornityna, lek stosowany w pasożytniczej chorobie - śpiączce afrykańskiej[62]. Również penicylina i aspiryna mają podobny mechanizm działania. Inhibitory te wstępnie, jeszcze nie kowalencyjnie, wiążą się z miejscami aktywnymi enzymów, po czym dopiero aktywność enzymu przekształca je w reaktywne formy, które wiążą się nieodwracalnie z jedną lub więcej resztami aminokwasowymi, blokując enzym.
Regulacja aktywności enzymów przez inhibitory
[edytuj | edytuj kod]W wielu reakcjach inhibitory biorą udział w mechanizmie regulacji aktywności enzymatycznej na drodze sprzężenia zwrotnego. Jeśli enzym produkuje jedną substancję ponad potrzeby komórki, to ta substancja może stać się inhibitorem dla tego enzymu, co zmniejsza lub całkowicie hamuje aktywność enzymu, co z kolei zmniejsza stężenie produktu. Taka regulacja jest formą ujemnego sprzężenia zwrotnego. Enzymy, które poddane są tego typu regulacji, to często układy wielopodjednostkowe, posiadające kilka miejsc aktywnych dla substancji regulatorowych. Dla takich enzymów, wykres prędkości katalizowanej reakcji w zależności od stężenia substratu, nie ma przebiegu hiperbolicznego, ale sigmoidalny (kształt litery S).
Funkcje biologiczne
[edytuj | edytuj kod]Enzymy są podstawą istnienia życia, jako że są niezbędne do zajścia niemalże wszystkich reakcji chemicznych z szybkością i wydajnością znacząco wysoką dla procesów biologicznych. Bez enzymów większość reakcji zachodziła by zbyt wolno lub zbyt mało wydajnie, by miało to zauważalne w czasie znaczenie.
Większość enzymów, poprzez swój udział w reakcjach katabolicznych i anabolicznych, definiuje metabolizm komórki czy organizmu (elementami metabolizmu komórkowego są także enzymy zewnątrzkomórkowe, np. enzymy trawienne). Enzymy metaboliczne działają zwykle w grupach (kompleksach), w szeregu sekwencyjnie następujących po sobie reakcji, określanych mianem szlaków metabolicznych. Szlaki te często wykorzystują wspólne produkty pośrednie reakcji, przebiegają równolegle, prowadzą do tego samego produktu lub od tego samego substratu w skomplikowanej sieci zależności reakcji enzymatycznych. Dodatkowo regulacja tych szlaków, a także sygnalizacja wewnątrz- i zewnątrzkomórkowa także są zależne od enzymów.
Enzymy biorą udział także w reakcjach niemetabolicznych. Na przykład hydrolizująca ATP miozyna bierze udział w skurczach mięśni[63], a inne motory molekularne o aktywności enzymatycznej są odpowiedzialne za ruch na poziomie wewnątrzkomórkowym.
Enzymy są także ważnymi składnikami interakcji między organizmami czy komórkami. Leukocyty używają enzymów do zwalczania patogenów, z kolei bakterie patogenne używają ich podczas infekcji i do obrony przed elementami systemu immunologicznego. Także wirusy używają enzymów (kodowanych przez swój materiał genetyczny lub enzymów gospodarza) do swojej replikacji (np. odwrotna transkryptaza wirusa HIV), a także podczas infekcji i opuszczania komórek gospodarza (np. neuraminidaza wirusa grypy). Enzymy są także składnikami jadów i toksyn, używanych i wytwarzanych przez wiele organizmów.
Enzymy pełnią także bardziej wyszukane funkcje, wszędzie tam, gdzie potrzebna jest wydajna, specyficzna kataliza chemiczna. Na przykład bioluminescencja u świetlików wywołana jest przez enzym lucyferazę, a strzel bombardier, chrząszcz z rodzaju Brachynus w celach obronnych wykorzystuje peroksydazę do rozkładu nadtlenku wodoru, dzięki czemu może bronić się przed zagrożeniem, wyrzucając w jego stronę strumień wrzącej cieczy pod ciśnieniem[1].
Udział w chorobach
[edytuj | edytuj kod]
Ponieważ aktywność enzymatyczna i jej ścisła kontrola są elementem homeostazy organizmu, defekt w jednym nawet enzymie (mutacja zaburzająca funkcję, nadprodukcja, za mała produkcja, delecja) czy mechanizmach kontroli jego aktywności, może prowadzić do stanu chorobowego lub śmierci organizmu.
Wrodzone uszkodzenia genów kodujących enzymy lub elementów kontroli ich aktywności mogą prowadzić do szeregu nieuleczalnych (albo leczonych wyłącznie objawowo) genetycznych chorób metabolicznych (tj. związanych z konkretnym szlakiem metabolicznym lub przemianami metabolicznymi). Należą do nich bloki metaboliczne (w tym choroby spichrzowe, złogowe), w których wskutek zakłócenia (braku) pracy enzymu, nie są metabolizowane konkretne substancje, co prowadzi do ich niedoboru lub gromadzenia się półproduktów, które często są toksyczne. Do takich chorób należą np. galaktozemia czy fenyloketonuria. Ponieważ końcowe produkty danego szlaku są często inhibitorami enzymów jego wczesnych etapów, ich brak tym bardziej może kumulować szkodliwe półprodukty, produkowane przez nieregulowane enzymy (jak ma to miejsce w zespole Lescha-Nyhana[64]). Także otyłość może być powodowana przez ogólnoustrojowe zaburzenie pracy enzymów metabolicznych[65][66].
Jedne enzymy często utrzymują aktywność innych na odpowiednim poziomie (np. poprzez rozkład ich nadmiaru). Gdy te pierwsze w jakiś sposób nie działają optymalnie, niekontrolowane już odpowiednio przez nie enzymy, mogą działać na szkodę własnych komórek organizmu. Może dojść do autolizy komórek i w następstwie do uszkodzenia tkanek. Przykładem jest tu jedna z form rozedmy płuc powodowana niekontrolowana aktywnością elastazy niszczącej strukturę tkanki[64].
Z kolei zaburzenie pracy enzymów uwikłanych w naprawę materiału genetycznego może prowadzić do śmierci komórek lub wykształcenia się nowotworów.
W chorobach wirusowych i bakteryjnych aktywność i inwazyjność patogenów często zależy od aktywności ich lub gospodarza enzymów. Także niektóre toksyny to enzymy ingerujące w metabolizm lub o aktywności litycznej, trawiennej.
Konwencja nazewnictwa
[edytuj | edytuj kod]Zwykle nazwy enzymów pochodzą od substratów przetwarzanych w reakcji lub od samej reakcji przez nie obsługiwanych, do których dodaje się przyrostek -aza. Przykładowo celulaza to enzym rozkładający celulozę, a ligaza DNA to enzym ligujący cząsteczki DNA. Często nazwa jest dwuczłonowa, gdzie pierwszy człon określa reakcję, a drugi jej substrat, np. dehydrogenaza alkoholowa. Stąd różne enzymy obsługujące takie same reakcje lub przetwarzające te same substraty, nazywane izoenzymami, mimo tego, że chemicznie są cząsteczkami o różnym składzie aminokwasowym i właściwościach, będą miały takie same nazwy. Czasami nazwa enzymu nie pochodzi od reakcji obsługiwanej w warunkach fizjologicznych, in vivo, ale od jego aktywności in vitro. Przykładem jest izomeraza glukozowa, wykazująca in vitro aktywność izomeracji glukozy we fruktozę, ale in vivo będąca izomerazą ksylozową. Także zamiast od nazwy reakcji, enzymy są niekiedy nazywane od ogólnego procesu, jaki przeprowadzają, jak np. gyraza DNA (właściwie topoizomeraza II), której nazwa pochodzi od greckiego słowa γύρος, czyli obracać i przyrostka -aza. Podczas przeprowadzanej reakcji enzym ten rzeczywiście musi obrócić nić DNA[67].
Z kolei niektóre cząsteczki biologiczne, mimo nazwy z przyrostkiem -aza, nie są enzymami lub definiowana przez nazwę aktywność nie jest aktywnością enzymatyczną. Przykładem są flipazy, których główna aktywność przenoszenia lipidów pomiędzy monowarstwami dwuwarstwy (proces flip-flop) nie jest aktywnością enzymatyczną (jest to proces fizyczny). Flipazy niemniej jednak mogą mieć aktywność enzymatyczną (np. ATPazową) związaną lub nie z ich funkcją jako transporterów lipidów.
Duża część enzymów jest nazywana zwyczajowo, historycznie, jak np. pepsyna (nazwa pochodząca od greckiego słowa pepsis oznaczającego trawienie, w języku polskim funkcjonuje także nazwa podpuszczka) czy papaina (enzym z owocu papai). Nazwy niektórych enzymów powstały także poprzez dodanie przyrostka -zym (tego samego co w słowie enzym) do ogólnej funkcji cząsteczki lub opisu, skrótu opisowego. Przykładami są lizozym (od lizujących właściwości wobec bakterii) czy granzym (ang. granule-associated enzyme, czyli enzym związany z ziarnistościami).
Enzymy restrykcyjne są nazywane według własnej nomenklatury. Ich nazwa składa się z litery nazwy rodzajowej i dwóch pierwszych liter nazwy gatunkowej mikroorganizmu-źródła enzymu, pisanymi kursywą, po których występują cyfry arabskie lub litery oznaczających szczep mikroorganizmu. Nazwa zakończona jest cyfrą rzymską wskazującą, jako który w kolejności chronologicznej został wyizolowany z danego organizmu dany enzym restrykcyjny. Na przykład EcoRI, to pierwszy wyizolowany enzym restrykcyjny z bakterii Escherichia coli, szczep RY13.
Część enzymów jest natomiast bardziej znana pod skrótami pochodzącymi od ich pełnych nazw, jak RuBisCO (właściwie karboksylaza oksygenaza rybulozo-1,5-bisfosforanu, w ang. ribulose-1,5-bisphosphate carboxylase/oxygenase), albo od skrótu nazwy opisowej, jak BACE2 (z ang. beta-site APP-cleaving enzyme 2, czyli enzym tnący prekursorowe białko amyloidu 2).
W celu uregulowania i ujednoznaczenia nazewnictwa enzymów, Komitet Nazewnictwa (ang. Nomenclature Committee) Międzynarodowej Unii Biochemicznej i Biologii Molekularnej (ang. International Union of Biochemistry and Molecular Biology) w latach 1956-1972 opracował dla nich nomenklaturę naukową - numer EC. Według niej, każdy enzym jest opisany przez ciąg czterech segemntów cyfr, oddzielonych od siebie kropką, poprzedzonych literami "EC" (Enzyme Commission lub Enzyme Catalogue): EC xx.xx.xx.xx.
Pierwsze cyfry dzielą enzymy, według mechanizmu reakcji przez nie obsługiwanych, na sześć głównych klas[68][69]:
- EC 1 oksydoreduktazy: katalizują reakcje utleniania i redukcji
- EC 2 transferazy: przenoszą grupy funkcyjne
- EC 3 hydrolazy: katalizują hydrolizę różnych wiązań
- EC 4 liazy: rozcinają różne wiązania na drodze innej niż hydroliza czy utlenianie
- EC 5 izomerazy: katalizują zmiany izomeracyjne cząsteczek
- EC 6 ligazy: łączą cząsteczki wiązaniami kowalencyjnymi.
Zastosowanie przemysłowe
[edytuj | edytuj kod]Enzymy są stosowane w przemyśle chemicznym, spożywczym i innych, głównie jako niezwykle specyficzne, bezpieczne w użyciu katalizatory. Jakkolwiek ich wadą jest wrażliwość na skrajne warunki (np. temperatura, pH), niestabilność w środowiskach innych niż wodne (np. rozpuszczalników organicznych) oraz stopniowa degradacja podczas użytkowania. Także wysoka specyficzność, istotna z punktu biologicznego, w przemyśle jest ograniczeniem ich uniwersalności. Stąd, tak zwana inżynieria białka jest dynamicznie rozwijającą się dziedziną nauki, zajmującą się badaniem i projektowaniem enzymów o nowych właściwościach lub poprawionej wydajności czy stabilności. Aktualne podejście do tego zagadnienia to ukierunkowane projektowanie lub ewolucja in vitro[70][71]. Obecnie enzymy produkowane są na skalę przemysłową, głównie z zastosowaniem mikroorganizmów modyfikowanych genetycznie[72].
| Zastosowanie | Używane enzymy | Cel | ||
Przemysł piekarniczy  |
Grzybowe alfa-amylazy | Katalityczne przyspieszenie rozkładu skrobii z mąki na cukry. Te z kolei są wykorzystywane przez drożdże piekarnicze zawarte w cieście, wskutek czego produkowany jest dwutlenek węgla, spulchniający ciasto. Używane w produkcji pieczywa białego, bułek itp. | ||
| Proteazy | Producenci ciastek używają ich do obniżenia poziomu białka w mące. | |||
| Jedzenie dla niemowląt | Trypsyna | Do wstępnego nadtrawiania jedzenia. | ||
Browarnictwo, gorzelnictwo i przemysł winiarski  |
Enzymy jęczmienne uwalniane są podczas przyrządzania zacieru przy produkcji piwa. | Rokładają one skrobię i białka, uwalniając cukry proste i aminokwasy, służące następnie drożdżom w fermentacji. | ||
| Przemysłowo produkowane enzymy jęczmienne i drożdżowe | Używane jako substytuty naturalnych | |||
| Amylazy, glukanazy, proteazy | Rozkładają polisacharydy i białka zawarte w słodzie czy zacierze. | |||
| Betaglukozydaza | Poprawia proces filtrowania. | |||
| Amyloglukozydaza | Produkcja trunków niskokalorycznych. | |||
| Proteazy | Usuwają zmętnienia produktu. | |||
| Soki owocowe | Celulazy, pektynazy | Klarują soki (rokład celulozy i pektyn). | ||
Mleczarstwo  |
Podpuszczka, izolowana z żołądków młodych przeżuwaczy (cielęta, owce). | Produkcja serów, używane do hydrolizy białek. | ||
| Enzymy pochodzenia mikrobiologicznego | Zastępują te pochodzenia zwierzęcego. | |||
| Lipazy | Stosowane w produkcji sera Roquefort by usprawnić ich dojrzewanie. | |||
| Laktazy | Rozkład laktozy do glukozy i galaktozy. | |||
| Zmiękczacze mięsa | Papaina | Rozmiękczanie mięsa przy gotowaniu. | ||
Przemysł skrobiowy
|
Amylazy, amyloglukozydazy i glukoamylazy | Rozkład skrobii do glukozy i produkcja cukrów konwertowanych. | ||
| Izomeraza glukozowa | Konwersja glukozy we fruktozę przy produkcji wysokofruktozowych syropów ze skrobii. Syropy te są używane do słodzenia i są słodsze niż sacharoza, a mniej kaloryczne. | |||
Przemysł papierniczy |
Amylaza, ksylanaza, celulaza i ligninaza | Amylazy rozkładają skrobię w celu zmniejszenia lepkości, wspomagają regulację gramatury papieru i jego pokrycia. Ksylanazy redukują potrzebę wybielania. Celulazy wygładzają włókna, wspomagają schnięcie oraz usuwanie pigmentów (przy przeróbce makulatury. Ligninazy degradują ligniny i wspomagają zmiękczanie papieru. | ||
Przemysł biopaliwowy |
Celulaza | Używana do rozkładu celulozy do cukrów, które mogą być wykorzystane w procesie fermentacji przy produkcji etanolu celulozowego. | ||
| Ligninaza | Używana do rozkładu ligniny | |||
| Biodetergenty | Proteazy, pierwotnie pozyskiwane z wydzielających je na zewnątrz komórek bakterii. | Używane w praniu wstępnym i namaczniu, a także podczas prania zasadniczego, gdzie wspomagają usuwanie plam pochodzenia białkowego z ubrań. | ||
| Amylaza | Do usuwania plam ze skrobii. | |||
| Lipaza | Wspomagają usuwanie plam z tłuszczu. | |||
| Celulaza | Used in biological fabric conditioners. | |||
| Płyny do mycia soczewek kontaktowych | Proteazy | Do odbiałczania szkieł i zapobiegania zakażeniom. | ||
| Przemysł gumowy | Katalaza | Do wytwarzania tlenu z nadtlenków, by przetwarzać lateks w gumy spienione. | ||
| Przemysł fotograficzny | Proteazy | Rozkład żelatyny pokrywających filmy fotograficzne przy odzyskiwaniu z nich srebra. | ||
Biologia molekularna, biochemia i pokrewne, a także analityka laboratoryjna i kliniczna  |
M.in. enzymy restrykcyjne, ligazy DNA, polimerazy DNA i RNA, proteazy, DNazy, RNazy, enzymy szlaków metabolicznych i inne. | Używane m.in. w manipulacjach materiałem genetycznym, badaniach metabolizmu, proteomice, w diagnozowaniu klinicznym i kryminalnym, jako markery i indykatory, oraz wiele innych. |
Przypisy
[edytuj | edytuj kod]- ↑ a b c Solomon, Berg, Martin, Villee: Biologia. Warszawa: MULTICO Oficyna Wydawnicza, 1998. ISBN 83-7073-090-6.
- ↑ Bairoch A. The ENZYME database in 2000. „Nucleic Acids Res”. 28, s. 304–305, 2000. PMID 10592255.
- ↑ a b c d Berg Jeremy M., Tymoczko John L., Stryer Lubert: Biochemia. Warszawa: Wydawnictwo Naukowe PWN, 2007. ISBN 978-83-01-14379-4.
- ↑ Daniel RM, Finney JL, Réat V, Dunn R, Ferrand M, Smith JC. Enzyme dynamics and activity: time-scale dependence of dynamical transitions in glutamate dehydrogenase solution. „Biophys J”. Oct;77. 4, s. 2184-2190, 1999. PMID 10512837.
- ↑ Lilley D. Structure, folding and mechanisms of ribozymes. „Curr Opin Struct Biol”. 15, s. 313-23, 2005. PMID 15919196.
- ↑ Groves JT. Artificial enzymes. The importance of being selective. „Nature”. 389, s. 329-30, 1997. PMID 9311771.
- ↑ RAF de Réaumur. Observations sur la digestion des oiseaux. „Histoire de l'academie royale des sciences”. 1752, s. 266, 461, 1752.
- ↑ Williams, H. S. (1904) A History of Science: in Five Volumes. Volume IV: Modern Development of the Chemical and Biological Sciences Harper and Brothers (New York) (ang.)
- ↑ Dubos J.. Louis Pasteur: Free Lance of Science, Gollancz. Quoted in Manchester K. L. (1995) Louis Pasteur (1822–1895)—chance and the prepared mind.. „Trends Biotechnol”. 13, s. 511–515, 1951. PMID 8595136.
- ↑ ferment w internetowym "Słowniku wyrazów obcych i zwrotów obcojęzycznych Władysława Kopalińskiego"
- ↑ Nobel Laureate Biography of Eduard Buchner at http://nobelprize.org (ang.)
- ↑ Tekst wykładu Eduarda Buchnera z okazji nadania Nagrody Nobla w 1907 z http://nobelprize.org (ang.)
- ↑ 1946 Nagroda Nobla w dziedzinie chemii z http://nobelprize.org (ang.)
- ↑ Blake CC, Koenig DF, Mair GA, North AC, Phillips DC, Sarma VR.. Structure of hen egg-white lysozyme. A three-dimensional Fourier synthesis at 2 Angstrom resolution. „Nature”. 206, 1965. PMID 5891407.
- ↑ Chen LH, Kenyon GL, Curtin F, Harayama S, Bembenek ME, Hajipour G, Whitman CP. 4-Oxalocrotonate tautomerase, an enzyme composed of 62 amino acid residues per monomer. „J. Biol. Chem”. 267, s. 17716-21, 1992. PMID 1339435.
- ↑ Smith S. The animal fatty acid synthase: one gene, one polypeptide, seven enzymes. „FASEB J”. 15, s. 1248–59, 1994. PMID 8001737.
- ↑ Anfinsen C.B.. Principles that Govern the Folding of Protein Chains. „Science”, s. 223–230, 1973. PMID 4124164.
- ↑ The Catalytic Site Atlas at The European Bioinformatics Institute (ang.)
- ↑ Jaeger KE, Eggert T.. Enantioselective biocatalysis optimized by directed evolution.. „Curr Opin Biotechnol.”. 15(4), s. 305–313, 2004. PMID 15358000.
- ↑ Shevelev IV, Hubscher U.. The 3' 5' exonucleases.. „Nat Rev Mol Cell Biol.”. 3 (5), s. 364–376, 2002. PMID 11988770.
- ↑ Zenkin N, Yuzenkova Y, Severinov K.. Transcript-assisted transcriptional proofreading.. „Science.”. 313, s. 518–520, 2006. PMID 16873663.
- ↑ Ibba M, Soll D.. Aminoacyl-tRNA synthesis.. „Annu Rev Biochem.”. 69, s. 617–650, 2000. PMID 10966471.
- ↑ Rodnina MV, Wintermeyer W.. Fidelity of aminoacyl-tRNA selection on the ribosome: kinetic and structural mechanisms.. „Annu Rev Biochem.”. 70, s. 415–435, 2001. PMID 11395413.
- ↑ Richard Firn: The Screening Hypothesis - a new explanation of secondary product diversity and function. [dostęp 2006-10-11].
- ↑ Fischer E.. Einfluss der Configuration auf die Wirkung der Enzyme. „Ber. Dt. Chem. Ges.”. 27, s. 2985–2993, 1894.
- ↑ Koshland D. E.. Application of a Theory of Enzyme Specificity to Protein Synthesis. „Proc. Natl. Acad. Sci.”. 44, s. 98–104, 1958. PMID 16590179.
- ↑ Vasella A, Davies GJ, Bohm M.. Glycosidase mechanisms.. „Curr Opin Chem Biol.”. 6, s. 619–629, 2002. PMID 12413546.
- ↑ 6. W: Concepts in Biochemistry. New York, Chichester, Weinheim, Brisbane, Singapore, Toronto.: John Wiley & Sons, Inc., 2002, s. 137–138. ISBN 0-470-00379-0.
- ↑ Fersht, A (1985) Enzyme Structure and Mechanism (2nd ed) p50–52 W H Freeman & co, New York ISBN 0-7167-1615-1
- ↑ Jencks W.P. "Catalysis in Chemistry and Enzymology." 1987, Dover, New York
- ↑ Villa J, Strajbl M, Glennon TM, Sham YY, Chu ZT, Warshel A. How important are entropic contributions to enzyme catalysis?. „Proc. Natl. Acad. Sci. U.S.A.”. 97 (22), s. 11899-904, 2000. PMID: 11050223.
- ↑ Warshel A, Sharma PK, Kato M, Xiang Y, Liu H, Olsson MH. Electrostatic basis for enzyme catalysis. „Chem. Rev.”. 106 (8), s. 3210-35, 2006. PMID: 16895325.
- ↑ Eisenmesser EZ, Bosco DA, Akke M, Kern D. Enzyme dynamics during catalysis. Science. 2002 February 22;295(5559):1520–3. PMID: 11859194
- ↑ Agarwal PK. Role of protein dynamics in reaction rate enhancement by enzymes. J Am Chem Soc. 2005 November 2;127(43):15248-56. PMID: 16248667
- ↑ Eisenmesser EZ, Millet O, Labeikovsky W, Korzhnev DM, Wolf-Watz M, Bosco DA, Skalicky JJ, Kay LE, Kern D. Intrinsic dynamics of an enzyme underlies catalysis. Nature. 2005 November 3;438(7064):117-21. PMID: 16267559
- ↑ Yang LW, Bahar I.. Coupling between catalytic site and collective dynamics: A requirement for mechanochemical activity of enzymes.. „Structure.”. 13, s. 893–904, June 2005. PMID 15939021.
- ↑ Agarwal PK, Billeter SR, Rajagopalan PT, Benkovic SJ, Hammes-Schiffer S.. Network of coupled promoting motions in enzyme catalysis.. „Proc. Natl. Acad. Sci. U S A.”. 99, s. 2794–9, March 2002. PMID 11867722.
- ↑ Agarwal PK, Geist A, Gorin A. Protein dynamics and enzymatic catalysis: investigating the peptidyl-prolyl cis-trans isomerization activity of cyclophilin A. Biochemistry. 2004 August 24;43(33):10605-18. PMID: 15311922
- ↑ Tousignant A, Pelletier JN.. Protein motions promote catalysis.. „Chem Biol.”. 11 (8), s. 1037–42, Aug 2004. PMID 15324804.
- ↑ Olsson M.H.M., Parson W.W., Warshel A.. Dynamical Contributions to Enzyme Catalysis: Critical Tests of A Popular Hypothesis. „Chem. Rev”. 105, s. 1737-1756, 2006. PMID 16683752.
- ↑ M.W.G. de Bolster: Glossary of Terms Used in Bioinorganic Chemistry: Cofactors. 1997. [dostęp 2008-01-07]. (ang.).
- ↑ M.W.G. de Bolster: Glossary of Terms Used in Bioinorganic Chemistry: Coenzymes. 1997. [dostęp 2008-01-07]. (ang.).
- ↑ coenzymes and cofactors. [dostęp 2008-01-07]. (ang.).
- ↑ Enzyme Cofactors. [dostęp 2008-01-07]. (ang.).
- ↑ AF Wagner, KA Folkers: Vitamins and coenzymes. New York: Interscience Publishers, 1975. ISBN 0-88275-258-8.
- ↑ Henri,V.. Theorie generale de l'action de quelques diastases. „Compt. rend. hebd. Acad. Sci. Paris”. 135, s. 916-919, 1902.
- ↑ Sørensen,P.L.. Enzymstudien {II}. Über die Messung und Bedeutung der Wasserstoffionenkonzentration bei enzymatischen Prozessen. „Biochem. Z.”. 21, s. 131-304, 1909.
- ↑ Michaelis L., Menten M.. Die Kinetik der Invertinwirkung. „Biochem. Z.”. 49, s. 333–369, 1913.
- ↑ Briggs G. E., Haldane J. B. S.. A note on the kinetics of enzyme action. „Biochem. J.”. 19, s. 339–339, 1925. PMID 16743508.
- ↑ Without enzyme catalyst, slowest known biological reaction takes 1 trillion years. EurekAlert, 2003-05-3. [dostęp 2008-01-6]. (ang.).
- ↑ Radzicka A, Wolfenden R.. A proficient enzyme.. „Science”. 6 (267), s. 90–931, 1995. PMID 7809611.
- ↑ a b c pod redakcją A. Polanowskiego: Laboratorium z biochemii. Wrocław: Instytut Biochemii i Biologii Molekularnej Uniwersytetu Wrocławskiego, 2005. ISBN 83-921764-0-5.
- ↑ Garcia-Viloca M., Gao J., Karplus M., Truhlar D. G.. How enzymes work: analysis by modern rate theory and computer simulations.. „Science”. 303, s. 186–195, 2004. PMID 14716003.
- ↑ Olsson M. H., Siegbahn P. E., Warshel A.. Simulations of the large kinetic isotope effect and the temperature dependence of the hydrogen atom transfer in lipoxygenase. „J. Am. Chem. Soc.”. 126, s. 2820-1828, 2004. PMID 14995199.
- ↑ Masgrau L., Roujeinikova A., Johannissen L. O., Hothi P., Basran J., Ranaghan K. E., Mulholland A. J., Sutcliffe M. J., Scrutton N. S., Leys D.. Atomic Description of an Enzyme Reaction Dominated by Proton Tunneling. „Science”. 312, s. 237–241, 2006. PMID 16614214.
- ↑ Faergeman N. J, Knudsen J.. Role of long-chain fatty acyl-CoA esters in the regulation of metabolism and in cell signalling. „Biochem J”. 323, s. 1–12, April 1997. PMID 9173866.
- ↑ Doble B. W., Woodgett J. R.. GSK-3: tricks of the trade for a multi-tasking kinase. „J. Cell. Sci.”. 116, s. 1175–1186, April 2003. PMID 12615961.
- ↑ Del Papa MF, Hancock LE, Thomas VC, Perego M.. Full activation of Enterococcus faecalis gelatinase by a C-terminal proteolytic cleavage. „J Bacteriol.”. 189, s. 8835-43, Grudzień 2007. PMID 17921295.
- ↑ Stroud RM, Kossiakoff AA, Chambers JL.. Mechanisms of zymogen activation. „Annu Rev Biophys Bioeng.”. 6. s. 177-93. PMID 17350.
- ↑ Carr C. M., Kim P. S.. A spring-loaded mechanism for the conformational change of influenza hemagglutinin. „Cell”. 73, s. 823–832, April 2003. PMID 8500173.
- ↑ Cleland, W.W.. The Kinetics of Enzyme-catalyzed Reactions with two or more Substrates or Products 2. {I}nhibition: Nomenclature and Theory. „Biochim. Biophys. Acta”. 67, s. 173-187, 1963.
- ↑ Poulin R, Lu L, Ackermann B, Bey P, Pegg AE. Mechanism of the irreversible inactivation of mouse ornithine decarboxylase by alpha-difluoromethylornithine. Characterization of sequences at the inhibitor and coenzyme binding sites. J Biol Chem. 1992 Jan 5;267(1):150–8. PMID 1730582
- ↑ Berg JS, Powell BC, Cheney RE.. A millennial myosin census.. „Mol Biol Cell.”. 12(4), s. 780–794, 2001. PMID 11294886.
- ↑ a b enzymatic diseases. humpath.com. [dostęp 26-12-07]. (ang.).
- ↑ Mayer J, Thomas DW. Regulation of food intake and obesity. „Science”. Apr 21;156. 773, 1967. PMID 4886532.
- ↑ James WP, Trayhurn P. An integrated view of the metabolic and genetic basis for obesity. „Lancet”. Oct 9;2. 7989, 1976. PMID 61444.
- ↑ Gore J, Bryant Z, Stone MD, Nollmann M, Cozzarelli NR, Bustamante C. Mechanochemical Analysis of DNA Gyrase Using Rotor Bead Tracking. „Nature”. 439, s. 100-104, 2006.
- ↑ ExPASy: ENZYME (Enzyme nomenclature database).
- ↑ G.P. Moss: Recommendations of the Nomenclature Committee. [w:] International Union of Biochemistry and Molecular Biology on the Nomenclature and Classification of Enzymes by the Reactions they Catalyse [on-line].
- ↑ Renugopalakrishnan V, Garduno-Juarez R, Narasimhan G, Verma CS, Wei X, Li P. Rational design of thermally stable proteins: relevance to bionanotechnology. „J Nanosci Nanotechnol.”. 5, s. 1759–1767, 2005. PMID 16433409.
- ↑ Hult K, Berglund P.. Engineered enzymes for improved organic synthesis.. „Curr Opin Biotechnol.”. 14, s. 395–400, 2003. PMID 12943848.
- ↑ Headon DR, Walsh G.. The industrial production of enzymes. „Biotechnol Adv.”. 12, s. 635-646, 1994. PMID 14545919.
Linki zewnętrzne
[edytuj | edytuj kod]- Enzyme Structures Database - baza danych trójwymiarowych struktur enzymów w Protein Data Bank;
- ExPASy enzyme baza danych, linki do baz sekwencji Swiss-Prot, wejścia do innych baz danych;
- KEGG: Kyoto Encyclopedia of Genes and Genomes- graficzna i tekstowa dokumnetacja szlaków metabolicznych oraz enzymów;
- MACiE - baza danych mechanizmów reakcji enzymatycznych;
- MetaCyc - baza danych enzymów i szlaków metabolicznych;
- BRENDA - baza danych kompletnych informacji o enzymach; płatna dla użytkowników komercyjnych;
- Enzyme spotlight - miesięcznik przygotowywany przy udziale Europejskiego Instytutu Bioinformatyki na temat wybranych białek;